Протеасома


Протеасомы -это белковые комплексы , меченные убиквитин , которые разлагают белки , , протеолизом химическая реакция , которая разбивает пептидные связи . Ферменты , которые помогают таким реакциям, называются протеазами .
Протеасомы являются частью основного механизма, с помощью которого клетки регулируют концентрацию конкретных белков и разлагают неправильно свернутые белки . Белки помечены для деградации небольшим белком, называемым убиквитином . Реакция мечения катализируется ферментами, называемыми убиквитин -лигазами . Как только белок помечен одной молекулой убиквитина, это сигнал для других лигаз для прикрепления дополнительных молекул убиквитина. Результатом является цепь полиубиквитина , которая связана протеасомой, позволяющая ей разгромить мечевой белок. [ 1 ] Процесс деградации дает пептиды длиной около семи до восьми аминокислот , которые затем могут быть дополнительно разлагаются в более короткие аминокислотные последовательности и используются для синтеза новых белков. [ 1 ]
Протеасомы встречаются во всех эукариотах и археи , а также в некоторых бактериях . У эукариот протеасомы расположены как в ядре , так и в цитоплазме . [ 2 ]
In structure, the proteasome is a cylindrical complex containing a "core" of four stacked rings forming a central pore. Each ring is composed of seven individual proteins. The inner two rings are made of seven β subunits that contain three to seven protease active sites. These sites are located on the interior surface of the rings, so that the target protein must enter the central pore before it is degraded. The outer two rings each contain seven α subunits whose function is to maintain a "gate" through which proteins enter the barrel. These α subunits are controlled by binding to "cap" structures or regulatory particles that recognize polyubiquitin tags attached to protein substrates and initiate the degradation process. The overall system of ubiquitination and proteasomal degradation is known as the ubiquitin–proteasome system.[3]
The proteasomal degradation pathway is essential for many cellular processes, including the cell cycle, the regulation of gene expression, and responses to oxidative stress. The importance of proteolytic degradation inside cells and the role of ubiquitin in proteolytic pathways was acknowledged in the award of the 2004 Nobel Prize in Chemistry to Aaron Ciechanover, Avram Hershko and Irwin Rose.[4]
Discovery
[edit]Before the discovery of the ubiquitin–proteasome system, protein degradation in cells was thought to rely mainly on lysosomes, membrane-bound organelles with acidic and protease-filled interiors that can degrade and then recycle exogenous proteins and aged or damaged organelles.[1] However, work by Joseph Etlinger and Alfred L. Goldberg in 1977 on ATP-dependent protein degradation in reticulocytes, which lack lysosomes, suggested the presence of a second intracellular degradation mechanism.[5] This was shown in 1978 to be composed of several distinct protein chains, a novelty among proteases at the time.[6] Later work on modification of histones led to the identification of an unexpected covalent modification of the histone protein by a bond between a lysine side chain of the histone and the C-terminal glycine residue of ubiquitin, a protein that had no known function.[7] It was then discovered that a previously identified protein associated with proteolytic degradation, known as ATP-dependent proteolysis factor 1 (APF-1), was the same protein as ubiquitin.[8] The proteolytic activities of this system were isolated as a multi-protein complex originally called the multi-catalytic proteinase complex by Sherwin Wilk and Marion Orlowski.[9] Later, the ATP-dependent proteolytic complex that was responsible for ubiquitin-dependent protein degradation was discovered and was called the 26S proteasome.[10][11]
Much of the early work leading up to the discovery of the ubiquitin proteasome system occurred in the late 1970s and early 1980s at the Technion in the laboratory of Avram Hershko, where Aaron Ciechanover worked as a graduate student. Hershko's year-long sabbatical in the laboratory of Irwin Rose at the Fox Chase Cancer Center provided key conceptual insights, though Rose later downplayed his role in the discovery.[12] The three shared the 2004 Nobel Prize in Chemistry for their work in discovering this system.[4]
Although electron microscopy data revealing the stacked-ring structure of the proteasome became available in the mid-1980s,[13] the first structure of the proteasome core particle was not solved by X-ray crystallography until 1994.[14] In 2018, the first atomic structures of the human 26S proteasome holoenzyme in complex with a polyubiquitylated protein substrate were solved by cryogenic electron microscopy, revealing mechanisms by which the substrate is recognized, deubiquitylated, unfolded and degraded by the human 26S proteasome.[15]
Structure and organization
[edit]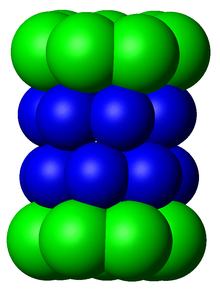
The proteasome subcomponents are often referred to by their Svedberg sedimentation coefficient (denoted S). The proteasome most exclusively used in mammals is the cytosolic 26S proteasome, which is about 2000 kilodaltons (kDa) in molecular mass containing one 20S protein subunit and two 19S regulatory cap subunits. The core is hollow and provides an enclosed cavity in which proteins are degraded; openings at the two ends of the core allow the target protein to enter. Each end of the core particle associates with a 19S regulatory subunit that contains multiple ATPase active sites and ubiquitin binding sites; it is this structure that recognizes polyubiquitinated proteins and transfers them to the catalytic core.[15] An alternative form of regulatory subunit called the 11S particle can associate with the core in essentially the same manner as the 19S particle; the 11S may play a role in degradation of foreign peptides such as those produced after infection by a virus.[16]
20S core particle
[edit]The number and diversity of subunits contained in the 20S core particle depends on the organism; the number of distinct and specialized subunits is larger in multicellular than unicellular organisms and larger in eukaryotes than in prokaryotes. All 20S particles consist of four stacked heptameric ring structures that are themselves composed of two different types of subunits; α subunits are structural in nature, whereas β subunits are predominantly catalytic. The α subunits are pseudoenzymes homologous to β subunits. They are assembled with their N-termini adjacent to that of the β subunits.[17] The outer two rings in the stack consist of seven α subunits each, which serve as docking domains for the regulatory particles and the alpha subunits N-termini (Pfam PF10584) form a gate that blocks unregulated access of substrates to the interior cavity.[18] The inner two rings each consist of seven β subunits and in their N-termini contain the protease active sites that perform the proteolysis reactions.[19] Three distinct catalytic activities were identified in the purified complex: chymotrypsin-like, trypsin-like and peptidylglutamyl-peptide hydrolyzing.[20] The size of the proteasome is relatively conserved and is about 150 angstroms (Å) by 115 Å. The interior chamber is at most 53 Å wide, though the entrance can be as narrow as 13 Å, suggesting that substrate proteins must be at least partially unfolded to enter.[21]
In archaea such as Thermoplasma acidophilum, all the α and all the β subunits are identical, whereas eukaryotic proteasomes such as those in yeast contain seven distinct types of each subunit. In mammals, the β1, β2, and β5 subunits are catalytic; although they share a common mechanism, they have three distinct substrate specificities considered chymotrypsin-like, trypsin-like, and peptidyl-glutamyl peptide-hydrolyzing (PHGH).[22] Alternative β forms denoted β1i, β2i, and β5i can be expressed in hematopoietic cells in response to exposure to pro-inflammatory signals such as cytokines, in particular, interferon gamma. The proteasome assembled with these alternative subunits is known as the immunoproteasome, whose substrate specificity is altered relative to the normal proteasome.[21] Recently an alternative proteasome was identified in human cells that lack the α3 core subunit.[23] These proteasomes (known as the α4-α4 proteasomes) instead form 20S core particles containing an additional α4 subunit in place of the missing α3 subunit. These alternative 'α4-α4' proteasomes have been known previously to exist in yeast.[24] Although the precise function of these proteasome isoforms is still largely unknown, cells expressing these proteasomes show enhanced resistance to toxicity induced by metallic ions such as cadmium.[23][25]
19S regulatory particle
[edit]The 19S particle in eukaryotes consists of 19 individual proteins and is divisible into two subassemblies, a 9-subunit base that binds directly to the α ring of the 20S core particle, and a 10-subunit lid. Six of the nine base proteins are ATPase subunits from the AAA Family, and an evolutionary homolog of these ATPases exists in archaea, called PAN (proteasome-activating nucleotidase).[26] The association of the 19S and 20S particles requires the binding of ATP to the 19S ATPase subunits, and ATP hydrolysis is required for the assembled complex to degrade folded and ubiquitinated proteins. Note that only the step of substrate unfolding requires energy from ATP hydrolysis, while ATP-binding alone can support all the other steps required for protein degradation (e.g., complex assembly, gate opening, translocation, and proteolysis).[27][28] In fact, ATP binding to the ATPases by itself supports the rapid degradation of unfolded proteins. However, while ATP hydrolysis is required for unfolding only, it is not yet clear whether this energy may be used in the coupling of some of these steps.[28][29]
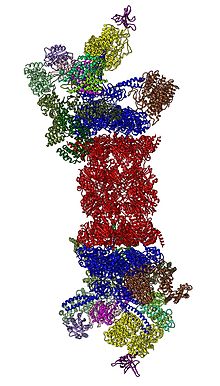
In 2012, two independent efforts have elucidated the molecular architecture of the 26S proteasome by single particle electron microscopy.[31][32] In 2016, three independent efforts have determined the first near-atomic resolution structure of the human 26S proteasome in the absence of substrates by cryo-EM.[33][34][35] In 2018, a major effort has elucidated the detailed mechanisms of deubiquitylation, initiation of translocation and processive unfolding of substrates by determining seven atomic structures of substrate-engaged 26S proteasome simultaneously.[15] In the heart of the 19S, directly adjacent to the 20S, are the AAA-ATPases (AAA proteins) that assemble to a heterohexameric ring of the order Rpt1/Rpt2/Rpt6/Rpt3/Rpt4/Rpt5. This ring is a trimer of dimers: Rpt1/Rpt2, Rpt6/Rpt3, and Rpt4/Rpt5 dimerize via their N-terminal coiled-coils. These coiled-coils protrude from the hexameric ring. The largest regulatory particle non-ATPases Rpn1 and Rpn2 bind to the tips of Rpt1/2 and Rpt6/3, respectively. The ubiquitin receptor Rpn13 binds to Rpn2 and completes the base sub-complex. The lid covers one half of the AAA-ATPase hexamer (Rpt6/Rpt3/Rpt4) and, unexpectedly, directly contacts the 20S via Rpn6 and to lesser extent Rpn5. The subunits Rpn9, Rpn5, Rpn6, Rpn7, Rpn3, and Rpn12, which are structurally related among themselves and to subunits of the COP9 complex and eIF3 (hence called PCI subunits) assemble to a horseshoe-like structure enclosing the Rpn8/Rpn11 heterodimer. Rpn11, the deubiquitinating enzyme, is placed at the mouth of the AAA-ATPase hexamer, ideally positioned to remove ubiquitin moieties immediately before translocation of substrates into the 20S. The second ubiquitin receptor identified to date, Rpn10, is positioned at the periphery of the lid, near subunits Rpn8 and Rpn9.
Conformational changes of 19S
[edit]The 19S regulatory particle within the 26S proteasome holoenzyme has been observed in six strongly differing conformational states in the absence of substrates to date.[36][37] A hallmark of the AAA-ATPase configuration in this predominant low-energy state is a staircase- or lockwasher-like arrangement of the AAA-domains.[30][31] In the presence of ATP but absence of substrate three alternative, less abundant conformations of the 19S are adopted primarily differing in the positioning of the lid with respect to the AAA-ATPase module.[33][37] In the presence of ATP-γS or a substrate, considerably more conformations have been observed displaying dramatic structural changes of the AAA-ATPase module.[15][36][38][39] Some of the substrate-bound conformations bear high similarity to the substrate-free ones, but they are not entirely identical, particularly in the AAA-ATPase module.[15][36] Prior to the 26S assembly, the 19S regulatory particle in a free form has also been observed in seven conformational states.[40] Notably, all these conformers are somewhat different and present distinct features. Thus, the 19S regulatory particle can sample at least 20 conformational states under different physiological conditions.
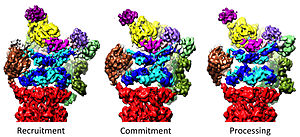
Regulation of the 20S by the 19S
[edit]The 19S regulatory particle is responsible for stimulating the 20S to degrade proteins. A primary function of the 19S regulatory ATPases is to open the gate in the 20S that blocks the entry of substrates into the degradation chamber.[41] The mechanism by which the proteasomal ATPase open this gate has been recently elucidated.[18] 20S gate opening, and thus substrate degradation, requires the C-termini of the proteasomal ATPases, which contains a specific motif (i.e., HbYX motif). The ATPases C-termini bind into pockets in the top of the 20S, and tether the ATPase complex to the 20S proteolytic complex, thus joining the substrate unfolding equipment with the 20S degradation machinery. Binding of these C-termini into these 20S pockets by themselves stimulates opening of the gate in the 20S in much the same way that a "key-in-a-lock" opens a door.[18] The precise mechanism by which this "key-in-a-lock" mechanism functions has been structurally elucidated in the context of human 26S proteasome at near-atomic resolution, suggesting that the insertion of five C-termini of ATPase subunits Rpt1/2/3/5/6 into the 20S surface pockets are required to fully open the 20S gate.[36][15][33]
Other regulatory particles
[edit]20S proteasomes can also associate with a second type of regulatory particle, the 11S regulatory particle, a heptameric structure that does not contain any ATPases and can promote the degradation of short peptides but not of complete proteins. It is presumed that this is because the complex cannot unfold larger substrates. This structure is also known as PA28, REG, or PA26.[17] The mechanisms by which it binds to the core particle through the C-terminal tails of its subunits and induces α-ring conformational changes to open the 20S gate suggest a similar mechanism for the 19S particle.[42] The expression of the 11S particle is induced by interferon gamma and is responsible, in conjunction with the immunoproteasome β subunits, for the generation of peptides that bind to the major histocompatibility complex.[16]
Yet another type of non-ATPase regulatory particle is the Blm10 (yeast) or PA200/PSME4 (human). It opens only one α subunit in the 20S gate and itself folds into a dome with a very small pore over it.[17]
Assembly
[edit]The assembly of the proteasome is a complex process due to the number of subunits that must associate to form an active complex. The β subunits are synthesized with N-terminal "propeptides" that are post-translationally modified during the assembly of the 20S particle to expose the proteolytic active site. The 20S particle is assembled from two half-proteasomes, each of which consists of a seven-membered pro-β ring attached to a seven-membered α ring. The association of the β rings of the two half-proteasomes triggers threonine-dependent autolysis of the propeptides to expose the active site. These β interactions are mediated mainly by salt bridges and hydrophobic interactions between conserved alpha helices whose disruption by mutation damages the proteasome's ability to assemble.[43] The assembly of the half-proteasomes, in turn, is initiated by the assembly of the α subunits into their heptameric ring, forming a template for the association of the corresponding pro-β ring. The assembly of α subunits has not been characterized.[44]
Only recently, the assembly process of the 19S regulatory particle has been elucidated to considerable extent. The 19S regulatory particle assembles as two distinct subcomponents, the base and the lid. Assembly of the base complex is facilitated by four assembly chaperones, Hsm3/S5b, Nas2/p27, Rpn14/PAAF1, and Nas6/gankyrin (names for yeast/mammals).[45] These assembly chaperones bind to the AAA-ATPase subunits and their main function seems to be to ensure proper assembly of the heterohexameric AAA-ATPase ring. To date it is still under debate whether the base complex assembles separately, whether the assembly is templated by the 20S core particle, or whether alternative assembly pathways exist. In addition to the four assembly chaperones, the deubiquitinating enzyme Ubp6/Usp14 also promotes base assembly, but it is not essential.[46] The lid assembles separately in a specific order and does not require assembly chaperones.[47]
Protein degradation process
[edit]
Ubiquitination and targeting
[edit]Proteins are targeted for degradation by the proteasome with covalent modification of a lysine residue that requires the coordinated reactions of three enzymes. In the first step, a ubiquitin-activating enzyme (known as E1) hydrolyzes ATP and adenylylates a ubiquitin molecule. This is then transferred to E1's active-site cysteine residue in concert with the adenylylation of a second ubiquitin.[48] This adenylylated ubiquitin is then transferred to a cysteine of a second enzyme, ubiquitin-conjugating enzyme (E2). In the last step, a member of a highly diverse class of enzymes known as ubiquitin ligases (E3) recognizes the specific protein to be ubiquitinated and catalyzes the transfer of ubiquitin from E2 to this target protein. A target protein must be labeled with at least four ubiquitin monomers (in the form of a polyubiquitin chain) before it is recognized by the proteasome lid.[49] It is therefore the E3 that confers substrate specificity to this system.[50] The number of E1, E2, and E3 proteins expressed depends on the organism and cell type, but there are many different E3 enzymes present in humans, indicating that there is a huge number of targets for the ubiquitin proteasome system.
The mechanism by which a polyubiquitinated protein is targeted to the proteasome is not fully understood. A few high-resolution snapshots of the proteasome bound to a polyubiquitinated protein suggest that ubiquitin receptors might be coordinated with deubiquitinase Rpn11 for initial substrate targeting and engagement.[15] Ubiquitin-receptor proteins have an N-terminal ubiquitin-like (UBL) domain and one or more ubiquitin-associated (UBA) domains. The UBL domains are recognized by the 19S proteasome caps and the UBA domains bind ubiquitin via three-helix bundles. These receptor proteins may escort polyubiquitinated proteins to the proteasome, though the specifics of this interaction and its regulation are unclear.[51]
The ubiquitin protein itself is 76 amino acids long and was named due to its ubiquitous nature, as it has a highly conserved sequence and is found in all known eukaryotic organisms.[52] The genes encoding ubiquitin in eukaryotes are arranged in tandem repeats, possibly due to the heavy transcription demands on these genes to produce enough ubiquitin for the cell. It has been proposed that ubiquitin is the slowest-evolving protein identified to date.[53] Ubiquitin contains seven lysine residues to which another ubiquitin can be ligated, resulting in different types of polyubiquitin chains.[54] Chains in which each additional ubiquitin is linked to lysine 48 of the previous ubiquitin have a role in proteasome targeting, while other types of chains may be involved in other processes.[55][56]

Deubiquitylation
[edit]Ubiquitin chains conjugated to a protein targeted for proteasomal degradation are normally removed by any one of the three proteasome-associated deubiquitylating enzymes (DUBs), which are Rpn11, Ubp6/USP14 and UCH37. This process recycles ubiquitin and is essential to maintain the ubiquitin reservoir in cells.[56] Rpn11 is an intrinsic, stoichiometric subunit of the 19S regulatory particle and is essential for the function of 26S proteasome. The DUB activity of Rpn11 is enhanced in the proteasome as compared to its monomeric form. How Rpn11 removes a ubiquitin chain en bloc from a protein substrate was captured by an atomic structure of the substrate-engaged human proteasome in a conformation named EB.[15] Interestingly, this structure also shows how the DUB activity is coupled to the substrate recognition by the proteasomal AAA-ATPase. In contrast to Rpn11, USP14 and UCH37 are the DUBs that do not always associated with the proteasome. In cells, about 10-40% of the proteasomes were found to have USP14 associated. Both Ubp6/USP14 and UCH37 are largely activated by the proteasome and exhibit a very low DUB activity alone. Once activated, USP14 was found to suppress proteasome function by its DUB activity and by inducing parallel pathways of proteasome conformational transitions, one of which turned out to directly prohibit substrate insertion into the AAA-ATPase, as intuitively observed by time-resolved cryogenic electron microscopy.[57] It appears that USP14 regulates proteasome function at multiple checkpoints by both catalytically competing with Rpn11 and allosterically reprogramming the AAA-ATPase states, which is rather unexpected for a DUB.[57] These observations imply that the proteasome regulation may depend on its dynamic transitions of conformational states.
Unfolding and translocation
[edit]After a protein has been ubiquitinated, it is recognized by the 19S regulatory particle in an ATP-dependent binding step.[15][28] The substrate protein must then enter the interior of the 20S subunit to come in contact with the proteolytic active sites. Because the 20S particle's central channel is narrow and gated by the N-terminal tails of the α ring subunits, the substrates must be at least partially unfolded before they enter the core.[15] The passage of the unfolded substrate into the core is called translocation and necessarily occurs after deubiquitination.[15][28] However, the order in which substrates are deubiquitinated and unfolded is not yet clear.[58] Which of these processes is the rate-limiting step in the overall proteolysis reaction depends on the specific substrate; for some proteins, the unfolding process is rate-limiting, while deubiquitination is the slowest step for other proteins.[27] The extent to which substrates must be unfolded before translocation is suggested to be around 20 amino acid residues by the atomic structure of the substrate-engaged 26S proteasome in the deubiquitylation-compatible state,[15] but substantial tertiary structure, and in particular nonlocal interactions such as disulfide bonds, are sufficient to inhibit degradation.[59] The presence of intrinsically disordered protein segments of sufficient size, either at the protein terminus or internally, has also been proposed to facilitate efficient initiation of degradation.[60][61]
The gate formed by the α subunits prevents peptides longer than about four residues from entering the interior of the 20S particle. The ATP molecules bound before the initial recognition step are hydrolyzed before translocation. While energy is needed for substrate unfolding, it is not required for translocation.[27][28] The assembled 26S proteasome can degrade unfolded proteins in the presence of a non-hydrolyzable ATP analog, but cannot degrade folded proteins, indicating that energy from ATP hydrolysis is used for substrate unfolding.[27] Passage of the unfolded substrate through the opened gate occurs via facilitated diffusion if the 19S cap is in the ATP-bound state.[62]
The mechanism for unfolding of globular proteins is necessarily general, but somewhat dependent on the amino acid sequence. Long sequences of alternating glycine and alanine have been shown to inhibit substrate unfolding, decreasing the efficiency of proteasomal degradation; this results in the release of partially degraded byproducts, possibly due to the decoupling of the ATP hydrolysis and unfolding steps.[63] Such glycine-alanine repeats are also found in nature, for example in silk fibroin; in particular, certain Epstein–Barr virus gene products bearing this sequence can stall the proteasome, helping the virus propagate by preventing antigen presentation on the major histocompatibility complex.[64]

Proteolysis
[edit]The proteasome functions as an endoprotease.[65][66][67][68] The mechanism of proteolysis by the β subunits of the 20S core particle is through a threonine-dependent nucleophilic attack. This mechanism may depend on an associated water molecule for deprotonation of the reactive threonine hydroxyl. Degradation occurs within the central chamber formed by the association of the two β rings and normally does not release partially degraded products, instead reducing the substrate to short polypeptides typically 7–9 residues long, though they can range from 4 to 25 residues, depending on the organism and substrate. The biochemical mechanism that determines product length is not fully characterized.[69] Although the three catalytic β subunits have a common mechanism, they have slightly different substrate specificities, which are considered chymotrypsin-like, trypsin-like, and peptidyl-glutamyl peptide-hydrolyzing (PHGH)-like. These variations in specificity are the result of interatomic contacts with local residues near the active sites of each subunit. Each catalytic β subunit also possesses a conserved lysine residue required for proteolysis.[22]
Хотя протеасома обычно производит очень короткие пептидные фрагменты, в некоторых случаях эти продукты сами являются биологически активными и функциональными молекулами. Некоторые факторы транскрипции, регулирующие экспрессию специфических генов, в том числе один компонент комплекса NF-κB млекопитающих , синтезируются как неактивные предшественники, чья убиквитинирование и последующая протеасомная деградация превращают их в активную форму. Такая активность требует, чтобы протеасома была внутренне расщеплять белок субстрата, а не обрабатывать его от одного конца. Было высказано предположение, что длинные петли на поверхностях этих белков служат протеасомными субстратами и попадают в центральную полость, в то время как большая часть белка остается снаружи. [ 70 ] Аналогичные эффекты наблюдались у дрожжевых белков; Этот механизм селективной деградации известен как регулируемый убиквитин/протеасома, зависящая от обработки (RUP). [ 71 ]
Убиквитин-независимая деградация
[ редактировать ]Хотя большинство протеасомных субстратов должны быть убиквитинированы перед деградацией, существуют некоторые исключения из этого общего правила, особенно когда протеасома играет нормальную роль в пост- трансляционной обработке белка. Протеасомная активация NF-κB путем обработки p105 в p50 через внутренний протеолиз является одним из основных примеров. [ 70 ] Некоторые белки, которые предполагают, являются нестабильными из -за внутренних неструктурированных областей, [ 72 ] деградируют, независимо от убиквитина. Наиболее известным примером убиквитин-независимой протеасомной субстрата является фермент орнитин декарбоксилаза . [ 73 ] что убиквитин-независимые механизмы, нацеленные на ключевые регуляторы клеточного цикла, такие как p53 , хотя p53 также подвержен убиквитин-зависимой деградации. Также сообщалось, [ 74 ] Наконец, структурно аномальные, неправильно свернутые или высоко окисленные белки также подвергаются убиквитин-независимой и 19S-независимой деградации в условиях клеточного стресса. [ 75 ]
Эволюция
[ редактировать ]
Протеасома 20 -х годов является повсеместной и важной у эукариот и археи. бактериального , чьи генные продукты представляют собой мультимерную протеазу , порядка Актиномицеталы также имеют гомологи протеасомы 20S, тогда как большинство бактерий обладают теплового шока генами HSLV и HSLU расположенную в двухслойном кольце и атфазе. [ 76 ] Был предположил, что белок HSLV напоминает вероятный предок протеасомы 20 -х годов. [ 77 ] В целом, HSLV не является необходимым у бактерий, и не все бактерии обладают им, тогда как некоторые протисты обладают как системами 20 -х, так и HSLV. [ 76 ] Многие бактерии также обладают другими гомологами протеасомы и ассоциированной АТФазы, особенно CLPP и CLPX . Эта избыточность объясняет, почему система HSLUV не является обязательной.
Анализ последовательности показывает, что каталитические β -субъединицы расходились ранее в эволюции, чем преимущественно структурные α -субъединицы. У бактерий, которые экспрессируют протеасому 20S, β -субъединицы имеют высокую идентичность последовательности для архейных и эукариотических β -субъединиц, тогда как идентичность α -последовательности намного ниже. Присутствие 20S протеасомов у бактерий может быть результатом бокового переноса генов , в то время как диверсификация субъединиц среди эукариот приписывается множественным событиям дупликации генов . [ 76 ]
Контроль клеточного цикла
[ редактировать ]Прогрессирование клеточного цикла контролируется упорядоченным действием циклина-зависимых киназ (CDK), активируемых специфическими циклинами , которые разграничивают фазы клеточного цикла . Митотические циклины, которые сохраняются в клетке в течение всего нескольких минут, имеют один из самых коротких пролетов с жильем всех внутриклеточных белков. [ 1 ] После того, как комплекс CDK-циклина выполнил свою функцию, связанный циклин полиубиквитинирован и разрушается протеасомой, которая обеспечивает направленность для клеточного цикла. В частности, выход из митоза требует протеасомной диссоциации регуляторного компонента циклина B из комплекса фактора, способствующего митозу . [ 78 ] В клетках позвоночных «проскальзывание» через митотическую контрольную точку, приводящую к преждевременному выходу из M -фазы , может произойти, несмотря на задержку этого выхода на контрольно -пропускной пункте веретена . [ 79 ]
Более ранние контрольно-пропускные пункты клеточного цикла, такие как проверка точек после ограничения между G 1 фазой и S аналогично, включают протеасомную деградацию циклина A , убиквитинирование которой способствует Anaphase Propering Complex E3 (APC), убиквитиновой лигазу . [ 80 ] APC и белковой комплекс SKP1/CUL1/F-BOX ( комплекс SCF ) являются двумя ключевыми регуляторами деградации циклина и контроля контрольной точки; Сам SCF регулируется APC посредством убиквитинирования белка адаптера SKP2, который предотвращает активность SCF до перехода G1-S. [ 81 ]
Отдельные компоненты частицы 19S имеют свои собственные регулирующие роли. Ганкирин , недавно идентифицированный онкопротеин , является одним из 19S подкомпонентов, которые также жестко связывают циклин-зависимую киназу CDK4 и играет ключевую роль в распознавании убиквитинированной P53 с помощью его сродства к убиквитиновой лигазе MDM2 . Ганкирин является антипоптотическим и , как было показано, сверхэкспрессируется у некоторых типов опухолевых клеток, таких как гепатоцеллюлярная карцинома . [ 82 ]
Как и эукариоты, некоторые археи также используют протеасому для контроля клеточного цикла, в частности, путем контроля деления клеток -опосредованного ESCRT -II. [ 83 ]
Регулирование роста растений
[ редактировать ]У растений , передачи сигналов ауксинов , или фитомормонов , которые упорядочивают направление и тропизм роста растений, вызывает нацеливание класса репрессоров транскрипционного фактора, известных как белки AUX/IAA для протеасомной деградации. Эти белки убиквитинируются SCFTIR1 или SCF в комплексе с рецептором ауксина TIR1. Деградация белков AUX/IAA Дерепрессирует транскрипционные факторы в семействе фактора ауксин-ответ (ARF) и индуцирует экспрессию генов, направленную ARF. [ 84 ] Клеточные последствия активации ARF зависят от типа растения и стадии развития, но участвуют в направлении роста корней и вен листьев. Считается, что специфический ответ на арф -дерепрессию опосредован специфичностью в спаривании отдельных белков ARF и AUX/IAA. [ 85 ]
Апоптоз
[ редактировать ]Как внутренние, так и внешние сигналы могут привести к индукции апоптоза или запрограммированной гибели клеток. Полученная деконструкция клеточных компонентов в первую очередь выполняется специализированными протеазами, известными как каспазы , но протеасома также играет важную и разнообразную роль в апоптотическом процессе. Участие протеасомы в этом процессе указывает как увеличение убиквитинирования белка, так и ферментов E1, E2 и E3, которое наблюдается намного до апоптоза. [ 86 ] [ 87 ] [ 88 ] Во время апоптоза также наблюдалось протеасомы в ядре, также наблюдалось, как транспортируется в внешние мембранные пузырьки , характерные для апоптоза. [ 89 ]
Ингибирование протеасомы оказывает различное влияние на индукцию апоптоза в разных типах клеток. В целом, протеасома не требуется для апоптоза, хотя ингибирование ее проапоптотической в большинстве типов клеток, которые были изучены. Апоптоз опосредуется путем нарушения регулируемой деградации белков клеточного цикла Pro-роста. [ 90 ] Однако некоторые клеточные линии - в частности, первичные культуры покоящихся и дифференцированных клеток , таких как тимоциты и нейроны - не позволяют подвергнуться апоптозу при воздействии ингибиторов протеасом. Механизм этого эффекта не ясен, но предположительно, как специфичный для клеток в покоящихся состояниях или в результате дифференциальной активности проапоптотической киназы JNK . [ 91 ] Способность ингибиторов протеасомы вызывать апоптоз в быстро делящихся клетках была использована в нескольких недавно разработанных химиотерапевтических агентах, таких как бортезомиб и салиноспорамид А.
Ответ на клеточный стресс
[ редактировать ]В ответ на клеточные стрессы, такие как инфекция , тепловой шок или окислительное повреждение , - белки теплового шока экспрессируются , которые идентифицируют неправильно свернутые или развернутые белки и нацелены на их для протеасомной разложения. Как HSP27 , так и HSP90 - белки шаперона были вовлечены в повышение активности системы убиквитин-протеасом, хотя они не являются прямыми участниками процесса. [ 92 ] HSP70 , с другой стороны, связывает обнаженные гидрофобные пятна на поверхности неправильно свернутых белков и рекрутирует E3 -убиквитин -лигазы, такие как ChIP, для помещения белков для протеасомной разложения. [ 93 ] Белок чипа (карбоксильный термин HSP70-взаимодействующего белка) сам по себе регулируется посредством ингибирования взаимодействия между чипом фермента E3 и его партнером по связыванию E2. [ 94 ]
Существуют аналогичные механизмы, чтобы способствовать деградации окислительно поврежденных белков через систему протеасом. В частности, протеасомы, локализованные в ядре, регулируются PARP и активно разлагают ненадлежащие окисленные гистоны . [ 95 ] Окисленные белки, которые часто образуют большие аморфные агрегаты в клетке, могут быть разлагаются непосредственно на частицу ядра 20S без регуляторной крышки 19S и не требуют гидролиза или мечения АТФ с помощью убиквитина. [ 75 ] Однако высокий уровень окислительного повреждения увеличивает степень сшивания между фрагментами белка, что делает агрегаты устойчивыми к протеолизу. Большие числа и размеры таких высоко окисленных агрегатов связаны со старением . [ 96 ]
Дисгуляция системы протеасом убиквитина может способствовать нескольким нейронным заболеваниям. Это может привести к опухолям мозга, таким как астроцитом . [ 97 ] При некоторых из последующих нейродегенеративных заболеваний, которые разделяют агрегацию неправильно свернутых белков как общую особенность, такие как болезнь Паркинсона и болезнь Альцгеймера , крупные нерастворимые агрегаты неправильно свернутых белков могут образовываться, а затем приводит нейротоксично к Полем Снижение активности протеасом было предложено в качестве причины агрегации и образования тела Льюи в Паркинсоне. [ 98 ] Эта гипотеза подтверждается наблюдением, что дрожжи модели Паркинсона более подвержены токсичности от α-синуклеина , основного белкового компонента тел Lewy, в условиях низкой активности протеасом. [ 99 ] Нарушение протеасомной активности может лежать в основе когнитивных расстройств, таких как расстройства аутистического спектра , и мышечные и нервные заболевания, такие как миопатия тела включения . [ 97 ]
Роль в иммунной системе
[ редактировать ]Протеасома играет простую, но критическую роль в функции адаптивной иммунной системы . Пептидные антигены отображаются с помощью основных белков комплекса гистосовместимости класса I (MHC) на поверхности антиген-презентативных клеток . Эти пептиды представляют собой продукты протеасомной деградации белков, возникающих в результате вторгаемого патогена . Хотя конститутивно экспрессируемые протеасомы могут участвовать в этом процессе, специализированный комплекс, состоящий из белков, экспрессия которых индуцируется гаммой интерферонов , являются основными производителями пептидов, которые являются оптимальными по размеру и составу для связывания MHC. Эти белки, экспрессия которых увеличивается во время иммунного ответа, включают в себя регуляторную частицу 11S, основная известная биологическая роль которых регулирует производство лигандов MHC, и специализированные β -субъединицы, называемые β1i, β2i и β5i с измененной специфичностью субстрата. Комплекс, образованный со специализированными β -субъединицами, известен как иммунопротеасом . [ 16 ] Другая субъединица β5i, β5T, экспрессируется в тимусе, что приводит к специфичной тимусу « тимопротеасоме », чья функция пока неясна. [ 100 ]
Сила связывания лиганда MHC I-лиганда зависит от состава C-конца лиганда , поскольку пептиды связываются с помощью водородных связей и близкими контактами с областью, называемой «B-карманом» на поверхности MHC. Многие аллели MHC класса I предпочитают гидрофобные С-концевые остатки, а комплекс иммунопротеаса с большей вероятностью генерирует гидрофобные С-конце. [ 101 ]
Благодаря своей роли в создании активированной формы NF-κB , антипоптотический и воспалительный регулятор экспрессии цитокинов , протеасомная активность была связана с воспалительными и аутоиммунными заболеваниями . Повышенные уровни активности протеасом коррелируют с активностью заболевания и участвуют в аутоиммунных заболеваниях, включая системную волчанку и ревматоидный артрит . [ 16 ]
Протеасома также участвует во внутриклеточных антитело, опосредованных протеолизом вирионов, связанных с антителами. В этом пути нейтрализации TRIM21 (белок семейства трехсторонних мотивов) связывается с иммуноглобулином G , чтобы направить вирион в протеасому, где она разлагается. [ 102 ]
Ингибиторы протеасом
[ редактировать ]

Ингибиторы протеасомы обладают эффективной антипухогнутой активностью в клеточной культуре , индуцируя апоптоз путем нарушения регулируемой деградации белков клеточного цикла Pro-роста. [ 90 ] Этот подход селективного индуцирования апоптоза в опухолевых клетках оказался эффективным в моделях на животных и исследованиях человека.
Lactacystin , натуральный продукт, синтезированный Streptomyces бактериями , был первым непептидным ингибитором протеасом [ 103 ] и широко используется в качестве инструмента исследования в области биохимии и клеточной биологии. Lactacystin был лицензирован на миогенцию/профик, который был приобретен Millennium Pharmaceuticals , в настоящее время частью Takeda Pharmaceuticals . Лактацистин ковалентно модифицирует амино-концевой треонин каталитических β-субъединиц протеасомы, особенно субъединицы β5, ответственной за химотрипсин-активность протеасомы. Это открытие помогло установить протеасому как механически новый класс протеазы: аминоконцевая треониновая протеаза .
Bortezomib (Boronated MG132), молекула, разработанная фармацевтическими препаратами Millennium и продаваемой как Velcade, является первым ингибитором протеасом, который достиг клинического использования в качестве химиотерапевтического агента. [ 104 ] Бортезомиб используется при лечении множественной миеломы . [ 105 ] Примечательно, что множественная миелома приводит к повышению уровней пептидов, полученных из протеасомы, в сыворотке крови , которые снижаются до нормальных уровней в ответ на успешную химиотерапию. [ 106 ] Исследования на животных показали, что бортезомиб также может оказывать клинически значимое влияние на рак поджелудочной железы . [ 107 ] [ 108 ] Доклинические и ранние клинические исследования начали изучать эффективность Bortezomib в лечении других раковых заболеваний, связанных с B -клетками , [ 109 ] В частности, некоторые виды неходжкинской лимфомы . [ 110 ] Клинические результаты также, по-видимому, оправдывают использование ингибитора протеасомы в сочетании с химиотерапией, для острого лимфобластного лейкоза B-клеток [ 111 ] Ингибиторы протеасом могут убивать некоторые виды культивируемых клеток лейкоза, которые устойчивы к глюкокортикоидам. [ 112 ]
молекулы Ритонавир , продаваемый как Норвир, был разработан в качестве ингибитора протеазы и использовался для нацеливания на ВИЧ -инфекцию. Тем не менее, было показано, что он ингибирует протеасомы, а также свободные протеазы; Чтобы быть специфическим, ритонавир, похожая на химотрипсин, ингибируется ритонавиром, в то время как активность, подобная трипсину , несколько усилена. [ 113 ] Исследования на животных моделях предполагают, что ритонавир может оказывать ингибирующее влияние на рост клеток глиомы . [ 114 ]
Ингибиторы протеасомы также показали перспективу при лечении аутоиммунных заболеваний на животных моделях. Например, исследования на мышах с кожными трансплантатами человека обнаружили уменьшение размера поражений от псориаза после лечения ингибитором протеасомы. [ 115 ] Ингибиторы также демонстрируют положительные эффекты на грызунов моделях астмы . [ 116 ]
Маркировка и ингибирование протеасомы также представляют интерес к лабораторным условиям как для in vitro , так и in vivo исследования протеасомной активности в клетках. Наиболее часто используемыми лабораторными ингибиторами являются лактацистин и пептид альдегид MG132, первоначально разработанные Goldberg Lab. Флуоресцентные ингибиторы также были разработаны, чтобы специально пометить активные участки собранной протеасомы. [ 117 ]
Клиническое значение
[ редактировать ]Протеасома и ее субъединицы имеют клиническое значение, по крайней мере, по двум причинам: (1) нарушенная сложная сборка или дисфункциональная протеасома может быть связана с основной патофизиологией специфических заболеваний, и (2) они могут использоваться в качестве мишеней лекарств для терапевтических вмешательства. Совсем недавно было предпринято больше усилий, чтобы рассмотреть протеасому для разработки новых диагностических маркеров и стратегий. Улучшение и всестороннее понимание патофизиологии протеасомы должно привести к клиническим применениям в будущем.
Протеасомы образуют ключевой компонент для системы убиквитин -протеасом (UPS) [ 118 ] и соответствующий контроль качества клеточного белка (PQC). белка Убиквитинирование и последующий протеолиз и деградация протеасомой являются важными механизмами в регуляции клеточного цикла , роста и дифференцировки клеток , транскрипции генов, трансдукции сигнала и апоптоза . [ 119 ] Дефекты протеасом приводят к снижению протеолитической активности и накоплению поврежденных или неправильно свернутых белков, которые могут способствовать нейродегенеративному заболеванию, [ 120 ] [ 121 ] сердечно -сосудистые заболевания, [ 122 ] [ 123 ] [ 124 ] воспалительные реакции и аутоиммунные заболевания, [ 125 ] и системные реакции повреждения ДНК, приводящие к злокачественным новообразованиям . [ 126 ]
Исследования привели к дефектам UPS в патогенезе нейродегенеративных и миодегенеративных расстройств, включая болезнь Альцгеймера , [ 127 ] Болезнь Паркинсона [ 128 ] И болезнь Пика , [ 129 ] амиотрофический боковой склероз (БАС), [ 129 ] Болезнь Хантингтона , [ 128 ] Creutzfeldt -Jakob болезнь , [ 130 ] и заболевания моторных нейронов, заболевания полиглутамина (полик), мышечная дистрофия [ 131 ] и несколько редких форм нейродегенеративных заболеваний, связанных с деменцией . [ 132 ] В рамках системы убиквитин -протеасом (UPS), протеасома поддерживает гомеостаз сердечного белка и, следовательно, играет значительную роль в ишемическом повреждении сердца, [ 133 ] желудочковая гипертрофия [ 134 ] и сердечная недостаточность . [ 135 ] Кроме того, накапливаются доказательства того, что UPS играет важную роль в злокачественной трансформации. Протеолиз UPS играет важную роль в реакциях раковых клеток на стимулирующие сигналы, которые имеют решающее значение для развития рака. Соответственно, экспрессия генов путем деградации факторов транскрипции , таких как p53 , c-Jun , c-fos , nf-κB , c-myc , hif-1α, matα2, stat3 , белки, регулируемые стеролом, и андрогенные рецепторы все являются контролируется UPS и, таким образом, участвует в развитии различных злокачественных новообразований. [ 136 ] Более того, UPS регулирует деградацию продуктов гена -супрессора опухоли, таких как аденоматозная полипоза Coli (APC) при колоректальном раке, ретинобластома (RB). и супрессор опухоли фон Хиппель-Линдау (VHL), а также ряд протоонкогенов ( RAF , MYC , MYB , REL , SRC , MOS , ABL ). UPS также участвует в регуляции воспалительных реакций. Эта активность обычно объясняется роли протеасомов в активации NF-κB, которая дополнительно регулирует экспрессию PRO-воспалительных цитокинов, таких как TNF-α , IL-β, IL-8 , молекулы адгезии ( ICAM-1 , VCAM-1 , P-Selectin ) и простагландины и оксид азота (нет). [ 125 ] Кроме того, UPS также играет роль в воспалительных реакциях в качестве регуляторов пролиферации лейкоцитов, главным образом посредством протеолиза циклов и деградации ингибиторов CDK . [ 137 ] Наконец, пациенты с аутоиммунным заболеванием с СКВ , синдромом Шегрена и ревматоидным артритом (РА) преимущественно демонстрируют циркулирующие протеасомы, которые могут применяться в качестве клинических биомаркеров. [ 138 ]
Смотрите также
[ редактировать ]- Карта протеолиза
- Семейство белков DSS1/SEM1
- Экзосомный комплекс
- Эндоплазмак-ретикаллум, связанная с деградацией белка
- Junq и iPod
Ссылки
[ редактировать ]- ^ Jump up to: а беременный в дюймовый Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J (2004). "3" . Молекулярная клеточная биология (5 -е изд.). Нью -Йорк: WH Freeman and Co. С. 66–72 . ISBN 978-0-7167-4366-8 .
- ^ Peters JM, Franke WW, Kleinschmidt JA (март 1994 г.). «Отчетливые 19 с и 20 с подкомплексов протеасомы 26 с и их распределение в ядре и цитоплазме» . Журнал биологической химии . 269 (10): 7709–18. doi : 10.1016/s0021-9258 (17) 37345-3 . PMID 8125997 .
- ^ Нассиф, Николас Д.; Кембрей, Саманта Э.; Краут, Даниэль А. (май 2014 г.). «Снижение: частичная деградация субстрата с помощью АТФ-зависимых протеаз» . Жизнь iubmb . 66 (5): 309–317. doi : 10.1002/iub.1271 . PMID 24823973 . S2CID 29860298 .
- ^ Jump up to: а беременный Комитет Нобелевской премии (2004). "Нобелевская премия в химии, 2004 год" . Получено 11 декабря 2006 года .
- ^ Etlinger JD, Goldberg AL (январь 1977 г.). «Растворимая АТФ-зависимая протеолитическая система, ответственная за деградацию аномальных белков в ретикулоцитах» . Труды Национальной академии наук Соединенных Штатов Америки . 74 (1): 54–8. Bibcode : 1977pnas ... 74 ... 54e . doi : 10.1073/pnas.74.1.54 . PMC 393195 . PMID 264694 .
- ^ Ciehanover A, Hod Y, Hershko A (апрель 1978 г.). «Теплостабильный полипептидный компонент АТФ-зависимой протеолитической системы из ретикулоцитов». Биохимическая и биофизическая исследовательская коммуникация . 81 (4): 1100–5. doi : 10.1016/0006-291x (78) 91249-4 . PMID 666810 .
- ^ Goldknopf IL, Busch H (март 1977 г.). «Изопептидная связь между полипептидами негистона и гистона 2а хромосомного конъюгата A24» . Труды Национальной академии наук Соединенных Штатов Америки . 74 (3): 864–8. Bibcode : 1977pnas ... 74..864G . doi : 10.1073/pnas.74.3.864 . PMC 430507 . PMID 265581 .
- ^ Ciechanover A (сентябрь 2005 г.). «Ранняя работа над системой протеасомы убиквитина, интервью с Aaron Ciechanover. Интервью CDD» . Гибель клеток и дифференциация . 12 (9): 1167–77. doi : 10.1038/sj.cdd.4401691 . PMID 16094393 .
- ^ Wilk S, Orlowski M (ноябрь 1980 г.). «Чувствительная к катионе нейтральная эндопептидаза: выделение и специфичность фермента для бычьего гипофиза». Журнал нейрохимии . 35 (5): 1172–82. doi : 10.1111/j.1471-4159.1980.tb07873.x . PMID 6778972 . S2CID 9028201 .
- ^ Arrigo AP, Tanaka, K, Goldberg F, Welch WJ (1988). «Идентификация 19S Prosome Partice с большим многофункциональным протеазой комплексом клеток млекопитающих». Природа . 331 (6152): 192–94. doi : 10.1038/331192A0 . PMID 3277060 . S2CID 97688 . Танака К., Ваксман Л., Голдберг А.Л. (июнь 1983 г.). «ATP выполняет две разные роли в деградации белка в ретикулоцитах, один требующий и одну независимо от убиквитина» . Журнал клеточной биологии . 96 (6): 1580–5. doi : 10.1083/jcb.96.6.1580 . PMC 2112434 . PMID 6304111 .
- ^ Hough R, Pratt G, Rechsteiner M (июнь 1987 г.). «Очистка двух высокомолекулярных протеаз из лизата ретикулоцитов кролика» . Журнал биологической химии . 262 (17): 8303–13. doi : 10.1016/s0021-9258 (18) 47564-3 . PMID 3298229 .
- ^ Хершко А (сентябрь 2005 г.). «Ранняя работа над системой протеасомы убиквитина, интервью с Avram Hershko. Интервью CDD» . Гибель клеток и дифференциация . 12 (9): 1158–61. doi : 10.1038/sj.cdd.4401709 . PMID 16094391 .
- ^ Kopp F, Steiner R, Dahlmann B, Kuehn L, Rearauer H (август 1986 г.). «Размер и форма мультикаталитической протеиназы из скелетных мышц крысы». Biochimica et Biophysica Acta (BBA) - структура белка и молекулярная ферма . 872 (3): 253–60. doi : 10.1016/0167-4838 (86) 90278-5 . PMID 3524688 .
- ^ Löwe J, Stock D, Jap B, Zwickl P, Baumeister W, Huber R (апрель 1995 г.). «Кристаллическая структура протеасомы 20S из археона T. acidophilum при 3.4 a разрешения». Наука . 268 (5210): 533–9. Bibcode : 1995sci ... 268..533L . doi : 10.1126/science.7725097 . PMID 7725097 .
- ^ Jump up to: а беременный в дюймовый и фон глин час я Дж k л Dong Y, Zhang S, Wu Z, Li X, Wang WL, Zhu Y, Stoilova-McPhie S, Lu Y, Finley D, Mao Y (ноябрь 2018). «Крио-эм структуры и динамика протеасомы человека 26-х годов» . Природа . 565 (7737): 49–55. doi : 10.1038/s41586-018-0736-4 . PMC 6370054 . PMID 30479383 .
- ^ Jump up to: а беременный в дюймовый Ван Дж., Мальдонадо, штат Массачусетс (август 2006 г.). «Система убиквитин-протеасом и ее роль в воспалительных и аутоиммунных заболеваниях». Клеточная и молекулярная иммунология . 3 (4): 255–61. PMID 16978533 .
- ^ Jump up to: а беременный в Stadtmueller, BM; Хилл, CP (7 января 2011 г.). "Протеасомные активаторы" . Молекулярная клетка . 41 (1): 8–19. doi : 10.1016/j.molcel.2010.12.020 . PMC 3040445 . PMID 21211719 .
- ^ Jump up to: а беременный в Смит Д.М., Чанг С.К., Парк С., Финли Д., Ченг Й, Голдберг А.Л. (сентябрь 2007 г.). «Док -санитарная стыковка карбоксильных терминах протеасомных АТФаз в альфа -кольце протеасомы 20S открывает ворота для входа подложки» . Молекулярная клетка . 27 (5): 731–44. doi : 10.1016/j.molcel.2007.06.033 . PMC 2083707 . PMID 17803938 .
- ^ "Merops Family T1" . Embl-Ebi . Получено 16 февраля 2019 года .
- ^ Wilk S, Orlowski M (март 1983 г.). «Доказательства того, что чувствительная к катионной катионной гипофизы нейтральная эндопептидаза представляет собой мультикаталитический протеазовый комплекс». Журнал нейрохимии . 40 (3): 842–9. doi : 10.1111/j.1471-4159.1983.tb08056.x . PMID 6338156 . S2CID 23508675 .
- ^ Jump up to: а беременный Нанди Д., Тахилиани П., Кумар А., Чанду Д (март 2006 г.). «Система убиквитина-протеемного» (PDF) . Журнал биологических наук . 31 (1): 137–55. doi : 10.1007/bf02705243 . PMID 16595883 . S2CID 21603835 .
- ^ Jump up to: а беременный Heinemeyer W, Fischer M, Krimmer T, Stachon U, Wolf DH (октябрь 1997 г.). «Активные участки эукариотической протеасомы 20 с и их участие в обработке предшественников субъединиц» . Журнал биологической химии . 272 (40): 25200–9. doi : 10.1074/jbc.272.40.25200 . PMID 9312134 .
- ^ Jump up to: а беременный Padmanabhan A, Vuong SA, Hochstrasser M (март 2016 г.). «Сборка эволюционно консервативной альтернативной изоформы протеасом в клетках человека» . Сотовые отчеты . 14 (12): 2962–74. doi : 10.1016/j.celrep.2016.02.068 . PMC 4828729 . PMID 26997268 .
- ^ Velichutina I, Connerly PL, Arendt CS, Li X, Hochstrasser M (февраль 2004 г.). «Пластичность в сборе протеасомы Eucaryotic 20S, выявленная удалением субъединицы в дрожжах» . Embo Journal . 23 (3): 500–10. doi : 10.1038/sj.emboj.7600059 . PMC 1271798 . PMID 14739934 .
- ^ Kusmierczyk AR, Kunjappu MJ, Funakoshi M, Hochstrasser M (март 2008 г.). «Мультимерный коэффициент сборки контролирует формирование альтернативных протеасомов 20S». Природа структурная и молекулярная биология . 15 (3): 237–44. doi : 10.1038/nsmb.1389 . PMID 18278055 . S2CID 21181637 .
- ^ Zwickl P, Ng D, Woo KM, Klenk HP, Goldberg AL (сентябрь 1999 г.). «Архебактериальная АТФаза, гомологичная АТФазы в протеасоме эукариотической 26 с, активирует распад белка на 20 с протеасомов» . Журнал биологической химии . 274 (37): 26008–14. doi : 10.1074/jbc.274.37.26008 . PMID 10473546 .
- ^ Jump up to: а беременный в дюймовый Смит Д.М., Кафри Г., Ченг Ю., Нг Д., Вальц Т., Голдберг А.Л. (декабрь 2005 г.). «Связывание АТФ с PAN или АТФазами 26S вызывает ассоциацию с протеасомой 20S, открытием ворот и транслокацией развернутых белков» . Молекулярная клетка . 20 (5): 687–98. doi : 10.1016/j.molcel.2005.10.019 . PMID 16337593 .
- ^ Jump up to: а беременный в дюймовый и Liu CW, Li X, Thompson D, Wording K, Chang TL, Tang Z, Yu H, Thomas PJ, Demartino GN (октябрь 2006 г.). «Связывание АТФ и гидролиз АТФ играют четкую роль в функции протеасомы 26S» . Молекулярная клетка . 24 (1): 39–50. doi : 10.1016/j.molcel.2006.08.025 . PMC 3951175 . PMID 17018291 .
- ^ Lam Ya, Lawson TG, Velayutham M, Zweier JL, Pickart CM (апрель 2002 г.). «Протеасомная субъединица АТФазы распознает сигнал деградации полиубикитина». Природа . 416 (6882): 763–7. Bibcode : 2002natur.416..763L . doi : 10.1038/416763a . PMID 11961560 . S2CID 4421764 .
- ^ Jump up to: а беременный Beck F, Unverdorben P, Bohn S, Schweitzer A, Pfeifer G, Sakata E, Nickell S, Plitzko JM, Villa E, Baumeister W, Förster F (сентябрь 2012 г.). «Структурная модель протеасомы дрожжей 26S почти атомного разрешения» . Труды Национальной академии наук Соединенных Штатов Америки . 109 (37): 14870–5. BIBCODE : 2012PNAS..10914870B . doi : 10.1073/pnas.1213333109 . PMC 3443124 . PMID 22927375 .
- ^ Jump up to: а беременный Ландер Г.К., Эстрон Е., Матискила М.Е., Башор С., Ногалес Э, Мартин А (февраль 2012 г.). «Полная архитектура субъединицы регуляторной частицы протеасомы» . Природа . 482 (7384): 186–91. Bibcode : 2012natur.482..186L . doi : 10.1038/nature10774 . PMC 3285539 . PMID 22237024 .
- ^ Lasker K, Förster F, Bohn S, Walzthoeni T, Villa E, Unverdorben P, Beck F, Aebersold R, Sali A, Baumeister W (январь 2012 г.). «Молекулярная архитектура протеасомного голокомплекса 26S, определяемое интегративным подходом» . Труды Национальной академии наук Соединенных Штатов Америки . 109 (5): 1380–7. doi : 10.1073/pnas.1120559109 . PMC 3277140 . PMID 22307589 .
- ^ Jump up to: а беременный в Chen S, Wu J, Lu Y, Ma YB, Lee BH, Yu Z, Ouyang Q, Finley DJ, Kirschner MW, Mao Y (ноябрь 2016 г.). «Структурная основа для динамической регуляции протеасомы человека 26» . Труды Национальной академии наук Соединенных Штатов Америки . 113 (46): 12991–12996. BIBCODE : 2016PNAS..11312991C . doi : 10.1073/pnas.1614614113 . PMC 5135334 . PMID 27791164 .
- ^ Huang X, Luan B, Wu J, Shi Y (сентябрь 2016 г.). «Атомная структура протеасомы человека 26S». Природа структурная и молекулярная биология . 23 (9): 778–785. doi : 10.1038/nsmb.3273 . PMID 27428775 . S2CID 21909333 .
- ^ Schweitzer A, Aufderheide A, Rudack T, Beck F, Pfeifer G, Plitzko JM, Sakata E, Schulten K, Förster F, Baumeister W (июль 2016 г.). «Структура протеасомы человека 26 с разрешения 3,9 Å» . Труды Национальной академии наук Соединенных Штатов Америки . 113 (28): 7816–7821. BIBCODE : 2016PNAS..11312991C . doi : 10.1073/pnas.1614614113 . PMC 5135334 . PMID 27791164 .
- ^ Jump up to: а беременный в дюймовый Zhu Y, Wang WL, Yu D, Ouyang Q, Lu Y, Mao Y (апрель 2018 г.). -управляемой ааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааааа «Структурный механизм для реконструкции нуклеотид Природная связь . 9 (1): 1360. Bibcode : 2018natco ... 9.1360z . doi : 10.1038/s41467-018-03785-w . PMC 5893597 . PMID 29636472 .
- ^ Jump up to: а беременный в Unverdorben P, Beck F, śledź P, Schweitzer A, Pfeifer G, Plitzko JM, Baumeister W, Förster F (апрель 2014). «Глубокая классификация большого набора данных крио-ЭМ определяет конформационный ландшафт протеасомы 26S» . Труды Национальной академии наук Соединенных Штатов Америки . 111 (15): 5544–9. Bibcode : 2014pnas..111.5544u . doi : 10.1073/pnas.1403409111 . PMC 3992697 . PMID 24706844 .
- ^ Śledź P, Unverdorben P, Beck F, Pfeifer G, Schweitzer A, Förster F, Baumeister W (апрель 2013 г.). «Структура протеасомы 26S с границей ATP-γS дает представление о механизме транслокации нуклеотид-зависимой субстрата» . Труды Национальной академии наук Соединенных Штатов Америки . 110 (18): 7264–7269. BIBCODE : 2013PNAS..110.7264S . doi : 10.1073/pnas.1305782110 . PMC 3645540 . PMID 23589842 .
- ^ Matyskiela Me, Lander GC, Martin A (июль 2013 г.). «Конформационное переключение протеасомы 26S обеспечивает деградацию субстрата» . Природа структурная и молекулярная биология . 20 (7): 781–788. doi : 10.1038/nsmb.2616 . PMC 3712289 . PMID 23770819 .
- ^ Lu Y, Wu J, Dong Y, Chen S, Sun S, Ma YB, Ouyang Q, Finley D, Kirschner MW, Mao Y (июль 2017 г.). «Конформационный ландшафт регуляторной частицы протеасомы человека, связанной с P28» . Молекулярная клетка . 67 (2): 322–333.e6. doi : 10.1016/j.molcel.2017.06.007 . PMC 5580496 . PMID 28689658 .
- ^ Келер А., Касчио П., Леггетт Д.С., Ву К.М., Голдберг А.Л., Финли Д. (июнь 2001 г.). «Осевой канал частицы ядра протеасомы управляется АТФазой RPT2 и контролирует как вход субстрата, так и выпуск продукта» . Молекулярная клетка . 7 (6): 1143–52. doi : 10.1016/s1097-2765 (01) 00274-x . PMID 11430818 .
- ^ Förster A, Masters EI, Whitby FG, Robinson H, Hill CP (май 2005). «1.9 Структура активаторного комплекса протеасомы-11 и последствий для взаимодействия протеасом-пен/PA700» . Молекулярная клетка . 18 (5): 589–99. doi : 10.1016/j.molcel.2005.04.016 . PMID 15916965 .
- ^ Witt S, Kwon YD, Sharon M, Felderer K, Beuttler M, Robinson CV, Baumeister W, Jap Bk (июль 2006 г.). «Сборка протеасомы запускает переключатель, необходимый для созревания активного сайта» . Структура 14 (7): 1179–88. doi : 10.1016/j.str.2006.05.019 . PMID 16843899 .
- ^ Krüger E, Kloetzel PM, Enenkel C (2001). «Биогенез протеасом 20S». Биохими . 83 (3–4): 289–93. doi : 10.1016/s0300-9084 (01) 01241-x . PMID 11295488 .
- ^ Мурата С., Ясирода Х, Танака К (февраль 2009 г.). «Молекулярные механизмы сборки протеасом». Природа обзор молекулярной клеточной биологии . 10 (2): 104–115. doi : 10.1038/nrm2630 . PMID 19165213 . S2CID 21263837 .
- ^ Стальф Э., Стивенель Ф., Фуакуканага К., Чжоу М., Фёрмстер Ф., Баумитер В., Блаженное, Робинсон СВИД (июнь 2011 г.). Полем Молекулярный Cel 42 : 637–6 doi : 10.1016/j.mulcel . PMID 21658604 .
- ^ Допустим, Kudo T, Toh-e A, Bonthy K, Saeki Y (июнь 2010 г.). Дискреция сборки. Биофические исследования 396 (4): 1048–1 doi : 10.1016/j . PMID 20471955 .
- ^ Haas Al, согревает JV, Hershko A, Rose IA (март 1982 г.). «Убиквитин-активирующий фермент. Механизм и роль в конъюгации белка-убиквитина» . Журнал биологической химии . 257 (5): 2543–8. doi : 10.1016/s0021-9258 (18) 34958-5 . PMID 6277905 .
- ^ Pickart CM (январь 2000 г.). Признание Embo Journal 19 (1): 94–1 doi : 10.1093/emboj/19.1.1.94 . 1171781PMC PMID 10619848 .
- ^ Risseeuw EP, Daskalchuk TE, Banks TW, Liu E, Cotelesage J, Hellmann H, Estelle M, Somers DE, Crosby WL (июнь 2003 г.). «Анализ взаимодействия белков субъединиц убиквитин E3 лигазы SCF от арабидопсиса» . Заводский журнал . 34 (6): 753–67. doi : 10.1046/j.1365-313x.2003.01768.x . PMID 12795696 .
- ^ Elsasser S, Finley D (август 2005 г.). «Доставка убиквитинированных субстратов в белковые машины». Природная клеточная биология . 7 (8): 742–9. doi : 10.1038/ncb0805-742 . PMID 16056265 . S2CID 21069699 .
- ^ Sadanandom A, Bailey M, Ewan R, Lee J, Nelis S (октябрь 2012 г.). «Система убиквитин-протеемного роста: центральный модификатор передачи сигналов растений» . Новый фитолог . 196 (1): 13–28. doi : 10.1111/j.1469-8137.2012.04266.x . PMID 22897362 .
- ^ Sharp PM, Li WH (1987). «Убиквитиновые гены как парадигма согласованной эволюции тандемных повторов». Журнал молекулярной эволюции . 25 (1): 58–64. Bibcode : 1987jmole..25 ... 58 с . doi : 10.1007/bf02100041 . PMID 3041010 . S2CID 7929162 .
- ^ Pickart CM, Fushman D (декабрь 2004 г.). «Полиубиквитиновые цепи: полимерные белковые сигналы». Современное мнение о химической биологии . 8 (6): 610–16. doi : 10.1016/j.cbpa.2004.09.009 . PMID 15556404 .
- ^ Сюй П., Дуонг Д.М., Сейфрид Н.Т., Ченг Д., Се Й, Роберт Дж., Раш Дж., Хохстрассер М., Финли Д., Пенг Дж (апрель 2009 г.). «Количественная протеомика выявляет функцию нетрадиционных цепей убиквитина при протеасомной деградации» . Клетка . 137 (1): 133–45. doi : 10.1016/j.cell.2009.01.041 . PMC 2668214 . PMID 19345192 .
- ^ Jump up to: а беременный Pickart CM (ноябрь 2000 г.). «Убиквитин в цепях». Тенденции в биохимических науках . 25 (11): 544–8. doi : 10.1016/s0968-0004 (00) 01681-9 . PMID 11084366 .
- ^ Jump up to: а беременный Zhang S, Zou S, Yin D, Zhao L, Finley D, Wu Z, Mao Y (апрель 2022 г.). «USP14-регулируемая аллостерия протеасомы человека с помощью крио-эм с разрешением по времени» . Природа . 605 (7910): 567–574. Bibcode : 2022natur.605..567Z . doi : 10.1038/s41586-022-04671-8 . PMC 9117149 . PMID 35477760 .
- ^ Zhu Q, Wani G, Wang QE, El-Mahdy M, Snapka RM, Wani AA (июль 2005 г.). «Деубиквитинирование протеасомой координируется с транслокацией субстрата для протеолиза in vivo». Экспериментальные исследования клеток . 307 (2): 436–51. doi : 10.1016/j.yexcr.2005.03.031 . PMID 15950624 .
- ^ Венцель Т., Бауместер В. (март 1995 г.). «Конформационные ограничения в деградации белка с помощью протеасомы 20S». Природа структурная биология . 2 (3): 199–204. doi : 10.1038/nsb0395-199 . PMID 7773788 . S2CID 41599619 .
- ^ INOBE T, Fishbain S, Prakash S, Matouschek A (март 2011 г.). «Определение геометрии двухкомпонентного дегрона протеасомы» . Природная химическая биология . 7 (3): 161–7. doi : 10.1038/nchembio.521 . PMC 3129032 . PMID 21278740 .
- ^ Van Der Lee R, Lang B, Kruse K, Gsponer J, Sánchez de Groot N, Huynen MA, Matouschek A, Fuxreiter M, Babu MM (сентябрь 2014 г.). «Внутренние беспорядочные сегменты влияют на период полураспада белка в клетке и во время эволюции» . Сотовые отчеты . 8 (6): 1832–44. doi : 10.1016/j.celrep.2014.07.055 . PMC 4358326 . PMID 25220455 .
- ^ Смит Д.М., Бенарудж Н., Голдберг А (октябрь 2006 г.). «Протеасомы и связанные с ними АТФазы: разрушительная комбинация». Журнал структурной биологии . 156 (1): 72–83. doi : 10.1016/j.jsb.2006.04.012 . PMID 16919475 .
- ^ Хойт М.А., Зич Дж., Такеучи Дж., Чжан М., Говаертс С, Кофе П (апрель 2006 г.). «Глицин-аланин повторяет надлежащий подложку, разворачивающийся протеасомой» . Embo Journal . 25 (8): 1720–9. doi : 10.1038/sj.emboj.7601058 . PMC 1440830 . PMID 16601692 .
- ^ Чжан М., Кофе П (март 2004 г.). «Повторная последовательность белка ядерного антигена 1-кодируемого вирусом Epstein-Barr ядерного белка 1 прерывает обработку субстрата протеасом» . Журнал биологической химии . 279 (10): 8635–41. doi : 10.1074/jbc.m310449200 . PMID 14688254 .
- ^ Semüller E, Lupas A, Stock D, Löwe J, Huber R, Baumeister W (апрель 1995 г.). «Протеасома из термоплазменной Acidophilum: треониновая протеаза». Наука . 268 (5210): 579–82. Bibcode : 1995sci ... 268..579s . doi : 10.1126/science.7725107 . PMID 7725107 .
- ^ Кукс О., Танака К., Голдберг А.Л. (1996). «Структура и функции протеасомов 20 и 26S». Ежегодный обзор биохимии . 65 : 801–47. doi : 10.1146/annurev.bi.65.070196.004101 . PMID 8811196 .
- ^ Groll M, Ditzel L, Löwe J, Stock D, Bochtler M, Bartunik HD, Huber R (апрель 1997 г.). «Структура протеасомы 20S от дрожжей в 2,4 резолюции». Природа . 386 (6624): 463–71. Bibcode : 1997natur.386..463g . doi : 10.1038/386463A0 . PMID 9087403 . S2CID 4261663 .
- ^ Дик Т.П., Нуссбаум А.К., Дег М., Хейнемейер В., Гролл М., Ширл М., Кейлхольц В., Стеванович С., Вольф Д.Х., Хубер Р., Рамминс Х.Г., Шильд Х (октябрь 1998). «Вклад протеасомных бета-субъединиц в расщепление пептидных субстратов, проанализированных с помощью дрожжевых мутантов» . Журнал биологической химии . 273 (40): 25637–46. doi : 10.1074/jbc.273.40.25637 . PMID 9748229 .
- ^ Voges D, Zwickl P, Baumeister W (1999). «Протеасома 26S: молекулярная машина, предназначенная для контролируемого протеолиза». Ежегодный обзор биохимии . 68 (1): 1015–68. doi : 10.1146/annurev.biochem.68.1.1015 . PMID 10872471 .
- ^ Jump up to: а беременный Изнасилование M, Jentsch S (май 2002). «Укус: протеасомная обработка белка». Природная клеточная биология . 4 (5): E113–6. doi : 10.1038/ncb0502-e113 . PMID 11988749 . S2CID 7126477 .
- ^ Изнасилование M, Jentsch S (ноябрь 2004 г.). «Продуктивный разрыв: активация транскрипционных факторов протеасомной обработкой» . Biochimica et Biophysica Acta (BBA) - исследования молекулярных клеток . 1695 (1–3): 209–13. doi : 10.1016/j.bbamcr.2004.09.022 . PMID 15571816 .
- ^ Asher G, Reuven N, Shaul Y (август 2006 г.). «Протеасомы 20S и деградация белка» по умолчанию ». Биологии . 28 (8): 844–9. doi : 10.1002/bies.20447 . PMID 16927316 .
- ^ Zhang M, Pickart CM, Coffino P (апрель 2003 г.). «Детерминанты распознавания протеасомы орнитин декарбоксилазы, убиквитин-независимый субстрат» . Embo Journal . 22 (7): 1488–96. doi : 10.1093/emboj/cdg158 . PMC 152902 . PMID 12660156 .
- ^ Ашер Г, Шаул Ю (август 2005 г.). «Протеасомная деградация P53: полибиквитинирование-это не вся история» . Клеточный цикл . 4 (8): 1015–8. doi : 10.4161/cc.4.8.1900 . PMID 16082197 .
- ^ Jump up to: а беременный Shringarpure R, Grune T, Mehlhase J, Davies KJ (январь 2003 г.). «Конъюгация убиквитина не требуется для деградации окисленных белков с помощью протеасомы» . Журнал биологической химии . 278 (1): 311–8. doi : 10.1074/jbc.m206279200 . PMID 12401807 .
- ^ Jump up to: а беременный в Gille C, Goede A, Schlöetelburg C, Preissner R, Kloetzel PM, Göbel UB, Frömmel C (март 2003 г.). «Комплексное представление о протеасомных последовательностях: последствия для эволюции протеасомы». Журнал молекулярной биологии . 326 (5): 1437–48. doi : 10.1016/s0022-2836 (02) 01470-5 . PMID 12595256 .
- ^ Bochtler M, Ditzel L, Groll M, Hartmann C, Huber R (1999). «Протесома». Ежегодный обзор биофизики и биомолекулярной структуры . 28 (1): 295–317. doi : 10.1146/annurev.biophys.28.1.295 . PMID 10410804 .
- ^ Chesnel F, Bazile F, Pascal A, Kubiak JZ (август 2006 г.). «Диссоциация циклина B от CDK1 предшествует его деградации при инактивации MPF в митотических экстрактах эмбрионов Xenopus laevis» . Клеточный цикл . 5 (15): 1687–98. doi : 10.4161/cc.5.15.3123 . PMID 16921258 .
- ^ Брито Д.А., Ридер К.Л. (июнь 2006 г.). «Митотическое проскальзывание контрольной точки у людей происходит посредством разрушения циклина В в присутствии активной контрольной точки» . Текущая биология . 16 (12): 1194–200. doi : 10.1016/j.cub.2006.04.043 . PMC 2749311 . PMID 16782009 .
- ^ Havens CG, Ho A, Yoshioka N, Dowdy SF (июнь 2006 г.). «Регуляция позднего фазового перехода G1/S и APC CDH1 с помощью реактивных форм кислорода» . Молекулярная и клеточная биология . 26 (12): 4701–11. doi : 10.1128/mcb.00303-06 . PMC 1489138 . PMID 16738333 .
- ^ Башир Т., Доррелло Н.В., Амадор В., Гвардаваккаро Д., Пагано М (март 2004 г.). «Контроль SCF (SKP2-CKS1) убиквитин-лигаза с помощью Ubiquitin-лигазы APC/C (CDH1)». Природа . 428 (6979): 190–3. doi : 10.1038/nature02330 . PMID 15014502 . S2CID 4401971 .
- ^ Higashitsuji H, Liu Y, Mayer RJ, Fujita J (октябрь 2005 г.). «Онкопротеин ганкирин негативно регулирует как р53, так и РБ, усиливая протеасомную деградацию» . Клеточный цикл . 4 (10): 1335–7. doi : 10.4161/cc.4.10.2107 . PMID 16177571 .
- ^ Tarrason Risa, Gabriel; Hurtig, Fredrik; Bray, Sian; Hafner, Anne E.; Harker-Kirschneck, Lena; Faull, Peter; Davis, Colin; Papatziamou, Dimitra; Mutavchiev, Delyan R.; Fan, Catherine; Meneguello, Leticia; Arashiro Pulschen, Andre; Dey, Gautam; Culley, Siân; Kilkenny, Mairi; Souza, Diorge P.; Pellegrini, Luca; de Bruin, Robertus A. M.; Henriques, Ricardo; Snijders, Ambrosius P.; Šarić, Anđela; Lindås, Ann-Christin; Robinson, Nicholas P.; Baum, Buzz (7 August 2020). "The proteasome controls ESCRT-III–mediated cell division in an archaeon". Science. 369 (6504): eaaz2532. doi:10.1126/science.aaz2532. PMC 7116001. PMID 32764038.
- ^ Dharmasiri S, Estelle M (2002). «Роль регулируемого деградации белка в ответе ауксина». Растительная молекулярная биология . 49 (3–4): 401–9. doi : 10.1023/a: 1015203013208 . PMID 12036263 . S2CID 7669386 .
- ^ Вейерс Д., Бенкова Е., Ягер К.Е., Шлерет А., Хаманн Т., Кенц М., Уилмот Дж.С., Рид Дж.В., Юргенс Г. (май 2005). «Специфика развития ответа ауксина парами ARF и регуляторов транскрипции AUX/IAA» . Embo Journal . 24 (10): 1874–85. doi : 10.1038/sj.emboj.7600659 . PMC 1142592 . PMID 15889151 .
- ^ Хаас А.Л., Бабишина О., Уильямс Б., Шварц Л.М. (апрель 1995 г.). «Скоординированная индукция пути конъюгации убиквитина сопровождает программированную в развитии смерть скелетных мышц насекомых» . Журнал биологической химии . 270 (16): 9407–12. doi : 10.1074/jbc.270.16.9407 . PMID 7721865 .
- ^ Schwartz LM, Myer A, Kosz L, Engelstein M, Maier C (октябрь 1990 г.). «Активация экспрессии гена полиубикитина во время программированной в развитии гибели клеток». Нейрон . 5 (4): 411–9. doi : 10.1016/0896-6273 (90) 90080-y . PMID 2169771 . S2CID 33829749 .
- ^ Löw P, Bussell K, Dawson SP, Billett MA, Mayer RJ, Reynolds SE (январь 1997 г.). «Экспрессия 26S -протеасомной субъединицы ATPase, MS73, в мышцах, которые подвергаются программированной гибели клеток в развитии, и ее контроль с помощью экдистероидных гормонов у насекомых Manduca Sexta» . Письма Febs . 400 (3): 345–9. doi : 10.1016/s0014-5793 (96) 01413-5 . PMID 9009228 . S2CID 10873052 .
- ^ Питцер Ф., Дантес А., Фукс Т., Бауместер В., Амстердам А (сентябрь 1996 г.). «Удаление протеасомов из ядра и их накопление в апоптотических пузырьках во время запрограммированной гибели клеток». Письма Febs . 394 (1): 47–50. doi : 10.1016/0014-5793 (96) 00920-9 . PMID 8925925 . S2CID 29256092 .
- ^ Jump up to: а беременный Adams J, Palombella VJ, Sausville EA, Johnson J, Destree A, Lazarus DD, Maas J, Pien CS, Prakash S, Elliott PJ (июнь 1999 г.). «Ингибиторы протеасом: новый класс мощных и эффективных противоопухолевых агентов». РАНКА . 59 (11): 2615–22. PMID 10363983 .
- ^ Орловский Р.З. (апрель 1999 г.). «Роль убиквитин-протеасомного пути при апоптозе» . Гибель клеток и дифференциация . 6 (4): 303–13. doi : 10.1038/sj.cdd.4400505 . PMID 10381632 .
- ^ Garrido C, Brunet M, Didelot C, Zermati Y, Schmitt E, Kroemer G (ноябрь 2006 г.). «Белки теплового шока 27 и 70: антиапоптотические белки с онкогенными свойствами» . Клеточный цикл . 5 (22): 2592–601. doi : 10.4161/cc.5.22.3448 . PMID 17106261 .
- ^ Park SH, Bolender N, Eisele F, Kostova Z, Takeuchi J, Coffino P, Wolf DH (январь 2007 г.). «Субъект-инфурический механизм цитоплазматической машины HSP70 неправильно свернул и импортирует белки для импортирования эндоплазматического ретикулума для деградации через систему убиквитин-протеасом» . Молекулярная биология клетки . 18 (1): 153–65. doi : 10.1091/mbc.e06-04-0338 . PMC 1751312 . PMID 17065559 .
- ^ Dai Q, Qian SB, Li HH, McDonough H, Borchers C, Huang D, Takayama S, Younger JM, Ren Hy, Cyr DM, Patterson C (ноябрь 2005 г.). «Регуляция пути деградации белка контроля цитоплазматического качества BAG2» . Журнал биологической химии . 280 (46): 38673–81. doi : 10.1074/jbc.m507986200 . PMID 16169850 .
- ^ Bader N, Grune T (2006). «Окисление белка и протеолиз». Биологическая химия . 387 (10–11): 1351–5. doi : 10.1515/bc.2006.169 . PMID 17081106 . S2CID 30385354 .
- ^ Дэвис К.Дж. (2003). «Разложение окисленных белков по протеасоме 20S». Биохими . 83 (3–4): 301–10. doi : 10.1016/s0300-9084 (01) 01250-0 . PMID 11295490 .
- ^ Jump up to: а беременный Lehman NL (сентябрь 2009 г.). «Система протеасома убиквитин в невропатологии» . Acta Neuropathologica . 118 (3): 329–47. doi : 10.1007/s00401-009-0560-x . PMC 2716447 . PMID 19597829 .
- ^ McNaught KS, Jackson T, Jnobaptiste R, Kapustin A, Olanow CW (май 2006 г.). «Протеасомная дисфункция при болезни Паркинсона» . Неврология . 66 (10 Suppl 4): S37–49. doi : 10.1212/01.wnl.0000221745.58886.2e . PMID 16717251 .
- ^ Шарма Н., Брэндис К.А., Эррера С.К., Джонсон Б.Е., Вайдья Т., Шреста Р., Деббурман С.К. (2006). «Модель дрожжей альфа-синуклеина: токсичность, усиленная нарушением протеасомы и окислительного стресса». Журнал молекулярной нейробиологии . 28 (2): 161–78. doi : 10.1385/jmn: 28: 2: 161 . PMID 16679556 . S2CID 27762513 .
- ^ Мурата С., Сасаки К., Кишимото Т., Нива С., Хаяси Х, Такахама Ю., Танака К (июнь 2007 г.). «Регуляция развития CD8+ T-клеток с помощью специфичных для тимуса протеасомов». Наука . 316 (5829): 1349–53. Bibcode : 2007sci ... 316.1349m . doi : 10.1126/science.1141915 . PMID 17540904 . S2CID 37185716 .
- ^ Cascio P, Hilton C, Kisselev AF, Rock KL, Goldberg AL (май 2001). «Протеасомы 26S и иммунопротеасом продуцируют в основном N-вытянутые версии антигенного пептида» . Embo Journal . 20 (10): 2357–66. doi : 10.1093/emboj/20.10.2357 . PMC 125470 . PMID 11350924 .
- ^ Mallery DL, McEwan WA, Bidgood SR, Towers GJ, Johnson CM, James LC (ноябрь 2010 г.). «Антитела опосредуют внутриклеточный иммунитет посредством трехстороннего мотива, содержащего 21 (TRIM21)» . Труды Национальной академии наук Соединенных Штатов Америки . 107 (46): 19985–19990. Bibcode : 2010pnas..10719985M . doi : 10.1073/pnas.1014074107 . PMC 2993423 . PMID 21045130 .
- ^ Fenteany G, Standaert RF, Lane WS, Choi S, Corey EJ, Schreiber SL (май 1995). «Ингибирование активности протеасом и субъединичная амино-концевая треониновая модификация лактацистином». Наука . 268 (5211): 726–31. Bibcode : 1995sci ... 268..726f . doi : 10.1126/science.7732382 . PMID 7732382 . S2CID 37779687 .
- ^ Соединенные Штаты Управление по контролю за продуктами и лекарствами Псюнкер Архивировал 19 февраля 2007 года на машине Wayback 13 мая 2003 г. Дата доступа 29 декабря 2006 года. См. Также информационную страницу FDA Velcade .
- ^ Фишер Р.И., Бернштейн С.Х., Кал Б.С., Джулбегович Б., Робертсон М.Дж., Де Вос С., Эпнер Е., Кришнан А., Леонард Дж.П., Лониал С., Стадтмауэр Е.А., О'Коннор О.А., Ши Х, Борал Ал, Гой (октябрь 2006 г. ) «Многоцентровое исследование фазы II Bortezomib у пациентов с рецидивирующей или рефрактерной мантийной клеточной лимфомой» . Журнал клинической онкологии . 24 (30): 4867–74. doi : 10.1200/jco.2006.07.9665 . PMID 17001068 .
- ^ Jakob C, Egerer K, Liebisch P, Türkmen S, Zavrski I, Kackelkorn U, Heider U, Kaiser M, Fleissner C, Sterz J, Kleeberg L, Feist E, Burmester GR, Kloetzel PM, Sezer O (март 2007 г.). «Циркуляционные уровни протеасомы являются независимым прогностическим фактором для выживания при множественной миеломе» . Кровь . 109 (5): 2100–5. doi : 10.1182/blood-2006-04-016360 . PMID 17095627 .
- ^ Shah SA, Potter MW, McDade TP, Ricciardi R, Perugini RA, Elliott PJ, Adams J, Callery MP (2001). «Ингибирование протеасомы 26S вызывает апоптоз и ограничивает рост рака поджелудочной железы человека». Журнал сотовой биохимии . 82 (1): 110–22. doi : 10.1002/jcb.1150 . PMID 11400168 . S2CID 21223980 .
- ^ Nawrocki St, Sweeney-Gotsch B, Takamori R, McConkey DJ (январь 2004 г.). «Ингибитор протеасомы Bortezomib усиливает активность доцетаксела у ортотопических ксенотрансплантатов опухоли поджелудочной железы человека» . Молекулярная терапия рака . 3 (1): 59–70. doi : 10.1158/1535-7163.59.3.1 . PMID 14749476 . S2CID 38429730 .
- ^ Schenkein D (июнь 2002 г.). «Ингибиторы протеасом при лечении злокачественных новообразований B-клеток». Клиническая лимфома . 3 (1): 49–55. doi : 10.3816/clm.2002.n.011 . PMID 12141956 .
- ^ О'Коннор О.А., Райт Дж., Московиц С., Муззи Дж., Макгрегор-Корелли Б., Стейблфилд М., Страус Д., Портлок С., Хэмлин П., Чой Э., Дюмереску О., Эсселтин Д., Треху Е., Адамс Дж., Шенкеин Д., Зеленец AD (февраль 2005 г.). «Клинический опыт фазы II с новым ингибитором протеасомы Bortezomib у пациентов с индолентной неходжкинской лимфомой и мантийной клеточной лимфомой». Журнал клинической онкологии . 23 (4): 676–84. doi : 10.1200/jco.2005.02.050 . PMID 15613699 .
- ^ Мессингер YH, Gaynon PS, Sposto R, Van der Giessen J, Eckroth E, Malvar J, Bostrom BC (июль 2012 г.). «Bortezomib с химиотерапией очень активен в прогрессирующем B-прекурсоре острого лимфобластного лейкоза: терапевтические достижения в исследовании детской лейкозы и лимфомы (TACL)» . Кровь . 120 (2): 285–90. doi : 10.1182/blood-2012-04-418640 . PMID 22653976 .
- ^ Lambrou Gi, Papadimitriou L, Chrousos GP, Vlahopoulos SA (апрель 2012 г.). «Ингибитор глюкокортикоидов и протеасомы на лейкозный лимфобласт: множественные, разнообразные сигналы, сходящиеся на нескольких ключевых регуляторах нижестоящих». Молекулярная и клеточная эндокринология . 351 (2): 142–51. doi : 10.1016/j.mce.2012.01.003 . PMID 22273806 . S2CID 28749125 .
- ^ Schmidtke G, Holzhütter Hg, Bogyo M, Kairies N, Groll M, De Giuli R, Emch S, Groettrup M (декабрь 1999). «Как ингибитор протеазы ВИЧ-I модулирует активность протеасом» . Журнал биологической химии . 274 (50): 35734–40. doi : 10.1074/jbc.274.50.35734 . PMID 10585454 .
- ^ Laurent N, De Boüard S, Guillamo JS, Christov C, Zini R, Jouault H, Andre P, Lotteau V, Peschanski M (февраль 2004 г.). «Влияние ингибитора протеасомы ритонавира на рост глиомы in vitro и in vivo» . Молекулярная терапия рака . 3 (2): 129–36. doi : 10.1158/1535-7163.129.3.2 . PMID 14985453 . S2CID 24423806 .
- ^ Zollner TM, Podda M, Pien C, Elliott PJ, Kaufmann R, Boehncke WH (март 2002 г.). «Ингибирование протеасомы уменьшает активацию Т-клеток, опосредованную супеантиген, и тяжесть псориаза в модели SCID-HU» . Журнал клинических исследований . 109 (5): 671–9. doi : 10.1172/jci12736 . PMC 150886 . PMID 11877475 .
- ^ Elliott PJ, Pien CS, McCormack TA, Chapman ID, Adams J (август 1999 г.). «Ингибирование протеасомы: новый механизм борьбы с астмой» . Журнал аллергии и клинической иммунологии . 104 (2 пт 1): 294–300. doi : 10.1016/s0091-6749 (99) 70369-6 . PMID 10452747 .
- ^ Защитный M, Florea Bi, Menenendz-Benito V, Maynard CJ, White MD, из Linden WA, Nuy Am, Court, Borkers CR, из Лег-Т.А., Леотербург М.А., Оваа Х, Филиппова Д.В. Dantuma NP, Aclosure HS (Novemer 2006). «Флуоресцентный спектр Draad Spectrum, ингибирующий лабораторных протеасам in vitro и in vivo» . Химия и билогия . 13 (11): 1217–26. doi : 10,1016/j.chembol . HDL : 1887/65477 . PMID 17114003 .
- ^ Kleiger G, мэр T (июнь 2014 г.). «Опасное путешествие: экскурсия по системе убиквитин-протеасома» . Тенденции в клеточной биологии . 24 (6): 352–9. doi : 10.1016/j.tcb.2013.12.003 . PMC 4037451 . PMID 24457024 .
- ^ Голдберг А.Л., Стейн Р., Адамс Дж. (Август 1995 г.). «Новое понимание функции протеасомы: от Archaebacteria до разработки лекарств» . Химия и биология . 2 (8): 503–8. doi : 10.1016/1074-5521 (95) 90182-5 . PMID 9383453 .
- ^ Сулистио Я, Хиз К (январь 2015 г.). «Система убиквитин -протеасом и дерегуляция молекулярной шаперона при болезни Альцгеймера». Молекулярная нейробиология . 53 (2): 905–31. doi : 10.1007/s12035-014-9063-4 . PMID 25561438 . S2CID 14103185 .
- ^ Ortega Z, Lucas JJ (2014). «Убив-протеемное участие в болезни Хантингтона» . Границы в молекулярной нейробиологии . 7 : 77. doi : 10.3389/fnmol.2014.00077 . PMC 4179678 . PMID 25324717 .
- ^ Сандри М, Роббинс Дж (июнь 2014 г.). «Протеотоксичность: недооцененная патология при болезни сердца» . Журнал молекулярной и клеточной кардиологии . 71 : 3–10. doi : 10.1016/j.yjmcc.2013.12.015 . PMC 4011959 . PMID 24380730 .
- ^ Дрюс О, Тагтмейер H (декабрь 2014 г.). «Нацеливание на систему убиквитин -протеасом при сердечных заболеваниях: основа для новых терапевтических стратегий» . Антиоксиданты и окислительно -восстановительная передача сигналов . 21 (17): 2322–43. doi : 10.1089/ars.2013.5823 . PMC 4241867 . PMID 25133688 .
- ^ Wang ZV, Hill Ja (февраль 2015 г.). «Контроль качества белка и метаболизм: двунаправленный контроль в сердце» . Клеточный метаболизм . 21 (2): 215–26. doi : 10.1016/j.cmet.2015.01.016 . PMC 4317573 . PMID 25651176 .
- ^ Jump up to: а беременный Карин М, Дельхасе М (февраль 2000 г.). «I Kappa B-киназа (IKK) и NF-Kappa B: ключевые элементы провоспалительной передачи сигналов». Семинары по иммунологии . 12 (1): 85–98. doi : 10.1006/smim.2000.0210 . PMID 10723801 .
- ^ Эмолаева М.А., Даховник А., Шумахер Б. (январь 2015 г.). «Механизмы контроля качества в клеточных и системных реакциях на повреждение ДНК» . Обзоры исследований старения . 23 (pt a): 3–11. doi : 10.1016/j.arr.2014.12.009 . PMC 4886828 . PMID 25560147 .
- ^ Checler F, Da Costa CA, Ancolio K, Chevaler N, Lopez-Perez E, Marabaud P (июль 2000 г.). «Роль протеазера при болезни Альцгеймера » Biochimica et Biophysica Acta (BB) - Молекулярная основа болезни 1502 (1): 133–8 Doi : 10.1016/s0925-4439 (00) 00039-9 10899438PMID
- ^ Jump up to: а беременный Chung KK, Dawson VL, Dawson TM (ноябрь 2001 г.). «Роль убиквитин-протеасомный путь при болезни Паркинсона и других нейродегенеративных расстройствах». Тенденции в нейронауках . 24 (11 Suppl): S7–14. doi : 10.1016/s0166-2236 (00) 01998-6 . PMID 11881748 . S2CID 2211658 .
- ^ Jump up to: а беременный Икеда К., Акияма Х., Арай Т, Уэно Х, Цучия К, Косака К (июль 2002 г.). «Морфометрическая переоценка системы моторных нейронов болезни Пика и амиотрофического бокового склероза с деменцией». Acta Neuropathologica . 104 (1): 21–8. doi : 10.1007/s00401-001-0513-5 . PMID 12070660 . S2CID 22396490 .
- ^ Mankaka H, Kato T, Kurita T, Table T, Shikad Y, Keaii K, Plain T, Suzuki Y, Nihei K, Shasaki H (май 1992). Увеличение рынка в Creutzfed. Нейробиологические буквы 139 (1): 47–9. doi : 0304-3 10.1016 / PMID 1328965 . S2CID 28190967 .
- ^ Мэтьюз К.Д., Мур С.А. (январь 2003 г.). «Мышечная дистрофия Текущая неврология и неврологические отчеты . 3 (1): 78–85. doi : 10.1007/s11910-003-0042-9 . PMID 12507416 . S2CID 5780576 .
- ^ Mayer RJ (март 2003 г.). «От нейродегенерации до нейрохомеостаза: роль убиквитина». News News & Perspectives . 16 (2): 103–8. doi : 10.1358/dnp.2003.16.2.829327 . PMID 12792671 .
- ^ Calise J, Powell Sr (февраль 2013 г.). «Система протеасом убиквитин и ишемия миокарда» . Американский журнал физиологии. Сердечная и циркуляторная физиология . 304 (3): H337–49. doi : 10.1152/ajpheart.00604.2012 . PMC 3774499 . PMID 23220331 .
- ^ Predmore JM, Wang P, Davis F, Bartolone S, Westfall MV, Dyke DB, Pagani F, Powell SR, Day SM (март 2010 г.). «Дисфункция убиквитина протеасом в гипертрофических и дилатационных кардиомиопатиях человека» . Циркуляция . 121 (8): 997–1004. doi : 10.1161/circulationaha.109.904557 . PMC 2857348 . PMID 20159828 .
- ^ Пауэлл С.Р. (июль 2006 г.). «Система убиквитин-протеемного протеасома в физиологии и патологии сердца». Американский журнал физиологии. Сердечная и циркуляторная физиология . 291 (1): H1 - H19. doi : 10.1152/ajpheart.00062.2006 . PMID 16501026 . S2CID 7073263 .
- ^ Адамс Дж (апрель 2003 г.). «Потенциал ингибирования протеасом при лечении рака». Drug Discovery сегодня . 8 (7): 307–15. doi : 10.1016/s1359-6446 (03) 02647-3 . PMID 12654543 .
- ^ Бен-Мерия Y (январь 2002 г.). «Регуляторные функции убиквитинирования в иммунной системе». Природа иммунология . 3 (1): 20–6. doi : 10.1038/ni0102-20 . PMID 11753406 . S2CID 26973319 .
- ^ Эгерер К., Кукелкорн У., Рудольф П.Е., Рюкерт Дж.С., Дёрнер Т., Бурместер Г.Р., Клотцель П.М., Файст Э (октябрь 2002 г.). «Циркулирующие протеасомы являются маркерами повреждения клеток и иммунологической активности при аутоиммунных заболеваниях». Журнал ревматологии . 29 (10): 2045–52. PMID 12375310 .
Дальнейшее чтение
[ редактировать ]- Glickman MH, Adir N (январь 2004 г.). «Протеасома и тонкий баланс между разрушением и спасением» . PLOS Биология . 2 (1): E13. doi : 10.1371/journal.pbio.0020013 . PMC 314468 . PMID 14737189 .
- Протеасома дрожжей 26 с списком субъединиц и картин
- Ciechanover A (сентябрь 2005 г.). «Ранняя работа над системой протеасомы убиквитина, интервью с Aaron Ciechanover. Интервью CDD» . Гибель клеток и дифференциация . 12 (9): 1167–77. doi : 10.1038/sj.cdd.4401691 . PMID 16094393 .
- Хершко А (сентябрь 2005 г.). «Ранняя работа над системой протеасомы убиквитина, интервью с Avram Hershko. Интервью CDD» . Гибель клеток и дифференциация . 12 (9): 1158–61. doi : 10.1038/sj.cdd.4401709 . PMID 16094391 .
- Роза I (сентябрь 2005 г.). «Ранняя работа над системой протеасомы убиквитина, интервью с Ирвином Роуз. Интервью CDD» . Гибель клеток и дифференциация . 12 (9): 1162–6. doi : 10.1038/sj.cdd.4401700 . PMID 16094392 .
- CVEK B, DVORAK Z (2007). «Целью ядерного фактора-каппаба и протеасомы дитиокарбаматными комплексами с металлами» . Текущий фармацевтический дизайн . 13 (30): 3155–67. doi : 10.2174/138161207782110390 . PMID 17979756 . Архивировано из оригинала 29 июля 2012 года.
Внешние ссылки
[ редактировать ]- Руководство по номенклатуре по протеасоме
- 3D -протеасомные структуры в банке данных EM (EMDB)
- Ключевые моменты протеасомы архивировали 17 ноября 2020 года на Wayback Machine функции