Синтетическая биология

| Часть серии статей о |
| Синтетическая биология |
|---|
| Синтетические биологические цепи |
| Genome editing |
| Artificial cells |
| Xenobiology |
| Other topics |
Синтетическая биология ( SynBio ) — это междисциплинарная область науки, которая фокусируется на живых системах и организмах и применяет инженерные принципы для разработки новых биологических частей, устройств и систем или для перепроектирования существующих систем, встречающихся в природе. [1]
Это отрасль науки, которая охватывает широкий спектр методологий из различных дисциплин, таких как биотехнология , биоматериалы , материаловедение/инженерия , генная инженерия , молекулярная биология , молекулярная инженерия , системная биология , мембранная наука , биофизика , химическая и биологическая инженерия . электротехника и вычислительная техника , техника управления и эволюционная биология .
Это включает в себя проектирование и создание биологических модулей , биологических систем и биологических машин или перепроектирование существующих биологических систем для полезных целей. [2]
Кроме того, это отрасль науки, которая фокусируется на новых способностях инженерии существующих организмов с целью перепроектирования их для полезных целей. [3]
In order to produce predictable and robust systems with novel functionalities that do not already exist in nature, it is also necessary to apply the engineering paradigm of systems design to biological systems. According to the European Commission, this possibly involves a molecular assembler based on biomolecular systems such as the ribosome.[4]
History[edit]
1910: First identifiable use of the term synthetic biology in Stéphane Leduc's publication Théorie physico-chimique de la vie et générations spontanées.[5] He also noted this term in another publication, La Biologie Synthétique in 1912.[6]
1944: Canadian-American scientist Oswald Avery shows that DNA is the material of which genes and chromosomes are made. This becomes the bedrock on which all subsequent genetic research is built.[7]
1953: Francis Crick and James Watson publish the structure of the DNA in Nature.
1961: Jacob and Monod postulate cellular regulation by molecular networks from their study of the lac operon in E. coli and envisioned the ability to assemble new systems from molecular components.[8]
1973: First molecular cloning and amplification of DNA in a plasmid is published in P.N.A.S. by Cohen, Boyer et al. constituting the dawn of synthetic biology.[9]
1978: Arber, Nathans and Smith win the Nobel Prize in Physiology or Medicine for the discovery of restriction enzymes, leading Szybalski to offer an editorial comment in the journal Gene:
The work on restriction nucleases not only permits us easily to construct recombinant DNA molecules and to analyze individual genes, but also has led us into the new era of synthetic biology where not only existing genes are described and analyzed but also new gene arrangements can be constructed and evaluated.[10]
1988: First DNA amplification by the polymerase chain reaction (PCR) using a thermostable DNA polymerase is published in Science by Mullis et al.[11] This obviated adding new DNA polymerase after each PCR cycle, thus greatly simplifying DNA mutagenesis and assembly.
2000: Two papers in Nature report synthetic biological circuits, a genetic toggle switch and a biological clock, by combining genes within E. coli cells.[12][13]
2003: The most widely used standardized DNA parts, BioBrick plasmids, are invented by Tom Knight.[14] These parts will become central to the International Genetically Engineered Machine (iGEM) competition founded at MIT in the following year.
2003: Researchers engineer an artemisinin precursor pathway in E. coli.[15]
2004: First international conference for synthetic biology, Synthetic Biology 1.0 (SB1.0) is held at MIT.
2005: Researchers develop a light-sensing circuit in E. coli.[16] Another group designs circuits capable of multicellular pattern formation.[17]
2006: Researchers engineer a synthetic circuit that promotes bacterial invasion of tumour cells.[18]
2010: Researchers publish in Science the first synthetic bacterial genome, called M. mycoides JCVI-syn1.0.[19][20] The genome is made from chemically-synthesized DNA using yeast recombination.
2011: Functional synthetic chromosome arms are engineered in yeast.[21]
2012: Charpentier and Doudna labs publish in Science the programming of CRISPR-Cas9 bacterial immunity for targeting DNA cleavage.[22] This technology greatly simplified and expanded eukaryotic gene editing.
2019: Scientists at ETH Zurich report the creation of the first bacterial genome, named Caulobacter ethensis-2.0, made entirely by a computer, although a related viable form of C. ethensis-2.0 does not yet exist.[23][24]
2019: Researchers report the production of a new synthetic (possibly artificial) form of viable life, a variant of the bacteria Escherichia coli, by reducing the natural number of 64 codons in the bacterial genome to 59 codons instead, in order to encode 20 amino acids.[25][26]
2020: Scientists created the first xenobot, a programmable synthetic organism derived from frog cells and designed by AI.[27]
2021: Scientists reported that xenobots are able to self-replicate by gathering loose cells in the environment and then forming new xenobot.[28]
Perspectives[edit]
It is a field whose scope is expanding in terms of systems integration, engineered organisms, and practical findings.[1]
Engineers view biology as technology (in other words, a given system includes biotechnology or its biological engineering).[29] Synthetic biology includes the broad redefinition and expansion of biotechnology, with the ultimate goal of being able to design and build engineered live biological systems that process information, manipulate chemicals, fabricate materials and structures, produce energy, provide food, and maintain and enhance human health, as well as advance fundamental knowledge of biological systems and our environment.[30]
Researchers and companies working in synthetic biology are using nature's power to solve issues in agriculture, manufacturing, and medicine.[3]
Due to more powerful genetic engineering capabilities and decreased DNA synthesis and sequencing costs, the field of synthetic biology is rapidly growing. In 2016, more than 350 companies across 40 countries were actively engaged in synthetic biology applications; all these companies had an estimated net worth of $3.9 billion in the global market.[31] Synthetic biology currently has no generally accepted definition. Here are a few examples:
It is the science of emerging genetic and physical engineering to produce new (and, therefore, synthetic) life forms. To develop organisms with novel or enhanced characteristics, this emerging field of study combines biology, engineering, and related disciplines' knowledge and techniques to design chemically synthesised DNA.[32][33]
Biomolecular engineering includes approaches that aim to create a toolkit of functional units that can be introduced to present new technological functions in living cells. Genetic engineering includes approaches to construct synthetic chromosomes or minimal organisms like Mycoplasma laboratorium.
Biomolecular design refers to the general idea of de novo design and additive combination of biomolecular components. Each of these approaches shares a similar task: to develop a more synthetic entity at a higher level of complexity by inventively manipulating a simpler part at the preceding level.[34][35] Optimizing these exogenous pathways in unnatural systems takes iterative fine-tuning of the individual biomolecular components to select the highest concentrations of the desired product.[36]
On the other hand, "re-writers" are synthetic biologists interested in testing the irreducibility of biological systems. Due to the complexity of natural biological systems, it would be simpler to rebuild the natural systems of interest from the ground up; to provide engineered surrogates that are easier to comprehend, control and manipulate.[37] Re-writers draw inspiration from refactoring, a process sometimes used to improve computer software.
Categories[edit]
Bioengineering, synthetic genomics, protocell synthetic biology, unconventional molecular biology, and in silico techniques are the five categories of synthetic biology.[38]
It is necessary to review the distinctions and analogies between the categories of synthetic biology for its social and ethical assessment, to distinguish between issues affecting the whole field and particular to a specific one.[38]
Bioengineering[edit]
The subfield of bioengineering concentrates on creating novel metabolic and regulatory pathways, and is currently the one that likely draws the attention of most researchers and funding. It is primarily motivated by the desire to establish biotechnology as a legitimate engineering discipline. When referring to this area of synthetic biology, the word "bioengineering" should not be confused with "traditional genetic engineering", which involves introducing a single transgene into the intended organism. Bioengineers adapted synthetic biology to provide a substantially more integrated perspective on how to alter organisms or metabolic systems.[38]
A typical example of single-gene genetic engineering is the insertion of the human insulin gene into bacteria to create transgenic proteins. The creation of whole new signalling pathways, containing numerous genes and regulatory components (such as an oscillator circuit to initiate the periodic production of green fluorescent protein (GFP) in mammalian cells), is known as bioengineering as part of synthetic biology.[38]
By utilising simplified and abstracted metabolic and regulatory modules as well as other standardized parts that may be freely combined to create new pathways or creatures, bioengineering aims to create innovative biological systems. In addition to creating infinite opportunities for novel applications, this strategy is anticipated to make bioengineering more predictable and controllable than traditional biotechnology.[38]
Synthetic genomics[edit]
The formation of animals with a chemically manufactured (minimal) genome is another facet of synthetic biology that is highlighted by synthetic genomics. This area of synthetic biology has been made possible by ongoing advancements in DNA synthesis technology, which now makes it feasible to produce DNA molecules with thousands of base pairs at a reasonable cost. The goal is to combine these molecules into complete genomes and transplant them into living cells, replacing the host cell's genome and reprogramming its metabolism to perform different functions.[38]
Scientists have previously demonstrated the potential of this approach by creating infectious viruses by synthesising the genomes of multiple viruses. These significant advances in science and technology triggered the initial public concerns concerning the risks associated with this technology.[38]
A simple genome might also work as a "chassis genome" that could be enlarged quickly by gene inclusion created for particular tasks. Such "chassis creatures" would be more suited for the insertion of new functions than wild organisms since they would have fewer biological pathways that could potentially conflict with the new functionalities in addition to having specific insertion sites. Synthetic genomics strives to create creatures with novel "architectures," much like the bioengineering method. It adopts an integrative or holistic perspective of the organism. In this case, the objective is the creation of chassis genomes based on necessary genes and other required DNA sequences rather than the design of metabolic or regulatory pathways based on abstract criteria.[38]
Protocell synthetic biology[edit]
The in vitro generation of synthetic cells is the protocell branch of synthetic biology. Lipid vesicles, which have all the necessary components to function as a complete system, can be used to create these artificial cells. In the end, these synthetic cells should meet the requirements for being deemed alive, namely the capacity for self-replication, self-maintenance, and evolution. The protocell technique has this as its end aim, however there are other intermediary steps that fall short of meeting all the criteria for a living cell. In order to carry out a specific function, these lipid vesicles contain cell extracts or more specific sets of biological macromolecules and complex structures, such as enzymes, nucleic acids, or ribosomes. For instance, liposomes may carry out particular polymerase chain reactions or synthesise a particular protein.[38]
Protocell synthetic biology takes artificial life one step closer to reality by eventually synthesizing not only the genome but also every component of the cell in vitro, as opposed to the synthetic genomics approach, which relies on coercing a natural cell to carry out the instructions encoded by the introduced synthetic genome. Synthetic biologists in this field view their work as basic study into the conditions necessary for life to exist and its origin more than in any of the other techniques. The protocell technique, however, also lends itself well to applications; similar to other synthetic biology byproducts, protocells could be employed for the manufacture of biopolymers and medicines.[38]
Unconventional molecular biology[edit]
The objective of the "unnatural molecular biology" strategy is to create new varieties of life that are based on a different kind of molecular biology, such as new types of nucleic acids or a new genetic code. The creation of new types of nucleotides that can be built into unique nucleic acids could be accomplished by changing certain DNA or RNA constituents, such as the bases or the backbone sugars.[38]
The normal genetic code is being altered by inserting quadruplet codons or changing some codons to encode new amino acids, which would subsequently permit the use of non-natural amino acids with unique features in protein production. It is a scientific and technological problem to adjust the enzymatic machinery of the cell for both approaches.[38]
A new sort of life would be formed by organisms with a genome built on synthetic nucleic acids or on a totally new coding system for synthetic amino acids. This new style of life would have some benefits but also some new dangers. On release into the environment, there would be no horizontal gene transfer or outcrossing of genes with natural species. Furthermore, these kinds of synthetic organisms might be created to require non-natural materials for protein or nucleic acid synthesis, rendering them unable to thrive in the wild if they accidentally escaped.[38]
On the other hand, if these organisms ultimately were able to survive outside of controlled space, they might have a particular benefit over natural organisms because they would be resistant to predatory living organisms or natural viruses, that could lead to an unmanaged spread of the synthetic organisms.[38]
In silico technique[edit]
Synthetic biology in silico and the various strategies are interconnected. The development of complex designs, whether they are metabolic pathways, fundamental cellular processes, or chassis genomes, is one of the major difficulties faced by the four synthetic-biology methods outlined above. Because of this, synthetic biology has a robust in silico branch, similar to systems biology, that aims to create computational models for the design of common biological components or synthetic circuits, which are essentially simulations of synthetic organisms.[38]
The practical application of simulations and models through bioengineering or other fields of synthetic biology is the long-term goal of in silico synthetic biology. Many of the computational simulations of synthetic organisms up to this point possess little to no direct analogy to living things. Due to this, in silico synthetic biology is regarded as a separate group in this article.[38]
It is sensible to integrate the five areas under the umbrella of synthetic biology as an unified area of study. Even though they focus on various facets of life, such as metabolic regulation, essential elements, or biochemical makeup, these five strategies all work toward the same end: creating new types of living organisms. Additionally, the varied methodologies begin with numerous methodological approaches, which leads to the diversity of synthetic biology approaches.[38]
Synthetic biology is an interdisciplinary field that draws from and is inspired by many different scientific disciplines, not one single field or technique. Synthetic biologists all have the same underlying objective of designing and producing new forms of life, despite the fact that they may employ various methodologies, techniques, and research instruments. Any evaluation of synthetic biology, whether it examines ethical, legal, or safety considerations, must take into account the fact that while some questions, risks, and issues are unique to each technique, in other circumstances, synthetic biology as a whole must be taken into consideration.[38]
Four engineering approaches[edit]
Synthetic biology has traditionally been divided into four different engineering approaches: top down, parallel, orthogonal and bottom up.[39]
To replicate emergent behaviours from natural biology and build artificial life, unnatural chemicals are used. The other looks for interchangeable components from biological systems to put together and create systems that do not work naturally. In either case, a synthetic objective compels researchers to venture into new area in order to engage and resolve issues that cannot be readily resolved by analysis. Due to this, new paradigms are driven to arise in ways that analysis cannot easily do. In addition to equipments that oscillate, creep, and play tic-tac-toe, synthetic biology has produced diagnostic instruments that enhance the treatment of patients with infectious diseases.[40]
Top-down approach[edit]
It involves using metabolic and genetic engineering techniques to impart new functions to living cells.[41] By comparing universal genes and eliminating non-essential ones to create a basic genome, this method seeks to lessen the complexity of existing cells. These initiatives are founded on the hypothesis of a single genesis for cellular life, the so-called Last Universal Common Ancestor, which supports the presence of a universal minimal genome that gave rise to all living things. Recent studies, however, raise the possibility that the eukaryotic and prokaryotic cells that make up the tree of life may have evolved from a group of primordial cells rather than from a single cell. As a result, even while the Holy Grail-like pursuit of the "minimum genome" has grown elusive, cutting out a number of non-essential functions impairs an organism's fitness and leads to "fragile" genomes.[39]
Bottom-up approach[edit]
This approach involves creating new biological systems in vitro by bringing together 'non-living' biomolecular components,[42] often with the aim of constructing an artificial cell.
Reproduction, replication, and assembly are three crucial self-organizational principles that are taken into account in order to accomplish this. Cells, which are made up of a container and a metabolism, are considered "hardware" in the definition of reproduction, whereas replication occurs when a system duplicates a perfect copy of itself, as in the case of DNA, which is considered "software." When vesicles or containers (such as Oparin's coacervates) formed of tiny droplets of molecules that are organic like lipids or liposomes, membrane-like structures comprising phospholipids, aggregate, assembly occur.[39]
The study of protocells exists along with other in vitro synthetic biology initiatives that seek to produce minimum cells, metabolic pathways, or "never-born proteins" as well as to mimic physiological functions including cell division and growth. The in vitro enhancement of synthetic pathways does have the potential to have an effect on some other synthetic biology sectors, including metabolic engineering, despite the fact that it no longer classified as synthetic biology research. This research, which is primarily essential, deserves proper recognition as synthetic biology research.[39]
Parallel approach[edit]
Parallel engineering is also known as bioengineering. The basic genetic code is the foundation for parallel engineering research, which uses conventional biomolecules like nucleic acids and the 20 amino acids to construct biological systems. For a variety of applications in biocomputing, bioenergy, biofuels, bioremediation, optogenetics, and medicine, it involves the standardisation of DNA components, engineering of switches, biosensors, genetic circuits, logic gates, and cellular communication operators. For directing the expression of two or more genes and/or proteins, the majority of these applications often rely on the use of one or more vectors (or plasmids). Small, circular, double-strand DNA units known as plasmids, which are primarily found in prokaryotic but can also occasionally be detected in eukaryotic cells, may replicate autonomously of chromosomal DNA.[39]
Orthogonal approach[edit]
It is also known as perpendicular engineering. This strategy, also referred to as "chemical synthetic biology," principally seeks to alter or enlarge the genetic codes of living systems utilising artificial DNA bases and/or amino acids. This subfield is also connected to xenobiology, a newly developed field that combines systems chemistry, synthetic biology, exobiology, and research into the origins of life. In recent decades, researchers have created compounds that are structurally similar to the DNA canonical bases to see if those "alien" or xeno (XNA) molecules may be employed as genetic information carriers. Similar to this, noncanonical moieties have taken the place of the DNA sugar (deoxyribose).[39] In order to express information other than the 20 conventional amino acids of proteins, the genetic code can be altered or enlarged. One method involves incorporating a specified unnatural, noncanonical, or xeno amino acid (XAA) into one or more proteins at one or more precise places using orthogonal enzymes and a transfer RNA adaptor from an other organism. By using "directed evolution," which entails repeated cycles of gene mutagenesis (genotypic diversity production), screening or selection (of a specific phenotypic trait), and amplification of a better variant for the following iterative round, orthogonal enzymes are produced Numerous XAAs have been effectively incorporated into proteins in more complex creatures like worms and flies as well as in bacteria, yeast, and human cell lines. As a result of canonical DNA sequence changes, directed evolution also enables the development of orthogonal ribosomes, which make it easier to incorporate XAAs into proteins or create "mirror life," or biological systems that contain biomolecules made up of enantiomers with different chiral orientations.[39]
Enabling technologies[edit]
Several novel enabling technologies were critical to the success of synthetic biology. Concepts include standardization of biological parts and hierarchical abstraction to permit using those parts in synthetic systems.[43] DNA serves as the guide for how biological processes should function, like the score to a complex symphony of life. Our ability to comprehend and design biological systems has undergone significant modifications as a result of developments in the previous few decades in both reading (sequencing) and writing (synthesis) DNA sequences. These developments have produced ground-breaking techniques for designing, assembling, and modifying DNA-encoded genes, materials, circuits, and metabolic pathways, enabling an ever-increasing amount of control over biological systems and even entire organisms.[44]
Basic technologies include reading and writing DNA (sequencing and fabrication). Measurements under multiple conditions are needed for accurate modeling and computer-aided design (CAD).
DNA and gene synthesis[edit]
Driven by dramatic decreases in costs of oligonucleotide ("oligos") synthesis and the advent of PCR, the sizes of DNA constructions from oligos have increased to the genomic level.[45] In 2000, researchers reported synthesis of the 9.6 kbp (kilo bp) Hepatitis C virus genome from chemically synthesized 60 to 80-mers.[46] In 2002, researchers at Stony Brook University succeeded in synthesizing the 7741 bp poliovirus genome from its published sequence, producing the second synthetic genome, spanning two years.[47] In 2003, the 5386 bp genome of the bacteriophage Phi X 174 was assembled in about two weeks.[48] In 2006, the same team, at the J. Craig Venter Institute, constructed and patented a synthetic genome of a novel minimal bacterium, Mycoplasma laboratorium and were working on getting it functioning in a living cell.[49][50][51]
In 2007, it was reported that several companies were offering synthesis of genetic sequences up to 2000 base pairs (bp) long, for a price of about $1 per bp and a turnaround time of less than two weeks.[52] Oligonucleotides harvested from a photolithographic- or inkjet-manufactured DNA chip combined with PCR and DNA mismatch error-correction allows inexpensive large-scale changes of codons in genetic systems to improve gene expression or incorporate novel amino-acids (see George M. Church's and Anthony Forster's synthetic cell projects.[53][54]). This favors a synthesis-from-scratch approach.
Additionally, the CRISPR/Cas system has emerged as a promising technique for gene editing. It was described as "the most important innovation in the synthetic biology space in nearly 30 years".[55] While other methods take months or years to edit gene sequences, CRISPR speeds that time up to weeks.[55] Due to its ease of use and accessibility, however, it has raised ethical concerns, especially surrounding its use in biohacking.[56][57][58]
Sequencing[edit]
DNA sequencing determines the order of nucleotide bases in a DNA molecule. Synthetic biologists use DNA sequencing in their work in several ways. First, large-scale genome sequencing efforts continue to provide information on naturally occurring organisms. This information provides a rich substrate from which synthetic biologists can construct parts and devices. Second, sequencing can verify that the fabricated system is as intended. Third, fast, cheap, and reliable sequencing can facilitate rapid detection and identification of synthetic systems and organisms.[59]
Modularity[edit]
This is the ability of a system or component to operate without reference to its context.[60]
The most used[61]: 22–23 standardized DNA parts are BioBrick plasmids, invented by Tom Knight in 2003.[14] Biobricks are stored at the Registry of Standard Biological Parts in Cambridge, Massachusetts. The BioBrick standard has been used by tens of thousands of students worldwide in the international Genetically Engineered Machine (iGEM) competition. BioBrick Assembly Standard 10 promotes modularity by allowing BioBrick coding sequences to be spliced out and exchanged using restriction enzymes EcoRI or XbaI (BioBrick prefix) and SpeI and PstI (BioBrick suffix).[61]: 22–23
Sequence overlap between two genetic elements (genes or coding sequences), called overlapping genes, can prevent their individual manipulation.[62] To increase genome modularity, the practice of genome refactoring or improving "the internal structure of an existing system for future use, while simultaneously maintaining external system function"[63] has been adopted across synthetic biology disciplines.[62] Some notable examples of refactoring including the nitrogen fixation cluster[64] and type III secretion system[65] along with bacteriophages T7[63] and ΦX174.[66]
While DNA is most important for information storage, a large fraction of the cell's activities are carried out by proteins. Tools can send proteins to specific regions of the cell and to link different proteins together. The interaction strength between protein partners should be tunable between a lifetime of seconds (desirable for dynamic signaling events) up to an irreversible interaction (desirable for device stability or resilient to harsh conditions). Interactions such as coiled coils,[67] SH3 domain-peptide binding[68] or SpyTag/SpyCatcher[69] offer such control. In addition, it is necessary to regulate protein-protein interactions in cells, such as with light (using light-oxygen-voltage-sensing domains) or cell-permeable small molecules by chemically induced dimerization.[70]
In a living cell, molecular motifs are embedded in a bigger network with upstream and downstream components. These components may alter the signaling capability of the modeling module. In the case of ultrasensitive modules, the sensitivity contribution of a module can differ from the sensitivity that the module sustains in isolation.[71][72]
Modeling[edit]
Models inform the design of engineered biological systems by better predicting system behavior prior to fabrication. Synthetic biology benefits from better models of how biological molecules bind substrates and catalyze reactions, how DNA encodes the information needed to specify the cell and how multi-component integrated systems behave. Multiscale models of gene regulatory networks focus on synthetic biology applications. Simulations can model all biomolecular interactions in transcription, translation, regulation and induction of gene regulatory networks.[73][74][75][76]
Only extensive modelling can enable the exploration of dynamic gene expression in a form suitable for research and design due to the numerous involved species and the intricacy of their relationships. Dynamic simulations of the entire biomolecular interconnection involved in regulation, transport, transcription, induction, and translation enable the molecular level detailing of designs. As opposed to modelling artificial networks a posteriori, this is contrasted.[77]
Microfluidics[edit]
Microfluidics, in particular droplet microfluidics, is an emerging tool used to construct new components, and to analyze and characterize them.[78][79] It is widely employed in screening assays.[80]
Synthetic transcription factors[edit]
Studies have considered the components of the DNA transcription mechanism. One desire of scientists creating synthetic biological circuits is to be able to control the transcription of synthetic DNA in unicellular organisms (prokaryotes) and in multicellular organisms (eukaryotes). One study tested the adjustability of synthetic transcription factors (sTFs) in areas of transcription output and cooperative ability among multiple transcription factor complexes.[81] Researchers were able to mutate functional regions called zinc fingers, the DNA specific component of sTFs, to decrease their affinity for specific operator DNA sequence sites, and thus decrease the associated site-specific activity of the sTF (usually transcriptional regulation). They further used the zinc fingers as components of complex-forming sTFs, which are the eukaryotic translation mechanisms.[81]
Applications[edit]
Synthetic biology initiatives frequently aim to redesign organisms so that they can create a material, such as a drug or fuel, or acquire a new function, such as the ability to sense something in the environment. Examples of what researchers are creating using synthetic biology include:
- Utilizing microorganisms for bioremediation to remove contaminants from our water, soil, and air.
- Production of complex natural products that are usually extracted from plants but cannot be obtained in sufficient amounts, e.g. drugs of natural origin, such as artemisinin and paclitaxel.
- Beta-carotene, a substance typically associated with carrots that prevents vitamin A deficiency, is produced by rice that has been modified. Every year, between 250,000 and 500,000 children lose their vision due to vitamin A deficiency, which also significantly raises their chance of dying from infectious infections.
- As a sustainable and environmentally benign alternative to the fresh roses that perfumers use to create expensive smells, yeast has been created to produce rose oil.[82]
Biosensors[edit]
A biosensor refers to an engineered organism, usually a bacterium, that is capable of reporting some ambient phenomenon such as the presence of heavy metals or toxins. One such system is the Lux operon of Aliivibrio fischeri,[83] which codes for the enzyme that is the source of bacterial bioluminescence, and can be placed after a respondent promoter to express the luminescence genes in response to a specific environmental stimulus.[84] One such sensor created, consisted of a bioluminescent bacterial coating on a photosensitive computer chip to detect certain petroleum pollutants. When the bacteria sense the pollutant, they luminesce.[85] Another example of a similar mechanism is the detection of landmines by an engineered E.coli reporter strain capable of detecting TNT and its main degradation product DNT, and consequently producing a green fluorescent protein (GFP).[86]
Modified organisms can sense environmental signals and send output signals that can be detected and serve diagnostic purposes. Microbe cohorts have been used.[87]
Biosensors could also be used to detect pathogenic signatures—such as of SARS-CoV-2—and can be wearable.[88][89]
For the purpose of detecting and reacting to various and temporary environmental factors, cells have developed a wide range of regulatory circuits, ranging from transcriptional to post-translational. These circuits are made up of transducer modules that filter the signals and activate a biological response, as well as carefully designed sensitive sections that attach analytes and regulate signal-detection thresholds. Modularity and selectivity are programmed to biosensor circuits at the transcriptional, translational, and post-translational levels, to achieve the delicate balancing of the two basic sensing modules.[90]
Food and drink[edit]
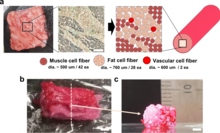
However, not all synthetic nutrition products are animal food products – for instance, as of 2021, there are also products of synthetic coffee that are reported to be close to commercialization.[98][99][100] Similar fields of research and production based on synthetic biology that can be used for the production of food and drink are:
- Genetically engineered microbial food cultures (e.g. for solar-energy-based protein powder)[101][102]
- Cell-free artificial synthesis (e.g. synthetic starch;[103][104] )
Materials[edit]
Photosynthetic microbial cells have been used as a step to synthetic production of spider silk.[105][106]
Biological computers[edit]
A biological computer refers to an engineered biological system that can perform computer-like operations, which is a dominant paradigm in synthetic biology. Researchers built and characterized a variety of logic gates in a number of organisms,[107] and demonstrated both analog and digital computation in living cells. They demonstrated that bacteria can be engineered to perform both analog and/or digital computation.[108][109] In 2007, in human cells, research demonstrated a universal logic evaluator that operates in mammalian cells.[110] Subsequently, researchers utilized this paradigm to demonstrate a proof-of-concept therapy that uses biological digital computation to detect and kill human cancer cells in 2011.[111] In 2016, another group of researchers demonstrated that principles of computer engineering can be used to automate digital circuit design in bacterial cells.[112] In 2017, researchers demonstrated the 'Boolean logic and arithmetic through DNA excision' (BLADE) system to engineer digital computation in human cells.[113] In 2019, researchers implemented a perceptron in biological systems opening the way for machine learning in these systems.[114]
Cell transformation[edit]
Cells use interacting genes and proteins, which are called gene circuits, to implement diverse function, such as responding to environmental signals, decision making and communication. Three key components are involved: DNA, RNA and Synthetic biologist designed gene circuits that can control gene expression from several levels including transcriptional, post-transcriptional and translational levels.
Traditional metabolic engineering has been bolstered by the introduction of combinations of foreign genes and optimization by directed evolution. This includes engineering E. coli and yeast for commercial production of a precursor of the antimalarial drug, Artemisinin.[115]
Entire organisms have yet to be created from scratch, although living cells can be transformed with new DNA. Several ways allow constructing synthetic DNA components and even entire synthetic genomes, but once the desired genetic code is obtained, it is integrated into a living cell that is expected to manifest the desired new capabilities or phenotypes while growing and thriving.[116] Cell transformation is used to create biological circuits, which can be manipulated to yield desired outputs.[12][13]
By integrating synthetic biology with materials science, it would be possible to use cells as microscopic molecular foundries to produce materials whose properties were genetically encoded. Re-engineering has produced Curli fibers, the amyloid component of extracellular material of biofilms, as a platform for programmable nanomaterial. These nanofibers were genetically constructed for specific functions, including adhesion to substrates, nanoparticle templating and protein immobilization.[117]
Designed proteins[edit]

Natural proteins can be engineered, for example, by directed evolution, novel protein structures that match or improve on the functionality of existing proteins can be produced. One group generated a helix bundle that was capable of binding oxygen with similar properties as hemoglobin, yet did not bind carbon monoxide.[119] A similar protein structure was generated to support a variety of oxidoreductase activities[120] while another formed a structurally and sequentially novel ATPase.[121] Another group generated a family of G-protein coupled receptors that could be activated by the inert small molecule clozapine N-oxide but insensitive to the native ligand, acetylcholine; these receptors are known as DREADDs.[122] Novel functionalities or protein specificity can also be engineered using computational approaches. One study was able to use two different computational methods: a bioinformatics and molecular modeling method to mine sequence databases, and a computational enzyme design method to reprogram enzyme specificity. Both methods resulted in designed enzymes with greater than 100 fold specificity for production of longer chain alcohols from sugar.[123]
Another common investigation is expansion of the natural set of 20 amino acids. Excluding stop codons, 61 codons have been identified, but only 20 amino acids are coded generally in all organisms. Certain codons are engineered to code for alternative amino acids including: nonstandard amino acids such as O-methyl tyrosine; or exogenous amino acids such as 4-fluorophenylalanine. Typically, these projects make use of re-coded nonsense suppressor tRNA-Aminoacyl tRNA synthetase pairs from other organisms, though in most cases substantial engineering is required.[124]
Other researchers investigated protein structure and function by reducing the normal set of 20 amino acids. Limited protein sequence libraries are made by generating proteins where groups of amino acids may be replaced by a single amino acid.[125] For instance, several non-polar amino acids within a protein can all be replaced with a single non-polar amino acid.[126] One project demonstrated that an engineered version of Chorismate mutase still had catalytic activity when only nine amino acids were used.[127]
Researchers and companies practice synthetic biology to synthesize industrial enzymes with high activity, optimal yields and effectiveness. These synthesized enzymes aim to improve products such as detergents and lactose-free dairy products, as well as make them more cost effective.[128] The improvements of metabolic engineering by synthetic biology is an example of a biotechnological technique utilized in industry to discover pharmaceuticals and fermentive chemicals. Synthetic biology may investigate modular pathway systems in biochemical production and increase yields of metabolic production. Artificial enzymatic activity and subsequent effects on metabolic reaction rates and yields may develop "efficient new strategies for improving cellular properties ... for industrially important biochemical production".[129]
Designed nucleic acid systems[edit]
Scientists can encode digital information onto a single strand of synthetic DNA. In 2012, George M. Church encoded one of his books about synthetic biology in DNA. The 5.3 Mb of data was more than 1000 times greater than the previous largest amount of information to be stored in synthesized DNA.[130] A similar project encoded the complete sonnets of William Shakespeare in DNA.[131] More generally, algorithms such as NUPACK,[132] ViennaRNA,[133] Ribosome Binding Site Calculator,[134] Cello,[135] and Non-Repetitive Parts Calculator[136] enables the design of new genetic systems.
Many technologies have been developed for incorporating unnatural nucleotides and amino acids into nucleic acids and proteins, both in vitro and in vivo. For example, in May 2014, researchers announced that they had successfully introduced two new artificial nucleotides into bacterial DNA. By including individual artificial nucleotides in the culture media, they were able to exchange the bacteria 24 times; they did not generate mRNA or proteins able to use the artificial nucleotides.[137][138][139]
Space exploration[edit]
Synthetic biology raised NASA's interest as it could help to produce resources for astronauts from a restricted portfolio of compounds sent from Earth.[140][141][142] On Mars, in particular, synthetic biology could lead to production processes based on local resources, making it a powerful tool in the development of occupied outposts with less dependence on Earth.[140] Work has gone into developing plant strains that are able to cope with the harsh Martian environment, using similar techniques to those employed to increase resilience to certain environmental factors in agricultural crops.[143]
Synthetic life[edit]
One important topic in synthetic biology is synthetic life, that is concerned with hypothetical organisms created in vitro from biomolecules and/or chemical analogues thereof. Synthetic life experiments attempt to either probe the origins of life, study some of the properties of life, or more ambitiously to recreate life from non-living (abiotic) components. Synthetic life biology attempts to create living organisms capable of carrying out important functions, from manufacturing pharmaceuticals to detoxifying polluted land and water.[145] In medicine, it offers prospects of using designer biological parts as a starting point for new classes of therapies and diagnostic tools.[145]
A living "artificial cell" has been defined as a completely synthetic cell that can capture energy, maintain ion gradients, contain macromolecules as well as store information and have the ability to mutate.[146] Nobody has been able to create such a cell.[146]
A completely synthetic bacterial chromosome was produced in 2010 by Craig Venter, and his team introduced it to genomically emptied bacterial host cells.[19] The host cells were able to grow and replicate.[147][148] The Mycoplasma laboratorium is the only living organism with completely engineered genome.
The first living organism with 'artificial' expanded DNA code was presented in 2014; the team used E. coli that had its genome extracted and replaced with a chromosome with an expanded genetic code. The nucleosides added are d5SICS and dNaM.[139]
In May 2019, in a milestone effort, researchers reported the creation of a new synthetic (possibly artificial) form of viable life, a variant of the bacteria Escherichia coli, by reducing the natural number of 64 codons in the bacterial genome to 59 codons instead, in order to encode 20 amino acids.[25][26]
In 2017, the international Build-a-Cell large-scale open-source research collaboration for the construction of synthetic living cells was started,[149] followed by national synthetic cell organizations in several countries, including FabriCell,[150] MaxSynBio[151] and BaSyC.[152] The European synthetic cell efforts were unified in 2019 as SynCellEU initiative.[153]
In 2023, researchers were able to create the first synthetically made human embryos derived from stem cells.[154]
Drug delivery platforms[edit]
In therapeutics, synthetic biology has achieved significant advancements in altering and simplifying the therapeutics scope in a relatively short period of time. In fact, new therapeutic platforms, from the discovery of disease mechanisms and drug targets to the manufacture and transport of small molecules, are made possible by the logical and model-guided design construction of biological components.[60]
Synthetic biology devices have been designed to act as therapies in therapeutic treatment. It is possible to control complete created viruses and organisms to target particular pathogens and diseased pathways. Thus, in two independent studies 91,92, researchers utilised genetically modified bacteriophages to fight antibiotic-resistant bacteria by giving them genetic features that specifically target and hinder bacterial defences against antibiotic activity.[155]
In the therapy of cancer, since conventional medicines frequently indiscriminately target tumours and normal tissues, artificially created viruses and organisms that can identify and connect their therapeutic action to pathological signals may be helpful. For example, p53 pathway activity in human cells was put into adenoviruses to control how they replicated.[155]
Engineered bacteria-based platform[edit]
Bacteria have long been used in cancer treatment. Bifidobacterium and Clostridium selectively colonize tumors and reduce their size.[156] Recently synthetic biologists reprogrammed bacteria to sense and respond to a particular cancer state. Most often bacteria are used to deliver a therapeutic molecule directly to the tumor to minimize off-target effects. To target the tumor cells, peptides that can specifically recognize a tumor were expressed on the surfaces of bacteria. Peptides used include an affibody molecule that specifically targets human epidermal growth factor receptor 2[157] and a synthetic adhesin.[158] The other way is to allow bacteria to sense the tumor microenvironment, for example hypoxia, by building an AND logic gate into bacteria.[159] Then the bacteria only release target therapeutic molecules to the tumor through either lysis[160] or the bacterial secretion system.[161] Lysis has the advantage that it can stimulate the immune system and control growth. Multiple types of secretion systems can be used and other strategies as well. The system is inducible by external signals. Inducers include chemicals, electromagnetic or light waves.
Multiple species and strains are applied in these therapeutics. Most commonly used bacteria are Salmonella typhimurium, Escherichia coli, Bifidobacteria, Streptococcus, Lactobacillus, Listeria and Bacillus subtilis. Each of these species have their own property and are unique to cancer therapy in terms of tissue colonization, interaction with immune system and ease of application.
Engineered yeast-based platform
Synthetic biologists are developing genetically modified live yeast that can deliver therapeutic biologic medicines. When orally delivered, these live yeast act like micro-factories and will make therapeutic molecules directly in the gastrointestinal tract. Because yeast are eukaryotic, a key benefit is that they can be administered together with antibiotics. Probiotic yeast expressing human P2Y2 purinergic receptor suppressed intestinal inflammation in mouse models of inflammatory bowel disease.[162] A live S. boulardii yeast delivering a tetra-specific anti-toxin that potently neutralizes Toxin A and Toxin B of Clostridioides difficile has been developed. This therapeutic anti-toxin is a fusion of four single-domain antibodies (nanobodies) that potently and broadly neutralize the two major virulence factors of C. difficile at the site of infection in preclinical models.[163] The first in human clinical trial of engineered live yeast for the treatment of Clostridium difficile infection is anticipated in 2024 and will be sponsored by the developer Fzata, Inc.
Cell-based platform[edit]
The immune system plays an important role in cancer and can be harnessed to attack cancer cells. Cell-based therapies focus on immunotherapies, mostly by engineering T cells.
T cell receptors were engineered and 'trained' to detect cancer epitopes. Chimeric antigen receptors (CARs) are composed of a fragment of an antibody fused to intracellular T cell signaling domains that can activate and trigger proliferation of the cell. Multiple second generation CAR-based therapies have been approved by FDA.[164]
Gene switches were designed to enhance safety of the treatment. Kill switches were developed to terminate the therapy should the patient show severe side effects.[165] Mechanisms can more finely control the system and stop and reactivate it.[166][167] Since the number of T-cells are important for therapy persistence and severity, growth of T-cells is also controlled to dial the effectiveness and safety of therapeutics.[168]
Although several mechanisms can improve safety and control, limitations include the difficulty of inducing large DNA circuits into the cells and risks associated with introducing foreign components, especially proteins, into cells.
Biofuels, pharmaceuticals and biomaterials[edit]
The most popular biofuel is ethanol produced from corn or sugar cane, but this method of producing biofuels is troublesome and constrained due to the high agricultural cost and inadequate fuel characteristics of ethanol. An substitute and potential source of renewable energy is microbes that have had their metabolic pathways altered to be more efficient at converting biomass into biofuels. Only if their production costs could be made to match or even exceed those of present fuel production can these techniques be expected to be successful. Related to this, there are several medicines whose pricey manufacturing procedures prevent them from having a larger therapeutic range. The creation of new materials and the microbiological manufacturing of biomaterials would both benefit substantially from novel artificial biology tools.[155]
CRISPR/Cas9[edit]
The clustered frequently interspaced short palindromic repetitions (CRISPR)/CRISPR associated (Cas) system is a powerful method of genome engineering in a range of organisms because of its simplicity, modularity, and scalability. In this technique, a guide RNA (gRNA) attracts the CRISPR nuclease Cas9 to a particular spot in the genome, causing a double strand break. Several DNA repair processes, including homology-directed recombination and non-homology end joining, can be used to accomplish the desired genome change (i.e., gene deletion or insertion). Additionally, dCas9 (dead Cas9 or nuclease-deficient Cas9), a Cas9 double mutant (H840A, D10A), has been utilised to control gene expression in bacteria or when linked to a stimulation of suppression site in yeast.[169]
Regulatory elements[edit]
To build and develop biological systems, regulating components including regulators, ribosome-binding sites (RBSs), and terminators are crucial. Despite years of study, there are many various varieties and numbers of promoters and terminators for Escherichia coli, but also for the well-researched model organism Saccharomyces cerevisiae, as well as for other organisms of interest, these tools are quite scarce. Numerous techniques have been invented for the finding and identification of promoters and terminators in order to overcome this constraint, including genome mining, random mutagenesis, hybrid engineering, biophysical modelling, combinatorial design, and rational design.[169]
Organoids[edit]
Synthetic biology has been used for organoids, which are lab-grown organs with application to medical research and transplantation.[170]
Bioprinted organs[edit]
3D bioprinting can be used to reconstruct tissue from various regions of the body. The precursor to the adoption of 3D printing in healthcare was a series of trials conducted by researchers at Boston Children's Hospital. The team built replacement urinary bladders by hand for seven patients by constructing scaffolds, then layering the scaffolds with cells from the patients and allowing them to grow. The trials were a success as the patients remained in good health 7 years after implantation, which led a research fellow named Anthony Atala, MD, to search or ways to automate the process.[171] Patients with end-stage bladder disease can now be treated by using bio-engineered bladder tissues to rebuild the damaged organ.[172] This technology can also potentially be applied to bone, skin, cartilage and muscle tissue.[173] Though one long-term goal of 3D bioprinting technology is to reconstruct an entire organ as well as minimize the problem of the lack of organs for transplantation.[174] There has been little success in bioprinting of fully functional organs e.g. liver, skin, meniscus or pancreas.[175][176][177] Unlike implantable stents, organs have complex shapes and are significantly harder to bioprint. A bioprinted heart, for example, must not only meet structural requirements, but also vascularization, mechanical load, and electrical signal propagation requirements.[178] In 2022, the first success of a clinical trial for a 3D bioprinted transplant that is made from the patient's own cells, an external ear to treat microtia,[179] was reported.[180]
3D bioprinting contributes to significant advances in the medical field of tissue engineering by allowing for research to be done on innovative materials called biomaterials. Some of the most notable bioengineered substances are usually stronger than the average bodily materials, including soft tissue and bone. These constituents can act as future substitutes, even improvements, for the original body materials. In addition, the Defense Threat Reduction Agency aims to print mini organs such as hearts, livers, and lungs as the potential to test new drugs more accurately and perhaps eliminate the need for testing in animals.[181]
Other transplants and induced regeneration[edit]
There is ongoing research and development into synthetic biology based methods for inducing regeneration in humans[relevant?] as well the creation of transplantable artificial organs.
Nanoparticles, artificial cells and micro-droplets[edit]
Synthetic biology can be used for creating nanoparticles which can be used for drug-delivery as well as for other purposes.[182] Complementing research and development seeks to and has created synthetic cells that mimics functions of biological cells. Applications include medicine such as designer-nanoparticles that make blood cells eat away—from the inside out—portions of atherosclerotic plaque that cause heart attacks.[183][184][185] Synthetic micro-droplets for algal cells or synergistic algal-bacterial multicellular spheroid microbial reactors, for example, could be used to produce hydrogen as hydrogen economy biotechnology.[186][187]
Electrogenetics[edit]
Mammalian designer cells are engineered by humans to behave a specific way, such as an immune cell that expresses a synthetic receptor designed to combat a specific disease.[188][189] Electrogenetics is an application of synthetic biology that involves utilizing electrical fields to stimulate a response in engineered cells.[190] Controlling the designer cells can be done with relative ease through the use of common electronic devices, such as smartphones. Additionally, electrogenetics allows for the possibility of creating devices that are much smaller and compact than devices that use other stimulus through the use of microscopic electrodes.[190] One example of how electrogenetics is used to benefit public health is through stimulating designer cells that are able to produce/deliver therapeutics.[191] This was implemented in ElectroHEK cells, cells that contain voltage-gated calcium channels that are electrosensitive, meaning that the ion channel can be controlled by electrical conduction between electrodes and the ElectroHEK cells.[190] The expression levels of the artificial gene that these ElectroHEK cells contained was shown to be able to be controlled by changing the voltage or electrical pulse length. Further studies have expanded on this robust system, one of which is a beta cell line system designed to control the release of insulin based on electric signals.[192]
Ethics[edit]
This section needs to be updated. (January 2019) |
The creation of new life and the tampering of existing life has raised ethical concerns in the field of synthetic biology and are actively being discussed.[193][194]
Common ethical questions include:
- Is it morally right to tamper with nature?
- Is one playing God when creating new life?
- What happens if a synthetic organism accidentally escapes?
- What if an individual misuses synthetic biology and creates a harmful entity (e.g., a biological weapon)?
- Who will have control of and access to the products of synthetic biology?
- Who will gain from these innovations? Investors? Medical patients? Industrial farmers?
- Does the patent system allow patents on living organisms? What about parts of organisms, like HIV resistance genes in humans?[195]
- What if a new creation is deserving of moral or legal status?
The ethical aspects of synthetic biology has three main features: biosafety, biosecurity, and the creation of new life forms.[196] Other ethical issues mentioned include the regulation of new creations, patent management of new creations, benefit distribution, and research integrity.[197][193]
Ethical issues have surfaced for recombinant DNA and genetically modified organism (GMO) technologies and extensive regulations of genetic engineering and pathogen research were in place in many jurisdictions. Amy Gutmann, former head of the Presidential Bioethics Commission, argued that we should avoid the temptation to over-regulate synthetic biology in general, and genetic engineering in particular. According to Gutmann, "Regulatory parsimony is especially important in emerging technologies...where the temptation to stifle innovation on the basis of uncertainty and fear of the unknown is particularly great. The blunt instruments of statutory and regulatory restraint may not only inhibit the distribution of new benefits, but can be counterproductive to security and safety by preventing researchers from developing effective safeguards.".[198]
The "creation" of life[edit]
One ethical question is whether or not it is acceptable to create new life forms, sometimes known as "playing God". Currently, the creation of new life forms not present in nature is at small-scale, the potential benefits and dangers remain unknown, and careful consideration and oversight are ensured for most studies.[193] Many advocates express the great potential value—to agriculture, medicine, and academic knowledge, among other fields—of creating artificial life forms. Creation of new entities could expand scientific knowledge well beyond what is currently known from studying natural phenomena. Yet there is concern that artificial life forms may reduce nature's "purity" (i.e., nature could be somehow corrupted by human intervention and manipulation) and potentially influence the adoption of more engineering-like principles instead of biodiversity- and nature-focused ideals. Some are also concerned that if an artificial life form were to be released into nature, it could hamper biodiversity by beating out natural species for resources (similar to how algal blooms kill marine species). Another concern involves the ethical treatment of newly created entities if they happen to sense pain, sentience, and self-perception. There is an ongoing debate as to whether such life forms should be granted moral or legal rights, though no consensus exists as to how these rights would be administered or enforced.
Ethical support for synthetic biology[edit]
Ethics and moral rationales that support certain applications of synthetic biology include their potential mitigation of substantial global problems of detrimental environmental impacts of conventional agriculture (including meat production), animal welfare, food security, and human health,[199][200][201][202] as well as potential reduction of human labor needs and, via therapies of diseases, reduction of human suffering and prolonged life.
Biosafety and biocontainment[edit]
What is most ethically appropriate when considering biosafety measures? How can accidental introduction of synthetic life in the natural environment be avoided? Much ethical consideration and critical thought has been given to these questions. Biosafety not only refers to biological containment; it also refers to strides taken to protect the public from potentially hazardous biological agents. Even though such concerns are important and remain unanswered, not all products of synthetic biology present concern for biological safety or negative consequences for the environment. It is argued that most synthetic technologies are benign and are incapable of flourishing in the outside world due to their "unnatural" characteristics as there is yet to be an example of a transgenic microbe conferred with a fitness advantage in the wild.
In general, existing hazard controls, risk assessment methodologies, and regulations developed for traditional genetically modified organisms (GMOs) are considered to be sufficient for synthetic organisms. "Extrinsic" biocontainment methods in a laboratory context include physical containment through biosafety cabinets and gloveboxes, as well as personal protective equipment. In an agricultural context, they include isolation distances and pollen barriers, similar to methods for biocontainment of GMOs. Synthetic organisms may offer increased hazard control because they can be engineered with "intrinsic" biocontainment methods that limit their growth in an uncontained environment, or prevent horizontal gene transfer to natural organisms. Examples of intrinsic biocontainment include auxotrophy, biological kill switches, inability of the organism to replicate or to pass modified or synthetic genes to offspring, and the use of xenobiological organisms using alternative biochemistry, for example using artificial xeno nucleic acids (XNA) instead of DNA.[203][204] Regarding auxotrophy, bacteria and yeast can be engineered to be unable to produce histidine, an important amino acid for all life. Such organisms can thus only be grown on histidine-rich media in laboratory conditions, nullifying fears that they could spread into undesirable areas.
Biosecurity and bioterrorism[edit]
Some ethical issues relate to biosecurity, where biosynthetic technologies could be deliberately used to cause harm to society and/or the environment. Since synthetic biology raises ethical issues and biosecurity issues, humanity must consider and plan on how to deal with potentially harmful creations, and what kinds of ethical measures could possibly be employed to deter nefarious biosynthetic technologies. With the exception of regulating synthetic biology and biotechnology companies,[205][206] however, the issues are not seen as new because they were raised during the earlier recombinant DNA and genetically modified organism (GMO) debates, and extensive regulations of genetic engineering and pathogen research are already in place in many jurisdictions.[207]
Additionally, the development of synthetic biology tools has made it easier for individuals with less education, training, and access to equipment to modify and use pathogenic organisms as bioweapons. This increases the threat of bioterrorism, especially as terrorist groups become aware of the significant social, economic, and political disruption caused by pandemics like COVID-19. As new techniques are developed in the field of synthetic biology, the risk of bioterrorism is likely to continue to grow.[208] Juan Zarate, who served as Deputy National Security Advisor for Combating Terrorism from 2005 to 2009, noted that "the severity and extreme disruption of a novel coronavirus will likely spur the imagination of the most creative and dangerous groups and individuals to reconsider bioterrorist attacks."[209]
European Union[edit]
The European Union-funded project SYNBIOSAFE[210] has issued reports on how to manage synthetic biology. A 2007 paper identified key issues in safety, security, ethics, and the science-society interface, which the project defined as public education and ongoing dialogue among scientists, businesses, government and ethicists.[211][212] The key security issues that SYNBIOSAFE identified involved engaging companies that sell synthetic DNA and the biohacking community of amateur biologists. Key ethical issues concerned the creation of new life forms.
A subsequent report focused on biosecurity, especially the so-called dual-use challenge. For example, while synthetic biology may lead to more efficient production of medical treatments, it may also lead to synthesis or modification of harmful pathogens (e.g., smallpox).[213] The biohacking community remains a source of special concern, as the distributed and diffuse nature of open-source biotechnology makes it difficult to track, regulate or mitigate potential concerns over biosafety and biosecurity.[214]
COSY, another European initiative, focuses on public perception and communication.[215][216][217] To better communicate synthetic biology and its societal ramifications to a broader public, COSY and SYNBIOSAFE published SYNBIOSAFE, a 38-minute documentary film, in October 2009.[210]
The International Association Synthetic Biology has proposed self-regulation.[218] This proposes specific measures that the synthetic biology industry, especially DNA synthesis companies, should implement. In 2007, a group led by scientists from leading DNA-synthesis companies published a "practical plan for developing an effective oversight framework for the DNA-synthesis industry".[205]
United States[edit]
In January 2009, the Alfred P. Sloan Foundation funded the Woodrow Wilson Center, the Hastings Center, and the J. Craig Venter Institute to examine the public perception, ethics and policy implications of synthetic biology.[219]
9–10 июля 2009 г. Комитет национальных академий науки, технологий и права созвал симпозиум на тему «Возможности и проблемы в новой области синтетической биологии». [220]
После публикации первого синтетического генома и сопутствующего освещения в СМИ создаваемой «жизни» президент Барак Обама учредил Президентскую комиссию по изучению биоэтических проблем для изучения синтетической биологии. [221] Комиссия созвала серию встреч и в декабре 2010 года опубликовала отчет под названием «Новые направления: этика синтетической биологии и новые технологии». Комиссия заявила, что «хотя достижения Вентера ознаменовали собой значительный технический прогресс в демонстрации того, что относительно большой геном может быть точно синтезирован и заменен другим, это не равносильно «созданию жизни». [222] Он отметил, что синтетическая биология является новой областью, которая создает потенциальные риски и выгоды. Комиссия не рекомендовала изменения в политике или надзоре и призвала к продолжению финансирования исследований и новому финансированию мониторинга, изучения возникающих этических проблем и просвещения общественности. [207]
Синтетическая биология, как главный инструмент биологических достижений, приводит к «потенциалу разработки биологического оружия, возможным непредвиденным негативным последствиям для здоровья человека... и любому потенциальному воздействию на окружающую среду». [223] Распространение таких технологий может также сделать производство биологического и химического оружия доступным для более широкого круга государственных и негосударственных субъектов . [224] Этих проблем безопасности можно избежать, регулируя использование биотехнологий в промышленности посредством политического законодательства. Федеральные руководящие принципы по генетическим манипуляциям предлагает «Президентская комиссия по биоэтике... в ответ на объявленное создание самовоспроизводящейся клетки из химически синтезированного генома выдвинула 18 рекомендаций не только для регулирования науки... для образования общественность». [223]
Оппозиция [ править ]
13 марта 2012 года более 100 экологических групп и групп гражданского общества, включая «Друзей Земли» , Международный центр оценки технологий и Группу ETC , выпустили манифест «Принципы надзора за синтетической биологией» . Этот манифест призывает к введению во всем мире моратория на выпуск и коммерческое использование синтетических организмов до тех пор, пока не будут установлены более строгие правила и строгие меры биобезопасности. Группы конкретно призывают к полному запрету на использование синтетической биологии в отношении человеческого генома или человеческого микробиома . [225] [226] Ричард Левонтин писал, что некоторые из принципов безопасности надзора, обсуждаемых в «Принципах надзора за синтетической биологией», разумны, но основная проблема с рекомендациями манифеста заключается в том, что «общественность в целом не имеет возможности добиться какой-либо значимой реализации». этих рекомендаций». [227]
Здоровье и безопасность [ править ]
Опасности синтетической биологии включают угрозы биобезопасности для работников и населения, опасности биобезопасности , возникающие в результате преднамеренной разработки организмов с целью причинения вреда, а также опасности для окружающей среды. Опасности для биобезопасности аналогичны опасностям, возникающим в существующих областях биотехнологии, в основном это воздействие патогенов и токсичных химикатов, хотя новые синтетические организмы могут нести новые риски. [203] Что касается биобезопасности, существуют опасения, что синтетические или модифицированные организмы теоретически могут быть использованы для биотерроризма . Потенциальные риски включают воссоздание известных патогенов с нуля, создание более опасных существующих патогенов и создание микробов для производства вредных биохимических веществ. [228] Наконец, экологические опасности включают неблагоприятное воздействие на биоразнообразие и экосистемные услуги , включая потенциальные изменения в землепользовании в результате использования синтетических организмов в сельском хозяйстве. [229] [230] Синтетическая биология является примером технологии двойного назначения , которую можно использовать таким образом, чтобы намеренно или непреднамеренно нанести вред людям и/или окружающей среде. Часто «ученые, принимающие их учреждения и финансирующие организации» рассматривают вопрос о том, может ли запланированное исследование быть использовано не по назначению, и иногда принимают меры для снижения вероятности неправомерного использования. [231]
Существующие системы анализа рисков для ГМО обычно считаются достаточными для синтетических организмов, хотя могут возникнуть трудности для организма, построенного «снизу вверх» из отдельных генетических последовательностей. [204] [232] Синтетическая биология обычно подпадает под действие существующих правил в отношении ГМО и биотехнологии в целом, а также любых правил, которые существуют для последующих коммерческих продуктов, хотя, как правило, ни в одной юрисдикции нет правил, специфичных для синтетической биологии. [233] [234]
См. также [ править ]
- ACS Synthetic Biology (журнал)
- Биоинженерия
- Биомимикрия
- Кривая Карлсона
- Концепция хиральной жизни
- Вычислительная биология
- Компьютерное биомоделирование
- Хранение цифровых данных ДНК
- Инженерная биология
- Международная генно-инженерная машина
- Неклеточная жизнь
- Открытая синтетическая биология
- Регенеративная медицина
- Синтетический интеллект
- Синтетическая морфология
- Синтетическая вирусология
- Системы и синтетическая биология (журнал)
- Тканевая инженерия
- Ксенобиология
- Протоклетка # Искусственные модели
- Дживану
- Гипотетические типы биохимии
- Игра в Бога (этика)
Ссылки [ править ]
- ^ Jump up to: Перейти обратно: а б Ганчиц М.М. (май 2020 г.). «Инженерная жизнь: обзор синтетической биологии». Искусственная жизнь . 26 (2): 260–273. дои : 10.1162/artl_a_00318 . hdl : 11572/302757 . ISSN 1064-5462 . ПМИД 32271630 . S2CID 215550945 .
- ^ Накано Т., Экфорд А.В., Харагути Т. (12 сентября 2013 г.). Молекулярная связь . Издательство Кембриджского университета. ISBN 978-1-107-02308-6 .
- ^ Jump up to: Перейти обратно: а б «Синтетическая биология» . Genome.gov . Проверено 3 января 2023 г.
- ^ «Продуктивные наносистемы: технологическая дорожная карта» (PDF) . Институт Форсайта . Архивировано из оригинала (PDF) 25 октября 2016 г. Проверено 2 мая 2020 г.
- ^ Герцог С. (26 октября 1910 г.). Физико-химическая теория жизни и самопроизвольного зарождения . А. Пуанат. OL 23348076M – через Открытую библиотеку.
- ^ Ледюк С. (1912). Пуйнат А (ред.). Синтетическая биология, изучение биофизики . Архивировано из оригинала 27 сентября 2016 г. Проверено 23 января 2011 г.
- ^ Кэри, Несса (2019). Взлом кода жизни . Лондон: Книги об иконах. стр. Страница=12. ISBN 978-178578-497-2 .
- ^ Джейкоб, F.ß. И Моно, Дж. О регуляции активности генов. Холодный источник Харб. Симп. Квант. Биол. 26, 193–211 (1961).
- ^ Коэн С.Н., Чанг А.С., Бойер Х.В., Хеллинг Р.Б. (1973). «Конструирование биологически функциональных бактериальных плазмид in vitro» . Учеб. Натл. акад. наук. США . 70 (11): 3240–3244. Бибкод : 1973PNAS...70.3240C . дои : 10.1073/pnas.70.11.3240 . ПМК 427208 . ПМИД 4594039 .
- ^ Шибальский В., Скалка А. (ноябрь 1978 г.). «Нобелевские премии и ферменты рестрикции». Джин . 4 (3): 181–2. дои : 10.1016/0378-1119(78)90016-1 . ПМИД 744485 .
- ^ Сайки Р.К., Гельфанд Д.Х., Стоффель С., Шарф С.Дж., Хигучи Р., Хорн Г.Т., Муллис К.Б., Эрлих Х.А. (1988). «Праймер-направленная ферментативная амплификация ДНК с помощью термостабильной ДНК-полимеразы». Наука . 239 (4839): 487–491. дои : 10.1126/science.239.4839.487 . ПМИД 2448875 .
- ^ Jump up to: Перейти обратно: а б Еловиц М.Б., Лейблер С. (январь 2000 г.). «Синтетическая колебательная сеть регуляторов транскрипции». Природа . 403 (6767): 335–8. Бибкод : 2000Natur.403..335E . дои : 10.1038/35002125 . ПМИД 10659856 . S2CID 41632754 .
- ^ Jump up to: Перейти обратно: а б Гарднер Т.С., Кантор Ч.Р., Коллинз Дж.Дж. (январь 2000 г.). «Создание генетического тумблера в Escherichia coli». Природа . 403 (6767): 339–42. Бибкод : 2000Natur.403..339G . дои : 10.1038/35002131 . ПМИД 10659857 . S2CID 345059 .
- ^ Jump up to: Перейти обратно: а б Найт Т. (2003). Идемпотентный векторный дизайн для стандартной сборки биокирпичей (отчет). Лаборатория искусственного интеллекта Массачусетского технологического института. hdl : 1721.1/21168 .
- ^ Мартин В.Дж., Питера Дж., Уизерс С.Т., Ньюман Дж.Д., Кислинг Дж.Д. (июль 2003 г.). «Разработка мевалонатного пути в Escherichia coli для производства терпеноидов». Природная биотехнология . 21 (7): 796–802. дои : 10.1038/nbt833 . ПМИД 12778056 . S2CID 17214504 .
- ^ Левская А., Шевалье А.А., Табор Дж.Дж., Симпсон З.Б., Лавери Л.А., Леви М. и др. (ноябрь 2005 г.). «Синтетическая биология: инженерия Escherichia coli, чтобы увидеть свет». Природа . 438 (7067): 441–442. Бибкод : 2005Natur.438..441L . дои : 10.1038/nature04405 . ПМИД 16306980 . S2CID 4428475 .
- ^ Басу С., Герчман Ю., Коллинз Ч.Х., Арнольд Ф.Х., Вайс Р. (апрель 2005 г.). «Синтетическая многоклеточная система для формирования запрограммированных образов» . Природа . 434 (7037): 1130–4. Бибкод : 2005Natur.434.1130B . дои : 10.1038/nature03461 . ПМИД 15858574 . S2CID 4370309 .
- ^ Андерсон Дж.К., Кларк Э.Дж., Аркин А.П., Фойгт Калифорния (январь 2006 г.). «Контролируемое окружающей средой вторжение в раковые клетки искусственно созданными бактериями». Журнал молекулярной биологии . 355 (4): 619–627. дои : 10.1016/j.jmb.2005.10.076 . ПМИД 16330045 .
- ^ Jump up to: Перейти обратно: а б Гибсон Д.Г., Гласс Дж.И., Лартиг С., Носков В.Н., Чуанг Р.Ю., Алжир М.А., Бендерс Г.А., Монтегю М.Г., Ма Л., Муди М.М., Мерриман С., Ваши С., Кришнакумар Р., Асад-Гарсия Н., Эндрюс-Пфанкох С., Денисова Э.А., Янг Л., Ци ЗК, Сегалл-Шапиро Т.Х., Калви Ч., Пармар П.П., Хатчисон К.А., Смит Х.О., Вентер Дж.К. (июль 2010 г.). «Создание бактериальной клетки, управляемой химически синтезированным геномом» . Наука . 329 (5987): 52–6. Бибкод : 2010Sci...329...52G . дои : 10.1126/science.1190719 . ПМИД 20488990 .
- ^ «Американский учёный, создавший искусственную жизнь, отрицает «игру в Бога» » . Телеграф . Май 2010 г. Архивировано из оригинала 12 января 2022 г.
- ^ Даймонд Дж.С., Ричардсон С.М., Кумбс С.Э., Бабац Т., Мюллер Х., Анналуру Н. и др. (сентябрь 2011 г.). «Синтетические плечи хромосом функционируют у дрожжей и задумано создают фенотипическое разнообразие» . Природа . 477 (7365): 471–476. Бибкод : 2011Natur.477..471D . дои : 10.1038/nature10403 . ПМЦ 3774833 . ПМИД 21918511 .
- ^ Джинек М., Чилински К., Фонфара И., Хауэр М., Дудна Дж.А., Шарпантье Э. (2012). «Программируемая ДНК-эндонуклеаза, управляемая двойной РНК, в адаптивном бактериальном иммунитете» . Наука . 337 (6096): 816–821. Бибкод : 2012Sci...337..816J . дои : 10.1126/science.1225829 . ПМК 6286148 . ПМИД 22745249 .
- ^ ETH Цюрих (1 апреля 2019 г.). «Первый бактериальный геном, полностью созданный с помощью компьютера» . ЭврекАлерт! . Проверено 2 апреля 2019 г.
- ^ Венец Дж.Э., Дель Медико Л., Вольфле А., Шехле П., Бухер Ю., Апперт Д. и др. (апрель 2019 г.). «Химический синтез: переписывание бактериального генома для достижения гибкости дизайна и биологической функциональности» . Труды Национальной академии наук Соединенных Штатов Америки . 116 (16): 8070–8079. Бибкод : 2019PNAS..116.8070V . дои : 10.1073/pnas.1818259116 . ПМК 6475421 . ПМИД 30936302 .
- ^ Jump up to: Перейти обратно: а б Циммер С (15 мая 2019 г.). «Ученые создали бактерии с синтетическим геномом. Это искусственная жизнь? Это важная веха для синтетической биологии: колонии кишечной палочки процветают благодаря ДНК, созданной с нуля людьми, а не природой» . Нью-Йорк Таймс . Проверено 16 мая 2019 г.
- ^ Jump up to: Перейти обратно: а б Фреденс Дж., Ван К., де ла Торре Д., Функе Л.Ф., Робертсон В.Е., Христова Ю. и др. (май 2019 г.). «Тотальный синтез Escherichia coli с перекодированным геномом» . Природа . 569 (7757): 514–518. Бибкод : 2019Natur.569..514F . дои : 10.1038/s41586-019-1192-5 . ПМК 7039709 . ПМИД 31092918 .
- ^ Сокол Ю (03.04.2020). «Знакомьтесь, ксеноботы: ожившие виртуальные существа» . Нью-Йорк Таймс .
- ^ Кригман С., Блэкистон Д., Левин М., Бонгард Дж. (декабрь 2021 г.). «Кинематическая самовоспроизведение в реконфигурируемых организмах» . Труды Национальной академии наук Соединенных Штатов Америки . 118 (49). Бибкод : 2021PNAS..11812672K . дои : 10.1073/pnas.2112672118 . ПМЦ 8670470 . ПМИД 34845026 .
- ^ Цзэн Дж. «О концепции системной биоинженерии». Сотрудничество по трансгенным животным, июнь 1994 г., CAS, КНР . 6 .
- ^ Чопра П., Камма А. «Инженерная жизнь посредством синтетической биологии». В кремниевой биологии . 6 .
- ^ Флорес Буэсо Ю., Тэнгни М. (май 2017 г.). «Синтетическая биология у руля биоэкономики». Тенденции в биотехнологии . 35 (5): 373–378. дои : 10.1016/j.tibtech.2017.02.002 . ПМИД 28249675 .
- ^ Хантер Д. (октябрь 2013 г.). «Как на основе справедливости возражать против радикально новых технологий: случай синтетической биологии». Биоэтика . 27 (8): 426–434. дои : 10.1111/bioe.12049 . ПМИД 24010854 . S2CID 23895514 .
- ^ Гутманн А (2011). «Этика синтетической биологии: руководящие принципы для новых технологий». Отчет Гастингсского центра . 41 (4): 17–22. дои : 10.1002/j.1552-146x.2011.tb00118.x . ПМИД 21845917 . S2CID 20662786 .
- ^ Ченнон К., Бромли Э.Х., Вульфсон Д.Н. (август 2008 г.). «Синтетическая биология посредством биомолекулярного дизайна и инженерии». Современное мнение в области структурной биологии . 18 (4): 491–498. дои : 10.1016/j.sbi.2008.06.006 . ПМИД 18644449 .
- ^ Дюран-Небреда С., Пла Х., Видиелла Б., Пиньеро Х., Конде-Пуэйо Н., Соле Р. (февраль 2021 г.). «Синтетическое латеральное ингибирование в периодическом паттерне формирования микробных колоний» . ACS Синтетическая биология . 10 (2): 277–285. doi : 10.1021/acsynbio.0c00318 . ПМЦ 8486170 . ПМИД 33449631 . S2CID 231624252 .
- ^ ван Вармердам Т. (25 декабря 2021 г.). «Изучение биотехнологических достижений в экзогенном биологическом производстве промышленных малых молекул и фармацевтических препаратов» . ВашBioHelper.com .
- ^ Стоун М (2006). «Жизнь, переработанная с учетом требований инженеров» (PDF) . Микроб . 1 (12): 566–570. S2CID 7171812 . Архивировано из оригинала (PDF) 12 июня 2018 г.
- ^ Jump up to: Перейти обратно: а б с д и ж г час я дж к л м н тот п д р Деплазес, Анна (май 2009 г.). «Собираем головоломку: изложение синтетической биологии» . Отчеты ЭМБО . 10 (5): 428–432. дои : 10.1038/embor.2009.76 . ISSN 1469-221X . ПМК 2680885 . ПМИД 19415076 .
- ^ Jump up to: Перейти обратно: а б с д и ж г Асеведо-Роча, Карлос Г. (2016), Хаген, Кристин; Энгельхард, Маргрет; Топфер, Георг (ред.), «Синтетическая природа биологии» , Амбивалентность создания жизни: социальные и философские аспекты синтетической биологии , Этика оценки науки и технологий, том. 45, Чам: Springer International Publishing, стр. 9–53, номер номера : 10.1007/978-3-319-21088-9_2 , ISBN. 978-3-319-21088-9 , PMC 7123144 , получено 23 января 2023 г.
- ^ Беннер С.А., Сисмур А.М. (июль 2005 г.). «Синтетическая биология» . Обзоры природы. Генетика . 6 (7): 533–543. дои : 10.1038/nrg1637 . ПМК 7097405 . ПМИД 15995697 .
- ^ Швилле, Петра (2 сентября 2011 г.). «Синтетическая биология снизу вверх: инженерия в мире ремесленников» . Наука . 333 (6047): 1252–1254. Бибкод : 2011Sci...333.1252S . дои : 10.1126/science.1211701 . ISSN 0036-8075 . ПМИД 21885774 . S2CID 43354332 .
- ^ Швилле П. (сентябрь 2011 г.). «Синтетическая биология снизу вверх: инженерия в мире мастеров». Наука . 333 (6047): 1252–4. Бибкод : 2011Sci...333.1252S . дои : 10.1126/science.1211701 . ПМИД 21885774 . S2CID 43354332 .
- ^ Бейкер Д., Черч Дж., Коллинз Дж., Энди Д., Джейкобсон Дж., Кислинг Дж. и др. (июнь 2006 г.). «Инженерная жизнь: строительство фабрики по биологии». Научный американец . 294 (6): 44–51. Бибкод : 2006SciAm.294f..44B . doi : 10.1038/scientificamerican0606-44 . ПМИД 16711359 .
- ^ Хьюз Р.А., Эллингтон AD (январь 2017 г.). «Синтез и сборка синтетической ДНК: использование синтетического в синтетической биологии» . Перспективы Колд-Спринг-Харбор в биологии . 9 (1): а023812. doi : 10.1101/cshperspect.a023812 . ПМК 5204324 . ПМИД 28049645 .
- ^ Косури С., генеральный директор Церкви (май 2014 г.). «Крупномасштабный синтез ДНК de novo: технологии и приложения» . Природные методы . 11 (5): 499–507. дои : 10.1038/nmeth.2918 . ПМК 7098426 . ПМИД 24781323 .
- ^ Блайт К.Дж., Колыхалов А.А., Райс К.М. (декабрь 2000 г.). «Эффективное инициирование репликации РНК ВГС в культуре клеток». Наука . 290 (5498): 1972–4. Бибкод : 2000Sci...290.1972B . дои : 10.1126/science.290.5498.1972 . ПМИД 11110665 .
- ^ Кузин Дж. (июль 2002 г.). «Вирусология. Активный полиовирус, испеченный с нуля». Наука . 297 (5579): 174–5. дои : 10.1126/science.297.5579.174b . ПМИД 12114601 . S2CID 83531627 .
- ^ Смит Х.О., Хатчисон К.А., Пфанкох К., Вентер Дж.К. (декабрь 2003 г.). «Создание синтетического генома путем сборки всего генома: бактериофаг phiX174 из синтетических олигонуклеотидов» . Труды Национальной академии наук Соединенных Штатов Америки . 100 (26): 15440–5. Бибкод : 2003PNAS..10015440S . дои : 10.1073/pnas.2237126100 . ПМК 307586 . ПМИД 14657399 .
- ^ Уэйд Н (29 июня 2007 г.). «Ученые трансплантируют геном бактерий» . Нью-Йорк Таймс . ISSN 0362-4331 . Проверено 28 декабря 2007 г.
- ^ Гибсон Д.Г., Бендерс Г.А., Эндрюс-Пфанкох С., Денисова Е.А., Баден-Тилсон Х., Завери Дж., Стоквелл Т.Б., Браунли А., Томас Д.В., Алжир М.А., Мерриман С., Янг Л., Носков В.Н., Гласс Дж.И., Вентер Дж.К., Хатчисон Калифорния, Смит Х.О. (февраль 2008 г.). «Полный химический синтез, сборка и клонирование генома Mycoplasmaogenicium». Наука . 319 (5867): 1215–20. Бибкод : 2008Sci...319.1215G . дои : 10.1126/science.1151721 . ПМИД 18218864 . S2CID 8190996 .
- ^ Болл П (2016). «Сделано человеком: история синтетической жизни» . Дистилляции . 2 (1): 15–23 . Проверено 22 марта 2018 г.
- ^ Поллак А (12 сентября 2007 г.). «Как вам ваши гены? Биофаббы принимают заказы» . Нью-Йорк Таймс . ISSN 0362-4331 . Проверено 28 декабря 2007 г.
- ^ «Проекты синтетической биологии» . arep.med.harvard.edu . Проверено 17 февраля 2018 г.
- ^ Форстер AC, генеральный директор Черча (22 августа 2006 г.). «На пути к синтезу минимальной клетки» . Молекулярная системная биология . 2 (1): 45. дои : 10.1038/msb4100090 . ПМК 1681520 . ПМИД 16924266 .
- ^ Jump up to: Перейти обратно: а б Басульто Д. (4 ноября 2015 г.). «Все, что вам нужно знать о том, почему CRISPR является такой популярной технологией» . Вашингтон Пост . Проверено 5 декабря 2015 г.
- ^ Кан Дж. (9 ноября 2015 г.). «Загадка Криспра» . Нью-Йорк Таймс . Проверено 5 декабря 2015 г.
- ^ Ледфорд Х (3 июня 2015 г.). «CRISPR, разрушитель» . Природа . 522 (7554): 20–4. Бибкод : 2015Natur.522...20L . дои : 10.1038/522020a . ПМИД 26040877 .
{{cite journal}}: Неизвестный параметр|agency=игнорируется ( помогите ) - ^ Хиггинботэм С. (4 декабря 2015 г.). «Ведущие венчурные капиталисты говорят, что редактирование генов более рискованно, чем искусственный интеллект» . Удача . Проверено 5 декабря 2015 г.
- ^ Ролли; и др. (2012). «Проектирование биологических систем: системная инженерия встречается с синтетической биологией». Химико-техническая наука . 69 (1): 1–29. Бибкод : 2012ЧЭнС..69....1Р . дои : 10.1016/j.ces.2011.10.068 .
- ^ Jump up to: Перейти обратно: а б Халил А.С., Коллинз Дж.Дж. (май 2010 г.). «Синтетическая биология: приложения достигают совершеннолетия» . Обзоры природы. Генетика . 11 (5): 367–379. дои : 10.1038/nrg2775 . ПМК 2896386 . ПМИД 20395970 .
- ^ Jump up to: Перейти обратно: а б Фримонт П.С., Китни Т.И. (2012). Учебник Синтетическая биология . . Всемирная научная. дои : 10.1142/p837 . ISBN 978-1-84816-863-3 .
- ^ Jump up to: Перейти обратно: а б Райт Б.В., Моллой, член парламента, Яшке П.Р. (март 2022 г.). «Перекрывающиеся гены в природных и искусственно созданных геномах» . Обзоры природы. Генетика . 23 (3): 154–168. дои : 10.1038/s41576-021-00417-w . ПМЦ 8490965 . ПМИД 34611352 .
- ^ Jump up to: Перейти обратно: а б Чан Л.И., Косури С., Энди Д. (2005). «Рефакторинг бактериофага Т7» . Молекулярная системная биология . 1 :2005.0018. дои : 10.1038/msb4100025 . ПМК 1681472 . ПМИД 16729053 .
- ^ Темме К., Чжао Д., Фойгт Калифорния (май 2012 г.). «Рефакторинг кластера генов азотфиксации Klebsiella oxytoca» . Труды Национальной академии наук Соединенных Штатов Америки . 109 (18): 7085–7090. Бибкод : 2012PNAS..109.7085T . дои : 10.1073/pnas.1120788109 . ПМК 3345007 . ПМИД 22509035 .
- ^ Сонг М, Сукович Д.Д., Сиссарелли Л., Майр Дж., Фернандес-Родригес Дж., Мирский Е.А. и др. (май 2017 г.). «Контроль секреции белка типа III с использованием минимальной генетической системы» . Природные коммуникации . 8 (1): 14737. Бибкод : 2017NatCo...814737S . дои : 10.1038/ncomms14737 . ПМК 5436071 . ПМИД 28485369 .
- ^ Яшке П.Р., Либерман Э.К., Родригес Дж., Сьерра А., Энди Д. (декабрь 2012 г.). «Полностью декомпрессированный синтетический геном бактериофага øX174, собранный и заархивированный в дрожжах» . Вирусология . 434 (2): 278–284. дои : 10.1016/j.virol.2012.09.020 . ПМИД 23079106 .
- ^ Вульфсон Д.Н., Бартлетт Г.Дж., Брюнинг М., Томсон А.Р. (август 2012 г.). «Новая валюта за старый канат: от витых узлов до α-спиральных стволов». Современное мнение в области структурной биологии . 22 (4): 432–41. дои : 10.1016/j.sbi.2012.03.002 . ПМИД 22445228 .
- ^ Дубер Дж.Э., Ву Г.К., Малмирчегини Г.Р., Мун Т.С., Петцольд С.Дж., Уллал А.В., Пратер К.Л., Кислинг Дж.Д. (август 2009 г.). «Синтетические белковые каркасы обеспечивают модульный контроль над метаболическим потоком». Природная биотехнология . 27 (8): 753–9. дои : 10.1038/nbt.1557 . ПМИД 19648908 . S2CID 2756476 .
- ^ Реддингтон СК, Ховарт М. (декабрь 2015 г.). «Секреты ковалентного взаимодействия биоматериалов и биотехнологий: SpyTag и SpyCatcher» . Современное мнение в области химической биологии . 29 : 94–9. дои : 10.1016/j.cbpa.2015.10.002 . ПМИД 26517567 .
- ^ Бэйл Дж. Х., Гримли Дж. С., Станкунас К., Гествицки Дж. Э., Уэндлесс Т. Дж., Крэбтри Г. Р. (январь 2006 г.). «Аналоги рапамицина с дифференциальной специфичностью связывания позволяют осуществлять ортогональный контроль активности белка» . Химия и биология . 13 (1): 99–107. doi : 10.1016/j.chembiol.2005.10.017 . ПМИД 16426976 .
- ^ Альтшилер Э., Вентура А., Колман-Лернер А., Черноморец А. (октябрь 2014 г.). «Влияние ограничений восходящего и нисходящего потока на сверхчувствительность модуля сигнализации» . Физическая биология . 11 (6): 066003. Бибкод : 2014PhBio..11f6003A . дои : 10.1088/1478-3975/11/6/066003 . ПМЦ 4233326 . ПМИД 25313165 .
- ^ Альтшилер Э., Вентура А.С., Колман-Лернер А., Черноморец А. (2017). «Возврат к сверхчувствительности в сигнальных каскадах: объединение оценок локальной и глобальной сверхчувствительности» . ПЛОС ОДИН . 12 (6): e0180083. arXiv : 1608.08007 . Бибкод : 2017PLoSO..1280083A . дои : 10.1371/journal.pone.0180083 . ПМЦ 5491127 . ПМИД 28662096 .
- ^ Карбонелл-Баллестеро М., Дюран-Небреда С., Монтаньес Р., Соле Р., Масия Х., Родригес-Касо С. (декабрь 2014 г.). «Характеристика передаточных функций снизу вверх для проектов синтетической биологии: уроки энзимологии» . Исследования нуклеиновых кислот . 42 (22): 14060–14069. дои : 10.1093/nar/gku964 . ПМК 4267673 . ПМИД 25404136 .
- ^ Казнессис Ю.Н. (ноябрь 2007 г.). «Модели для синтетической биологии» . Системная биология BMC . 1 (1): 47. дои : 10.1186/1752-0509-1-47 . ПМК 2194732 . ПМИД 17986347 .
- ^ Туза З.А., Сингхал В., Ким Дж., Мюррей Р.М. (декабрь 2013 г.). «Набор инструментов моделирования in silico для быстрого прототипирования схем в биомолекулярной «макетной» системе». 52-я конференция IEEE по принятию решений и управлению . дои : 10.1109/CDC.2013.6760079 .
- ^ ЛаФлер Т.Л., Хоссейн А., Салис Х.М. (сентябрь 2022 г.). «Автоматизированный модельно-прогностический дизайн синтетических промоторов для контроля профилей транскрипции у бактерий» . Природные коммуникации . 13 (1): 5159. Бибкод : 2022NatCo..13.5159L . дои : 10.1038/s41467-022-32829-5 . ПМК 9440211 . ПМИД 36056029 .
- ^ Казнессис, Яннис Н (2007). «Модели для синтетической биологии» . Системная биология BMC . 1 (1): 47. дои : 10.1186/1752-0509-1-47 . ISSN 1752-0509 . ПМК 2194732 . ПМИД 17986347 .
- ^ Элани Ю. (июнь 2016 г.). «Создание мембраносвязанных искусственных клеток с использованием микрофлюидики: новый рубеж в восходящей синтетической биологии» . Труды Биохимического общества . 44 (3): 723–30. дои : 10.1042/BST20160052 . ПМЦ 4900754 . ПМИД 27284034 .
- ^ Гач ПК, Иваи К., Ким П.В., Хиллсон Н.Дж., Сингх А.К. (октябрь 2017 г.). «Капельная микрофлюидика для синтетической биологии» . Лаборатория на чипе . 17 (20): 3388–3400. дои : 10.1039/C7LC00576H . ОСТИ 1421856 . ПМИД 28820204 . S2CID 206094816 .
- ^ Винуселви П., Пак С., Ким М., Пак Дж.М., Ким Т., Ли С.К. (3 июня 2011 г.). «Микрофлюидные технологии для синтетической биологии» . Международный журнал молекулярных наук . 12 (6): 3576–93. дои : 10.3390/ijms12063576 . ПМЦ 3131579 . ПМИД 21747695 .
- ^ Jump up to: Перейти обратно: а б Халил А.С., Лу Т.К., Башор С.Дж., Рамирес К.Л., Пайенсон Н.С., Йонг Дж.К., Коллинз Дж.Дж. (август 2012 г.). «Система синтетической биологии для программирования функций транскрипции эукариот» . Клетка . 150 (3): 647–58. дои : 10.1016/j.cell.2012.05.045 . ПМЦ 3653585 . ПМИД 22863014 .
- ^ «Синтетическая биология» . Genome.gov . Проверено 23 января 2023 г.
- ^ де Алмейда П.Е., ван Раппард-младший, Ву Дж.К. (сентябрь 2011 г.). «Биолюминесценция in vivo для отслеживания судьбы и функции клеток» . Американский журнал физиологии. Физиология сердца и кровообращения . 301 (3): H663–71. дои : 10.1152/ajpheart.00337.2011 . ПМК 3191083 . ПМИД 21666118 .
- ^ Клоуз Д.М., Сюй Т., Сэйлер Г.С., Рипп С. (2011). «Биолюминесцентная визуализация in vivo (BLI): неинвазивная визуализация и исследование биологических процессов у живых животных» . Датчики . 11 (1): 180–206. дои : 10.3390/s110100180 . ПМК 3274065 . ПМИД 22346573 .
- ^ Гиббс WW (1997). «Звери на чипе» . Научный американец . Проверено 2 марта 2009 г.
- ^ Белкин С., Ягур-Кролл С., Кабесса Ю., Корума В., Септон Т., Анати Ю. и др. (апрель 2017 г.). «Дистанционное обнаружение заглубленных мин с помощью бактериального сенсора». Природная биотехнология . 35 (4): 308–310. дои : 10.1038/nbt.3791 . ПМИД 28398330 . S2CID 3645230 .
- ^ Данино Т., Приндл А., Квонг Г.А., Скалак М., Ли Х., Аллен К., Хасти Дж., Бхатия С.Н. (май 2015 г.). «Программируемые пробиотики для выявления рака в моче» . Наука трансляционной медицины . 7 (289): 289ра84. doi : 10.1126/scitranslmed.aaa3519 . ПМЦ 4511399 . ПМИД 26019220 .
- ^ «Маски для лица, которые позволяют диагностировать COVID-19» . www.medicalxpress.com . Проверено 11 июля 2021 г.
- ^ Нгуен П.К., Соенксен Л.Р., Донгиа Н.М., Ангенент-Мари Н.М., де Пуч Х., Хуанг А. и др. (ноябрь 2021 г.). «Носимые материалы со встроенными сенсорами синтетической биологии для обнаружения биомолекул» . Природная биотехнология . 39 (11): 1366–1374. дои : 10.1038/s41587-021-00950-3 . hdl : 1721.1/131278 . ПМИД 34183860 . S2CID 235673261 .
- ^ Халил, Ахмад С.; Коллинз, Джеймс Дж. (май 2010 г.). «Синтетическая биология: приложения достигают зрелости» . Обзоры природы Генетика . 11 (5): 367–379. дои : 10.1038/nrg2775 . ISSN 1471-0064 . ПМК 2896386 . ПМИД 20395970 .
- ^ «Более пристальный взгляд на клеточное сельское хозяйство и определяющие его процессы — AgFunderNews» . 05.07.2016 . Проверено 5 августа 2016 г.
- ^ Брайант, Кристофер Дж. (3 августа 2020 г.). «Культура, мясо и культивированное мясо» . Журнал зоотехники . 98 (8): скаа172. дои : 10.1093/jas/skaa172 . ISSN 0021-8812 . ПМЦ 7398566 . ПМИД 32745186 .
- ^ Хон, Тэ Гён; Шин, Донг-Мин; Чхве, Джунхёк; До, Чон Тэ; Хан, Сунг Гу (май 2021 г.). «Актуальные проблемы и технические достижения в производстве культивированного мяса: AReview» . Пищевая наука о животных ресурсах . 41 (3): 355–372. дои : 10.5851/kosfa.2021.e14 . ISSN 2636-0772 . ПМЦ 8112310 . ПМИД 34017947 .
- ^ Трайх, Николас (1 мая 2021 г.). «Культурное мясо: перспективы и вызовы» . Экономика окружающей среды и ресурсов . 79 (1): 33–61. дои : 10.1007/s10640-021-00551-3 . ISSN 1573-1502 . ПМЦ 7977488 . ПМИД 33758465 .
- ^ Мэттик, CS (январь 2018 г.). «Клеточное сельское хозяйство: грядущая революция в производстве продуктов питания». Бюллетень ученых-атомщиков . 74 (1): 32–35. Бибкод : 2018БуАтС..74а..32М . дои : 10.1080/00963402.2017.1413059 . S2CID 149404346 .
- ^ «Японские учёные впервые напечатали на 3D-принтере мраморную говядину вагю » Новый Атлас . 25 августа 2021 г. Проверено 21 сентября 2021 г.
- ^ Кан Д.Х., Луис Ф., Лю Х., Симода Х., Нисияма Ю., Нодзава Х. и др. (август 2021 г.). «Создана цельная мясоподобная ткань путем сборки клеточных волокон с использованием биопечати, интегрированной в сухожильный гель» . Природные коммуникации . 12 (1): 5059. Бибкод : 2021NatCo..12.5059K . дои : 10.1038/s41467-021-25236-9 . ПМК 8385070 . ПМИД 34429413 .
- ^ Лаварс Н. (20 сентября 2021 г.). «Кофе, выращенный в лаборатории, сокращает количество бобов и вырубку лесов» . Новый Атлас . Проверено 18 октября 2021 г.
- ^ «Экологически чистый, выращенный в лаборатории кофе уже в пути, но у него есть одна загвоздка» . Хранитель . 16 октября 2021 г. Проверено 26 октября 2021 г.
- ^ «Экологичный кофе, выращенный в Финляндии» . Новости ВТТ . 15 сентября 2021 г. Проверено 18 октября 2021 г.
- ^ «Выращивание продуктов питания с помощью воздуха и солнечной энергии: более эффективно, чем посадка сельскохозяйственных культур» . физ.орг . Проверено 11 июля 2021 г.
- ^ Леже Д., Матасса С., Нур Э., Шепон А., Майло Р., Бар-Эвен А. (июнь 2021 г.). «Производство микробного белка с помощью фотоэлектрической энергии может использовать землю и солнечный свет более эффективно, чем традиционные культуры» . Труды Национальной академии наук Соединенных Штатов Америки . 118 (26): e2015025118. Бибкод : 2021PNAS..11815025L . дои : 10.1073/pnas.2015025118 . ПМЦ 8255800 . ПМИД 34155098 . S2CID 235595143 .
- ^ «Первый в мире искусственный синтез крахмала из CO2 превосходит природный» . Новый Атлас . 28 сентября 2021 г. Проверено 18 октября 2021 г.
- ^ Цай Т., Сунь Х., Цяо Дж., Чжу Л., Чжан Ф., Чжан Дж. и др. (сентябрь 2021 г.). «Бесклеточный химиоферментативный синтез крахмала из углекислого газа» . Наука . 373 (6562): 1523–1527. Бибкод : 2021Sci...373.1523C . дои : 10.1126/science.abh4049 . ПМИД 34554807 . S2CID 237615280 .
- ^ «Паучий шелк, созданный фотосинтезирующими бактериями» . физ.орг . Архивировано из оригинала 7 августа 2020 года . Проверено 16 августа 2020 г. .
- ^ Фунг С.П., Хигучи-Такеучи М., Малай А.Д., Октавиани Н.А., Тагун С., Нумата К. (июль 2020 г.). «Фабрика морских фотосинтетических микробных клеток как платформа для производства паучьего шелка» . Коммуникационная биология . 3 (1). Springer Science and Business Media LLC: 357. doi : 10.1038/s42003-020-1099-6 . ПМЦ 7343832 . ПМИД 32641733 .
- ^ Сингх В. (декабрь 2014 г.). «Последние достижения и возможности в области разработки синтетических логических вентилей в живых клетках» . Системы и синтетическая биология . 8 (4): 271–82. дои : 10.1007/s11693-014-9154-6 . ПМЦ 4571725 . ПМИД 26396651 .
- ^ Перселл О, Лу Т.К. (октябрь 2014 г.). «Синтетические аналоговые и цифровые схемы для клеточных вычислений и памяти» . Современное мнение в области биотехнологии . Клеточная и дорожная инженерия. 29 : 146–55. дои : 10.1016/j.copbio.2014.04.009 . ПМК 4237220 . ПМИД 24794536 .
- ^ Дэниел Р., Рубенс-младший, Сарпешкар Р., Лу Т.К. (май 2013 г.). «Синтетические аналоговые вычисления в живых клетках». Природа . 497 (7451): 619–23. Бибкод : 2013Natur.497..619D . дои : 10.1038/nature12148 . ПМИД 23676681 . S2CID 4358570 .
- ^ Ринаудо К., Блерис Л., Маддамсетти Р., Субраманиан С., Вайс Р., Бененсон Ю. (июль 2007 г.). «Универсальный логический оценщик на основе РНКи, работающий в клетках млекопитающих». Природная биотехнология . 25 (7): 795–801. дои : 10.1038/nbt1307 . ПМИД 17515909 . S2CID 280451 .
- ^ Се З, Вроблевска Л, Прохазка Л, Вайс Р, Бененсон Ю (сентябрь 2011 г.). «Многовходовая логическая схема на основе РНКи для идентификации специфических раковых клеток» . Наука . 333 (6047): 1307–11. Бибкод : 2011Sci...333.1307X . дои : 10.1126/science.1205527 . ПМИД 21885784 . S2CID 13743291 .
- ^ Нильсен А.А., Дер Б.С., Шин Дж., Вайдьянатан П., Параланов В., Стричалски Е.А., Росс Д., Денсмор Д., Фойгт К.А. (апрель 2016 г.). «Автоматизация проектирования генетических схем» . Наука . 352 (6281): аас7341. дои : 10.1126/science.aac7341 . ПМИД 27034378 .
- ^ Вайнберг Б.Х., Фам Н.Т., Карабалло Л.Д., Лозаноски Т., Энгель А., Бхатия С., Вонг В.В. (май 2017 г.). «Крупномасштабное проектирование надежных генетических цепей с множеством входов и выходов для клеток млекопитающих» . Природная биотехнология . 35 (5): 453–462. дои : 10.1038/nbt.3805 . ПМЦ 5423837 . ПМИД 28346402 .
- ^ Панди А., Кох М., Войводик П.Л., Судье П., Бонне Дж., Кушваха М., Фаулон Дж.Л. (август 2019 г.). «Метаболические перцептроны для нейронных вычислений в биологических системах» . Природные коммуникации . 10 (1): 3880. Бибкод : 2019NatCo..10.3880P . дои : 10.1038/s41467-019-11889-0 . ПМК 6713752 . ПМИД 31462649 .
- ^ Вестфол П.Дж., Питера DJ, Ленихан Дж.Р., Энг Д., Вулард FX, Регентин Р., Хорнинг Т., Цурута Х., Мелис Дж.Д., Оуэнс А., Фикес С., Диола Д., Бенджамин К.Р., Кислинг Дж.Д., Ливелл М.Д., Макфи DJ, Реннингер Н.С. , Ньюман Джей Ди, Пэддон Си Джей (январь 2012 г.). «Производство аморфадиена в дрожжах и его превращение в дигидроартемизиновую кислоту, предшественник противомалярийного агента артемизинина» . Труды Национальной академии наук Соединенных Штатов Америки . 109 (3): E111–8. Бибкод : 2012PNAS..109E.111W . дои : 10.1073/pnas.1110740109 . ПМК 3271868 . ПМИД 22247290 .
- ^ Коннор С. (28 марта 2014 г.). «Эврика! Ученые совершили гигантский скачок к синтетической жизни» . Независимый . Архивировано из оригинала 26 мая 2022 г. Проверено 6 августа 2015 г.
- ^ Нгуен П.К., Ботянски З., Тай П.К., Джоши Н.С. (сентябрь 2014 г.). «Программируемые материалы на основе биопленок из инженерных нановолокон Curli» . Природные коммуникации . 5 : 4945. Бибкод : 2014NatCo...5.4945N . дои : 10.1038/ncomms5945 . ПМИД 25229329 .
- ^ Кульман Б., Дантас Дж., Иретон Г.К., Варани Г., Стоддард Б.Л., Бейкер Д. (ноябрь 2003 г.). «Разработка новой складки глобулярного белка с точностью на атомном уровне» . Наука . 302 (5649): 1364–8. Бибкод : 2003Sci...302.1364K . дои : 10.1126/science.1089427 . ПМИД 14631033 . S2CID 1939390 .
- ^ Кодер Р.Л., Андерсон Дж.Л., Соломон Л.А., Редди К.С., Мозер CC, Даттон П.Л. (март 2009 г.). «Дизайн и разработка белка-транспортера O (2)» . Природа . 458 (7236): 305–309. Бибкод : 2009Natur.458..305K . дои : 10.1038/nature07841 . ПМЦ 3539743 . ПМИД 19295603 .
- ^ Фарид Т.А., Кодали Г., Соломон Л.А., Лихтенштейн Б.Р., Шихан М.М., Фрай Б.А. и др. (декабрь 2013 г.). «Дизайн элементарного тетраспирального белка для разнообразных функций оксидоредуктазы» . Химическая биология природы . 9 (12): 826–833. дои : 10.1038/следующий.1362 . ПМК 4034760 . ПМИД 24121554 .
- ^ Ван М.С., Хехт М.Х. (сентябрь 2020 г.). «Полностью АТФаза De Novo из комбинаторного дизайна белков». Журнал Американского химического общества . 142 (36): 15230–15234. дои : 10.1021/jacs.0c02954 . ПМИД 32833456 . S2CID 221308093 .
- ^ Армбрустер Б.Н., Ли Х, Пауш М.Х., Херлитце С., Рот Б.Л. (март 2007 г.). «Развитие замка, чтобы он соответствовал ключу, для создания семейства рецепторов, связанных с G-белком, мощно активируемых инертным лигандом» . Труды Национальной академии наук Соединенных Штатов Америки . 104 (12): 5163–5168. Бибкод : 2007PNAS..104.5163A . дои : 10.1073/pnas.0700293104 . ПМЦ 1829280 . ПМИД 17360345 .
- ^ Мак В.С., Тран С., Маркески Р., Бертолани С., Томпсон Дж., Бейкер Д. и др. (ноябрь 2015 г.). «Интегративный геномный анализ функций ферментов, позволяющий разработать неприродный путь биосинтеза» . Природные коммуникации . 6 : 10005. Бибкод : 2015NatCo...610005M . дои : 10.1038/ncomms10005 . ПМЦ 4673503 . ПМИД 26598135 .
- ^ Ван Ц, Пэрриш А.Р., Ван Л. (март 2009 г.). «Расширение генетического кода для биологических исследований» . Химия и биология . 16 (3): 323–36. doi : 10.1016/j.chembiol.2009.03.001 . ПМК 2696486 . ПМИД 19318213 .
- ^ Дэвидсон А.Р., Ламб К.Дж., Зауэр Р.Т. (октябрь 1995 г.). «Кооперативно свернутые белки в библиотеках случайных последовательностей». Структурная биология природы . 2 (10): 856–864. дои : 10.1038/nsb1095-856 . ПМИД 7552709 . S2CID 31781262 .
- ^ Камтекар С., Шиффер Дж. М., Сюн Х., Бабик Дж. М., Хехт М. Х. (декабрь 1993 г.). «Дизайн белка путем бинарного формирования паттерна полярных и неполярных аминокислот». Наука . 262 (5140): 1680–1685. Бибкод : 1993Sci...262.1680K . дои : 10.1126/science.8259512 . ПМИД 8259512 .
- ^ Уолтер К.У., Вамвача К., Хилверт Д. (ноябрь 2005 г.). «Активный фермент, построенный на основе алфавита из 9 аминокислот» . Журнал биологической химии . 280 (45): 37742–37746. дои : 10.1074/jbc.M507210200 . ПМИД 16144843 .
- ^ «Приложения синтетической биологии» . www.thermofisher.com . Проверено 12 ноября 2015 г.
- ^ Лю Ю, Шин Х.Д., Ли Дж., Лю Л. (февраль 2015 г.). «На пути к метаболической инженерии в контексте системной биологии и синтетической биологии: достижения и перспективы». Прикладная микробиология и биотехнология . 99 (3): 1109–18. дои : 10.1007/s00253-014-6298-y . ПМИД 25547833 . S2CID 954858 .
- ^ ГМ Черча, Гао Ю, Косури С (сентябрь 2012 г.). «Цифровое хранилище информации нового поколения в ДНК» . Наука . 337 (6102): 1628. Бибкод : 2012Sci...337.1628C . дои : 10.1126/science.1226355 . ПМИД 22903519 . S2CID 934617 .
- ^ «Огромные объемы данных могут храниться в ДНК» . Небесные новости. 23 января 2013 г. Архивировано из оригинала 31 мая 2016 г. Проверено 24 января 2013 г.
- ^ Заде Дж.Н., Стинберг С.Д., Буа Дж.С., Вулф Б.Р., Пирс М.Б., Хан А.Р. и др. (январь 2011 г.). «НУПАК: Анализ и дизайн систем нуклеиновых кислот» . Журнал вычислительной химии . 32 (1): 170–173. дои : 10.1002/jcc.21596 . ПМИД 20645303 . S2CID 33709556 .
- ^ Лоренц Р., Бернхарт С.Х., Хёнер Цу Зидердиссен С., Тафер Х., Фламм С., Стадлер П.Ф., Хофакер И.Л. (ноябрь 2011 г.). «Пакет ВенскойРНК 2.0» . Алгоритмы молекулярной биологии . 6 (1): 26. дои : 10.1186/1748-7188-6-26 . ПМК 3319429 . ПМИД 22115189 .
- ^ Салис Х.М., Мирский Е.А., Фойгт К.А. (октябрь 2009 г.). «Автоматизированный дизайн сайтов связывания синтетических рибосом для контроля экспрессии белка» . Природная биотехнология . 27 (10): 946–950. дои : 10.1038/nbt.1568 . ПМЦ 2782888 . ПМИД 19801975 .
- ^ Нильсен А.А., Дер Б.С., Шин Дж., Вайдьянатан П., Параланов В., Стрыхальский Е.А. и др. (апрель 2016 г.). «Автоматизация проектирования генетических схем» . Наука . 352 (6281): аас7341. дои : 10.1126/science.aac7341 . ПМИД 27034378 .
- ^ Хоссейн А., Лопес Е., Халпер С.М., Цетнар Д.П., Рейс А.С., Стрикланд Д. и др. (декабрь 2020 г.). «Автоматическое проектирование тысяч неповторяющихся деталей для создания стабильных генетических систем» . Природная биотехнология . 38 (12): 1466–1475. дои : 10.1038/s41587-020-0584-2 . ПМИД 32661437 . S2CID 220506228 .
- ^ Поллак А (7 мая 2014 г.). «Исследователи сообщают о прорыве в создании искусственного генетического кода» . Нью-Йорк Таймс . Проверено 7 мая 2014 г.
- ^ Каллауэй Э (7 мая 2014 г.). «Первая жизнь с «чужой» ДНК» . Природа . дои : 10.1038/nature.2014.15179 . S2CID 86967999 . Проверено 7 мая 2014 г.
- ^ Jump up to: Перейти обратно: а б Малышев Д.А., Дхами К., Лавернь Т., Чен Т., Дай Н., Фостер Дж.М., Корреа И.Р., Ромесберг Ф.Е. (май 2014 г.). «Полусинтетический организм с расширенным генетическим алфавитом» . Природа . 509 (7500): 385–8. Бибкод : 2014Natur.509..385M . дои : 10.1038/nature13314 . ПМК 4058825 . ПМИД 24805238 .
- ^ Jump up to: Перейти обратно: а б Версе CN, Паулино-Лима И.Г., Баке М., Билли Д., Ротшильд Л.Дж. (2016). «Синтетическая биология для освоения космоса: перспективы и социальные последствия». Амбивалентности создания жизни . Этика оценки науки и технологий. Том. 45. Шпрингер-Верлаг. стр. 73–100. дои : 10.1007/978-3-319-21088-9_4 . ISBN 978-3-319-21087-2 .
- ^ Менезес А.А., Камберс Дж., Хоган Дж.А., Аркин А.П. (январь 2015 г.). «К синтетическим биологическим подходам к использованию ресурсов в космических полетах» . Журнал Королевского общества, Интерфейс . 12 (102): 20140715. doi : 10.1098/rsif.2014.0715 . ПМК 4277073 . ПМИД 25376875 .
- ^ Монтегю М., МакАртур Г.Х., Кокелл К.С., Хелд Дж., Маршалл В., Шерман Л.А. и др. (декабрь 2012 г.). «Роль синтетической биологии в использовании ресурсов in situ (ISRU)». Астробиология . 12 (12): 1135–1142. Бибкод : 2012AsBio..12.1135M . дои : 10.1089/ast.2012.0829 . ПМИД 23140229 .
- ^ Штайгервальд Б. «НАСА – дизайнерские растения на Марсе» . www.nasa.gov . Проверено 29 мая 2020 г.
- ^ Хатчисон К.А., Чуанг Р.Ю., Носков В.Н., Асад-Гарсия Н., Диринк Т.Дж., Эллисман М.Х., Гилл Дж., Каннан К., Карас Б.Дж., Ма Л., Пеллетье Дж.Ф., Ци ZQ, Рихтер Р.А., Стричалски Э.А., Сан Л., Сузуки Ю., Цветанова Б., Уайз К.С., Смит Х.О., Гласс Дж.И., Мерриман С., Гибсон Д.Г., Вентер Дж.К. (март 2016 г.). «Дизайн и синтез минимального бактериального генома» . Наука . 351 (6280): аад6253. Бибкод : 2016Sci...351.....H . дои : 10.1126/science.aad6253 . ПМИД 27013737 .
- ^ Jump up to: Перейти обратно: а б Коннор С. (1 декабря 2014 г.). «Крупный прорыв в области синтетической жизни: ученые создали первые искусственные ферменты» . Независимый . Лондон. Архивировано из оригинала 26 мая 2022 г. Проверено 6 августа 2015 г.
- ^ Jump up to: Перейти обратно: а б Димер Д. (июль 2005 г.). «Гигантский шаг к искусственной жизни?». Тенденции в биотехнологии . 23 (7): 336–338. дои : 10.1016/j.tibtech.2005.05.008 . ПМИД 15935500 .
- ^ «Ученые достигли важной вехи на пути к искусственной жизни» . ЭНЕРГЕТИЧЕСКИЙ ЯДЕРНЫЙ РЕАКТОР . 20 мая 2010 г. Проверено 9 июня 2010 г.
- ^ Вентер Дж.К. «От проектирования жизни к продлению здоровой жизни» . Ютуб . Телевидение Калифорнийского университета (UCTV). Архивировано из оригинала 21 декабря 2021 г. Проверено 1 февраля 2017 г.
- ^ «Построй ячейку» . Проверено 4 декабря 2019 г.
- ^ «ФабриСелл» . Проверено 8 декабря 2019 г.
- ^ «MaxSynBio - Исследовательская сеть Макса Планка в области синтетической биологии» . Проверено 8 декабря 2019 г.
- ^ «БаСик» . Проверено 8 декабря 2019 г.
- ^ «СинСелл ЕС» . Проверено 8 декабря 2019 г.
- ^ Девлин, Ханна; корреспондент Ханна Девлин Science (14 июня 2023 г.). «Синтетические человеческие эмбрионы, созданные с помощью революционных достижений» . Хранитель . ISSN 0261-3077 . Проверено 30 ноября 2023 г.
- ^ Jump up to: Перейти обратно: а б с Халил, Ахмад С.; Коллинз, Джеймс Дж. (май 2010 г.). «Синтетическая биология: приложения достигают зрелости» . Обзоры природы Генетика . 11 (5): 367–379. дои : 10.1038/nrg2775 . ISSN 1471-0056 . ПМК 2896386 . ПМИД 20395970 . S2CID 2926016 .
- ^ Цзу С, Ван Дж (август 2014 г.). «Бактерии, колонизирующие опухоль: потенциальная терапия, нацеленная на опухоль». Критические обзоры по микробиологии . 40 (3): 225–235. дои : 10.3109/1040841X.2013.776511 . ПМИД 23964706 . S2CID 26498221 .
- ^ Гуджрати В., Ким С., Ким Ш., Мин Дж.Дж., Чой Х.Э., Ким С.К., Джон С. (февраль 2014 г.). «Биоинженерные бактериальные везикулы наружной мембраны как клеточно-специфические средства доставки лекарств для терапии рака». АСУ Нано . 8 (2): 1525–1537. дои : 10.1021/nn405724x . ПМИД 24410085 .
- ^ Пиньеро-Ламбеа К., Боделлон Г., Фернандес-Перианес Р., Куэста А.М., Альварес-Валлина Л., Фернандес Ла (апрель 2015 г.). «Программирование контролируемой адгезии E. coli к целевым поверхностям, клеткам и опухолям с помощью синтетических адгезинов» . ACS Синтетическая биология . 4 (4): 463–473. дои : 10.1021/sb500252a . ПМЦ 4410913 . ПМИД 25045780 .
- ^ Дейнеко И.В., Касниц Н., Лешнер С., Вайс С. (2016). «Состав специфического для опухоли бактериального промотора» . ПЛОС ОДИН . 11 (5): e0155338. Бибкод : 2016PLoSO..1155338D . дои : 10.1371/journal.pone.0155338 . ПМЦ 4865170 . ПМИД 27171245 .
- ^ Райс К.К., Бэйлс К.В. (март 2008 г.). «Молекулярный контроль гибели и лизиса бактерий» . Обзоры микробиологии и молекулярной биологии . 72 (1): 85–109, оглавление. дои : 10.1128/mmbr.00030-07 . ПМК 2268280 . ПМИД 18322035 .
- ^ Ганай С., Аренас РБ, Forbes NS (ноябрь 2009 г.). «Нацеленная на опухоль доставка TRAIL с использованием Salmonella typhimurium повышает выживаемость при раке молочной железы у мышей» . Британский журнал рака . 101 (10): 1683–1691. дои : 10.1038/sj.bjc.6605403 . ПМЦ 2778534 . ПМИД 19861961 .
- ^ Скотт, Бенджамин М.; Гутьеррес-Васкес, Кристина; Санмарко, Лилиана М.; да Силва Перейра, Джессика А.; Ли, Чжаорун; Пласенсия, Агустин; Хьюсон, Патрик; Кокс, Лаура М.; О'Брайен, Мэдлинн; Чен, Стивен К.; Мораес-Виейра, Педро М.; Чанг, Белинда С.В.; Пейсайович, Серджио Г.; Кинтана, Франсиско Дж. (июль 2021 г.). «Самонастраиваемые дрожжевые пробиотики для лечения воспалительных заболеваний кишечника». Природная медицина . 27 (7): 1212–1222. дои : 10.1038/s41591-021-01390-x . ISSN 1546-170Х . ПМИД 34183837 . S2CID 235673499 .
- ^ Чен, Кевин; Чжу, Исюань; Чжан, Юнгронг; Хамза, Терва; Ю, Хуа; Сен-Флер, Эшли; Гален, Джеймс; Ян, Чжиюн; Фэн, Ханьпин (28 октября 2020 г.). «Пробиотическая дрожжевая иммунотерапия против инфекции Clostridioides difficile» . Наука трансляционной медицины . 12 (567): eaax4905. doi : 10.1126/scitranslmed.aax4905 . ISSN 1946-6234 . ПМЦ 7692727 . ПМИД 33115949 .
- ^ Чен, И-Джу; Абила, Бамс; Мостафа Камель, Ясир (2023). «CAR-T: Что дальше?» . Раки . 15 (3): 663. doi : 10.3390/cancers15030663 . ISSN 2072-6694 . ПМЦ 9913679 . ПМИД 36765623 .
- ^ Джонс, Б.С., Лэмб, Л.С., Гольдман, Ф. и Ди Стази, А. Повышение безопасности продуктов клеточной терапии путем переноса генов-самоубийц. Передний. Фармакол. 5, 254 (2014).
- ^ Вэй П., Вонг В.В., Парк Дж.С., Коркоран Э.Э., Пейзайович С.Г., Онуффер Дж.Дж. и др. (август 2012 г.). «Бактериальные белки вирулентности как инструменты для перестройки киназных путей в дрожжах и иммунных клетках» . Природа . 488 (7411): 384–388. Бибкод : 2012Natur.488..384W . дои : 10.1038/nature11259 . ПМЦ 3422413 . ПМИД 22820255 .
- ^ Данино Т., Мондрагон-Паломино О., Цимринг Л., Хасти Дж. (январь 2010 г.). «Синхронизированный кворум генетических часов» . Природа . 463 (7279): 326–330. Бибкод : 2010Natur.463..326D . дои : 10.1038/nature08753 . ПМЦ 2838179 . ПМИД 20090747 .
- ^ Чен YY, Jensen MC, Smolke CD (май 2010 г.). «Генетический контроль пролиферации Т-клеток млекопитающих с помощью систем регуляции синтетической РНК» . Труды Национальной академии наук Соединенных Штатов Америки . 107 (19): 8531–8536. Бибкод : 2010PNAS..107.8531C . дои : 10.1073/pnas.1001721107 . ПМЦ 2889348 . ПМИД 20421500 .
- ^ Jump up to: Перейти обратно: а б Кац, Леонард; Чен, Ивонн Ю; Гонсалес, Рамон; Петерсон, Тодд С; Чжао, Хуэйминь; Бальц, Ричард Х (01 июля 2018 г.). «Достижения и применение синтетической биологии в биотехнологической промышленности: перспективы» . Журнал промышленной микробиологии и биотехнологии . 45 (7): 449–461. дои : 10.1007/s10295-018-2056-y . ISSN 1476-5535 . ПМИД 29915997 . S2CID 49296740 .
- ^ Хофер М., Лютольф, депутат парламента (май 2021 г.). «Инженерные органоиды» . Обзоры природы. Материалы . 6 (5): 402–420. Бибкод : 2021NatRM...6..402H . дои : 10.1038/s41578-021-00279-y . ПМЦ 7893133 . ПМИД 33623712 .
- ^ Уитакер, Мэтью (июль 2014 г.). «История 3D-печати в здравоохранении» . Бюллетень Королевского колледжа хирургов Англии . 96 (7): 228–229. дои : 10.1308/147363514X13990346756481 . ISSN 1473-6357 .
- ^ Атала А., Бауэр С.Б., Сокер С., Ю Дж.Дж., Ретик А.Б. (апрель 2006 г.). «Тканеинженерные аутологичные мочевые пузыри для пациентов, нуждающихся в цистопластике» . Ланцет . 367 (9518): 1241–6. дои : 10.1016/S0140-6736(06)68438-9 . ПМИД 16631879 . S2CID 17892321 .
- ^ Хонг Н., Ян Г.Х., Ли Дж., Ким Дж. (январь 2018 г.). «3D-биопечать и ее применение in vivo» . Журнал исследований биомедицинских материалов. Часть B: Прикладные биоматериалы . 106 (1): 444–459. дои : 10.1002/jbm.b.33826 . ПМИД 28106947 .
- ^ Шинкар, Кальяни; Род, Кавал (1 августа 2022 г.). «Может ли 3D-экструзионная биопечать стать реальной альтернативой трансплантации органов в будущем?» . Анналы 3D-печатной медицины . 7 : 100066. doi : 10.1016/j.stlm.2022.100066 . ISSN 2666-9641 . S2CID 249083907 .
- ^ «90-OR: 3D-биопечать бионической поджелудочной железы с сосудистой системой — результаты трансплантации крупным животным» . Сайт диабетических журналов . Проверено 26 октября 2023 г.
- ^ Зоммер AC, Блюменталь EZ (сентябрь 2019 г.). «Внедрение 3D-печати в офтальмологии». Архив клинической и экспериментальной офтальмологии Грефе . 257 (9): 1815–1822. дои : 10.1007/s00417-019-04312-3 . ПМИД 30993457 . S2CID 116884575 .
- ^ Клак, Марта; Бринярский, Томаш; Ковальска, Патриция; Гомолка, Магдалена; Тимицкий, Гжегож; Косовская, Катажина; Цивонюк, Петр; Добжанский, Томаш; Туровский, Павел; Вшола, Михал (30 июня 2020 г.). «Новые стратегии в разработке искусственных органов: каково будущее медицины?» . Микромашины . 11 (7): 646. дои : 10,3390/ми11070646 . ISSN 2072-666X . ПМК 7408042 . ПМИД 32629779 .
- ^ Цуй Х, Мяо С, Эсуорси Т, Чжоу X, Ли SJ, Лю С, Ю ZX, Фишер Дж. П., Мохиуддин М, Чжан Л. Г. (июль 2018 г.). «3D-биопечать для регенерации сердечно-сосудистой системы и фармакологии» . Обзоры расширенной доставки лекарств . 132 : 252–269. дои : 10.1016/j.addr.2018.07.014 . ПМК 6226324 . ПМИД 30053441 .
- ^ «Многоцентровое, одностороннее, проспективное, открытое, поэтапное исследование безопасности и эффективности конструкции AuriNovo для реконструкции предсердия у субъектов с односторонней микротией» . www.clinicaltrials.gov. 15 октября 2021 г. Проверено 19 июля 2022 г.
- ^ Рабин, Рони Кэрин (2 июня 2022 г.). «Врачи пересаживают ухо человеческие клетки, сделанные на 3D-принтере» . Нью-Йорк Таймс . Проверено 19 июля 2022 г.
- ^ Купер-Уайт М (1 марта 2015 г.). «Как 3D-печать может положить конец смертельной нехватке донорских органов» . Хаффпост Наука . TheHuffingtonPost.com, Inc. Проверено 17 февраля 2016 г.
- ^ Эдмундсон MC, Кейпесс М, Хорсфолл Л (декабрь 2014 г.). «Исследование потенциала металлических наночастиц в синтетической биологии» . Новая биотехнология . 31 (6): 572–578. дои : 10.1016/j.nbt.2014.03.004 . hdl : 20.500.11820/5cd4fa26-dee8-4862-86af-cf6a79546a13 . ПМИД 24681407 . S2CID 15790244 .
- ^ «Наночастицы уничтожают бляшки, вызывающие сердечные приступы» . Мичиганский государственный университет. 27 января 2020 года. Архивировано из оригинала 29 января 2020 года . Проверено 31 января 2020 г.
- ^ «Наночастицы помогают разъедать смертельные артериальные бляшки» . Новый Атлас . 28 января 2020 года. Архивировано из оригинала 1 марта 2020 года . Проверено 13 апреля 2020 г. .
- ^ Флорес А.М., Хоссейни-Нассаб Н., Джарр К.У., Йе Дж., Чжу X, Вирка Р. и др. (февраль 2020 г.). «Проэффероцитарные наночастицы специфически поглощаются повреждающими макрофагами и предотвращают атеросклероз» . Природные нанотехнологии . 15 (2): 154–161. Бибкод : 2020NatNa..15..154F . дои : 10.1038/s41565-019-0619-3 . ПМЦ 7254969 . ПМИД 31988506 .
- ^ «Исследования создают живые капли, производящие водород, прокладывая путь к альтернативному источнику энергии будущего» . физ.орг . Архивировано из оригинала 16 декабря 2020 года . Проверено 9 декабря 2020 г.
- ^ Сюй З, Ван С., Чжао С., Ли С., Лю Икс, Ван Л. и др. (ноябрь 2020 г.). «Фотосинтетическое производство водорода капельными микробными микрореакторами в аэробных условиях» . Природные коммуникации . 11 (1): 5985. Бибкод : 2020NatCo..11.5985X . дои : 10.1038/s41467-020-19823-5 . ПМЦ 7689460 . ПМИД 33239636 .
- ^ Се, Минци; Фюссенеггер, Мартин (июль 2015 г.). «Дизайнерские клетки млекопитающих: инженерные принципы и биомедицинские применения» . Биотехнологический журнал . 10 (7): 1005–1018. дои : 10.1002/biot.201400642 . ISSN 1860-6768 . ПМИД 26010998 . S2CID 11838264 .
- ^ Academic.oup.com https://academic.oup.com/proteincell/article/13/7/476/6747017 . Проверено 31 января 2024 г.
{{cite web}}: Отсутствует или пусто|title=( помощь ) - ^ Jump up to: Перейти обратно: а б с Мансури, Майсам; Фюссенеггер, Мартин (01 июня 2022 г.). «Электрогенетика: соединение синтетической биологии и электроники для дистанционного управления поведением дизайнерских клеток млекопитающих» . Современное мнение в области химической биологии . 68 : 102151. дои : 10.1016/j.cbpa.2022.102151 . hdl : 20.500.11850/547194 . ISSN 1367-5931 . ПМИД 35483127 .
- ^ Кравчик, Кшиштоф; Сюэ, Шуай; Бухманн, Питер; Шарпен-Эль-Хамри, Гислен; Саксена, Пратик; Юшерр, Мари-Дидье; Шао, Цзявэй; Да, Хайфэн; Се, Минци; Фюссенеггер, Мартин (29 мая 2020 г.). «Электрогенное клеточное высвобождение инсулина для контроля гликемии в реальном времени у мышей с диабетом 1 типа» . Наука . 368 (6494): 993–1001. дои : 10.1126/science.aau7187 . hdl : 20.500.11850/418642 . ISSN 0036-8075 . ПМИД 32467389 . S2CID 218984797 .
- ^ Маккласки, Джейн Т.; Хамид, Мухаджир; Го-Парке, Хун; МакКленаган, Невилл Х.; Гомис, Рамон; Флэтт, Питер Р. (24 июня 2011 г.). «Разработка и функциональная характеристика линий бета-клеток поджелудочной железы человека, высвобождающих инсулин, полученных методом электрослияния *» . Журнал биологической химии . 286 (25): 21982–21992. дои : 10.1074/jbc.M111.226795 . ISSN 0021-9258 . ПМК 3121343 . ПМИД 21515691 .
- ^ Jump up to: Перейти обратно: а б с Ньюсон Эй Джей (2015). «Синтетическая биология: этика, исключительность и ожидания» . Юридический журнал Маккуори . 15:45 .
- ^ «Границы 2018/19: новые экологические проблемы» . Программа ООН по окружающей среде . 04.03.2019 . Проверено 03 января 2021 г.
- ^ «Первые в мире дети с отредактированными генами созданы в Китае, - утверждает ученый» . Хранитель . Ноябрь 2018.
- ^ Хяйри М (апрель 2017 г.). «Синтетическая биология и этика: прошлое, настоящее и будущее». Кембриджский ежеквартальный журнал по этике здравоохранения . 26 (2): 186–205. дои : 10.1017/S0963180116000803 . ПМИД 28361718 . S2CID 29992939 .
- ^ Джин С., Кларк Б., Кузнесоф С., Лин Икс, Фруэр Л.Дж. (сентябрь 2019 г.). «Синтетическая биология, применяемая в агропродовольственном секторе: общественное восприятие, отношение и значение для будущих исследований» . Тенденции в пищевой науке и технологиях . 91 : 454–466. дои : 10.1016/j.tifs.2019.07.025 . S2CID 199632852 .
- ^ Гутманн А (2012). «Этика синтетической биологии: руководящие принципы для новых технологий». Отчет Гастингсского центра . 41 (4): 17–22. дои : 10.1002/j.1552-146X.2011.tb00118.x . ПМИД 21845917 . S2CID 20662786 .
- ^ Брайант CJ (август 2020 г.). «Культура, мясо и культивированное мясо» . Журнал зоотехники . 98 (8): скаа172. дои : 10.1093/jas/skaa172 . ПМЦ 7398566 . ПМИД 32745186 .
- ^ Хонг Т.К., Шин Д.М., Чхве Дж., До Дж.Т., Хан С.Г. (май 2021 г.). «Актуальные проблемы и технические достижения в производстве культивированного мяса: обзор» . Пищевая наука о животных ресурсах . 41 (3): 355–372. дои : 10.5851/kosfa.2021.e14 . ПМЦ 8112310 . ПМИД 34017947 .
- ^ Трейх Н. (1 мая 2021 г.). «Культурное мясо: перспективы и вызовы» . Экономика окружающей среды и ресурсов . 79 (1): 33–61. дои : 10.1007/s10640-021-00551-3 . ПМЦ 7977488 . ПМИД 33758465 .
- ^ Трейх Н. (май 2021 г.). «Культурное мясо: перспективы и вызовы» . Экономика окружающей среды и ресурсов . 79 (1): 33–61. дои : 10.1007/s10640-021-00551-3 . ПМЦ 7977488 . ПМИД 33758465 .
- ^ Jump up to: Перейти обратно: а б Ховард Дж., Мурашов В., Шульте П. (март 2017 г.). «Синтетическая биология и профессиональный риск». Журнал гигиены труда и окружающей среды . 14 (3): 224–236. дои : 10.1080/15459624.2016.1237031 . ПМИД 27754800 . S2CID 205893358 .
- ^ Jump up to: Перейти обратно: а б Европейская комиссия. Генеральный директорат по делам потребителей медицинских товаров (12 февраля 2016 г.). Мнение о синтетической биологии II: Методологии оценки рисков и аспекты безопасности . Офис публикаций. дои : 10.2772/63529 . ISBN 9789279439162 .
{{cite book}}:|website=игнорируется ( помогите ) - ^ Jump up to: Перейти обратно: а б Бюгль Х., Даннер Дж.П., Молинари Р.Дж., Маллиган Дж.Т., Парк Х.О., Райхерт Б., Рот Д.А., Вагнер Р., Будоул Б., Скрипп Р.М., Смит Дж.А., Стил С.Дж., Черч Дж., Энди Д. (июнь 2007 г.). «Синтез ДНК и биологическая безопасность». Природная биотехнология . 25 (6): 627–629. дои : 10.1038/nbt0607-627 . ПМИД 17557094 . S2CID 7776829 .
- ^ «Этические проблемы синтетической биологии: обзор дебатов» (PDF) .
- ^ Jump up to: Перейти обратно: а б Президентская комиссия по изучению проблем биоэтики, декабрь 2010 г. НОВЫЕ НАПРАВЛЕНИЯ Этика синтетической биологии и новых технологий. Архивировано 18 января 2017 г. на Wayback Machine . Проверено 14 апреля 2012 г.
- ^ Хаммель К. (31 августа 2020 г.). «Инженерные патогены и неестественное биологическое оружие: будущая угроза синтетической биологии» . Центр по борьбе с терроризмом в Вест-Пойнте . Проверено 5 января 2023 г.
- ^ Хаммель К (29 июня 2020 г.). «Взгляд из окопа CT: виртуальный круглый стол по вопросам COVID-19 и борьбы с терроризмом с Одри Курт Кронин, генерал-лейтенантом (в отставке) Майклом Нагатой, Магнусом Рансторпом, Али Суфаном и Хуаном Сарате» . Центр по борьбе с терроризмом в Вест-Пойнте . Проверено 5 января 2023 г.
- ^ Jump up to: Перейти обратно: а б «Синбиосейф» . Синбиосейф .
- ^ Шмидт М., Гангули-Митра А., Торгерсен Х., Келле А., Деплазес А., Биллер-Андорно Н. (декабрь 2009 г.). «Приоритетный документ по социальным и этическим аспектам синтетической биологии» (PDF) . Системы и синтетическая биология . 3 (1–4): 3–7. дои : 10.1007/s11693-009-9034-7 . ПМЦ 2759426 . ПМИД 19816794 .
- ^ Шмидт М. Келле А. Гангули А., де Вриенд Х. (ред.) 2009. «Синтетическая биология. Технонаука и ее социальные последствия». Академическое издательство Спрингера.
- ^ Келле А. (декабрь 2009 г.). «Обеспечение безопасности синтетической биологии – к стратегии управления 5P» . Системы и синтетическая биология . 3 (1–4): 85–90. дои : 10.1007/s11693-009-9041-8 . ПМЦ 2759433 . ПМИД 19816803 .
- ^ Шмидт М. (июнь 2008 г.). «Распространение синтетической биологии: вызов биобезопасности» (PDF) . Системы и синтетическая биология . 2 (1–2): 1–6. дои : 10.1007/s11693-008-9018-z . ПМК 2671588 . ПМИД 19003431 .
- ^ COSY: Общение по синтетической биологии
- ^ Кронбергер Н., Хольц П., Кербе В., Штрассер Э., Вагнер В. (декабрь 2009 г.). «Информация о синтетической биологии: из лаборатории через средства массовой информации до широкой публики» . Системы и синтетическая биология . 3 (1–4): 19–26. дои : 10.1007/s11693-009-9031-x . ПМЦ 2759424 . ПМИД 19816796 .
- ^ Церер А., Зейрингер А. (декабрь 2009 г.). «Картины синтетической биологии: рефлексивное обсуждение репрезентации синтетической биологии (SB) в немецкоязычных СМИ и экспертами SB» . Системы и синтетическая биология . 3 (1–4): 27–35. дои : 10.1007/s11693-009-9038-3 . ПМЦ 2759430 . ПМИД 19816797 .
- ^ Отчет IASB «Технические решения для биобезопасности в синтетической биологии». Архивировано 19 июля 2011 г., в Wayback Machine , Мюнхен, 2008 г.
- ^ Паренс Э., Джонстон Дж., Мозес Дж. Этические проблемы в синтетической биологии. 2009.
- ^ «Политика и глобальные дела в национальных академиях» . сайты.nationalacademies.org .
- ^ Президентская комиссия по изучению вопросов биоэтики, декабрь 2010 г. Часто задаваемые вопросы. Архивировано 24 декабря 2015 г. на Wayback Machine.
- ^ «Часто задаваемые вопросы по синтетической биологии | Президентская комиссия по изучению проблем биоэтики» . Архивировано из оригинала 24 декабря 2015 г. Проверено 11 октября 2014 г.
- ^ Jump up to: Перейти обратно: а б Эриксон Б., Сингх Р., Уинтерс П. (сентябрь 2011 г.). «Синтетическая биология: регулирование промышленного использования новых биотехнологий». Наука . 333 (6047): 1254–6. Бибкод : 2011Sci...333.1254E . дои : 10.1126/science.1211066 . ПМИД 21885775 . S2CID 1568198 .
- ^ Сэйлер К.М. (8 июня 2021 г.). Учебник по обороне: новые технологии (PDF) (отчет). Исследовательская служба Конгресса . Проверено 22 июля 2021 г.
- ^ Кэтрин Сюэ для журнала Harvard Magazine. Сентябрь – октябрь 2014 г. Новый зверинец синтетической биологии
- ↑ Йоджана Шарма для Scidev.net, 15 марта 2012 г. НПО призывают к международному регулированию синтетической биологии.
- ^ Новая синтетическая биология: кто выиграет? (2014-05-08), Ричард К. Левонтин , New York Review of Books
- ^ Национальные академии наук; Отдел исследований земной жизни; Совет по наукам о жизни; Совет по технологиям химических наук; Комитет по стратегиям выявления потенциальных уязвимостей биозащиты, создаваемых синтетической биологией (19 июня 2018 г.). Биозащита в эпоху синтетической биологии . Национальные академии наук, техники и медицины . дои : 10.17226/24890 . ISBN 9780309465182 . ПМИД 30629396 . S2CID 90767286 .
- ^ «Краткая информация о будущем: синтетическая биология и биоразнообразие» . Европейская комиссия . Сентябрь 2016. стр. 14–15 . Проверено 14 января 2019 г.
- ^ Заключительное мнение о синтетической биологии III: Риски для окружающей среды и биоразнообразия, связанные с синтетической биологией и приоритетами исследований в области синтетической биологии . Европейская комиссия. 04.04.2016. стр. 8, 27. ISBN. 9789279549731 . Проверено 14 января 2019 г.
{{cite book}}:|website=игнорируется ( помогите ) - ^ Эль Каруи М., Ойос-Флайт М., Флетчер Л. (2019). «Будущие тенденции в синтетической биологии – отчет» . Границы биоинженерии и биотехнологии . 7 : 175. дои : 10.3389/fbioe.2019.00175 . ПМК 6692427 . ПМИД 31448268 .
- ^ Бэйли С., Меткалф Х., Крук Б. (2012). «Синтетическая биология: обзор технологии, а также текущих и будущих потребностей нормативно-правовой базы Великобритании» (PDF) . Исполнительный директор Великобритании по охране труда и технике безопасности . Проверено 29 ноября 2018 г.
- ^ Пей Л., Бар-Ям С., Байерс-Корбин Дж., Касагранде Р., Эйхлер Ф., Лин А. и др. (2012). «Регуляторная база синтетической биологии». Синтетическая биология . John Wiley & Sons, Ltd., стр. 157–226. дои : 10.1002/9783527659296.ch5 . ISBN 9783527659296 .
- ^ Трамп Б.Д. (ноябрь 2017 г.). «Регулирование и управление синтетической биологией: уроки TAPIC для США, Европейского Союза и Сингапура» . Политика здравоохранения . 121 (11): 1139–1146. doi : 10.1016/j.healthpol.2017.07.010 . ПМИД 28807332 .
Библиография [ править ]
- Черч G, Реджис Э (2012). Как синтетическая биология заново изобретет природу и нас самих . Нью-Йорк, штат Нью-Йорк: Основные книги . ISBN 978-0465021758 .
- Синтетическая биология и биоразнообразие; Наука для экологической политики (PDF) . Краткое описание будущего 15. Подготовлено для Генерального директората Европейской комиссии по окружающей среде Отделом научных коммуникаций UWE, Бристоль (Отчет). Европейская комиссия. 2016.
- Вентер С (2013). Жизнь со скоростью света: двойная спираль и заря цифровой жизни . Нью-Йорк, штат Нью-Йорк: Penguin Books. ISBN 978-0670025404 .
Внешние ссылки [ править ]
- Искусственные патогены и неестественное биологическое оружие: будущая угроза синтетической биологии . Угрозы и соображения
- Книги по синтетической биологии, научно-популярные книги и учебники
- Вводное резюме синтетической биологии. Архивировано 2 апреля 2018 г. в Wayback Machine . Краткий обзор концепций, разработок и приложений синтетической биологии.
- Совместная обзорная статья по синтетической биологии
- Спорный стартап ДНК хочет позволить клиентам создавать существ (3 января 2015 г.), San Francisco Chronicle
- Он жив, но это жизнь: синтетическая биология и будущее творения (28 сентября 2016 г.), Всемирный фестиваль науки
