Бактерии
| Бактерии | |
|---|---|
 | |
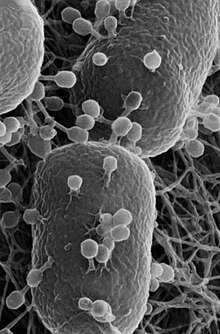
| Сканирующая электронная микрофотография палочек Escherichia coli . | |
| Научная классификация | |
| Domain: | Bacteria Woese et al. 1990 |
| Phyla | |
See § Phyla | |
| Synonyms | |
| |
Бактерии ( / b æ k ˈ t ɪər i ə / ; сг. :бактерия) — повсеместно распространённые, преимущественно свободноживущие организмы, часто состоящие из одной биологической клетки . обширную область прокариотических микроорганизмов . Они составляют несколько микрометров Бактерии, обычно длиной , были одними из первых форм жизни, появившихся на Земле , и присутствуют в большинстве ее сред обитания . Бактерии обитают в почве, воде, кислых горячих источниках , радиоактивных отходах и глубокой биосфере земной коры . Бактерии играют жизненно важную роль на многих стадиях круговорота питательных веществ , перерабатывая питательные вещества и фиксируя азот из атмосферы . Круговорот питательных веществ включает разложение мертвых тел ; Бактерии ответственны за стадию гниения в этом процессе. В биологических сообществах, окружающих гидротермальные источники и холодные выходы , экстремофильные бактерии обеспечивают питательные вещества, необходимые для поддержания жизни, преобразуя растворенные соединения, такие как сероводород и метан , в энергию. Бактерии также живут в мутуалистических , комменсальных и паразитических отношениях с растениями и животными. Большинство бактерий не охарактеризовано, и существует множество видов, которые невозможно охарактеризовать. выращенный в лаборатории. Изучение бактерий известно как бактериология , раздел микробиологии .
Like all animals, humans carry vast numbers (approximately 1013 to 1014) of bacteria.[2] Most are in the gut, though there are many on the skin. Most of the bacteria in and on the body are harmless or rendered so by the protective effects of the immune system, and many are beneficial,[3] particularly the ones in the gut. However, several species of bacteria are pathogenic and cause infectious diseases, including cholera, syphilis, anthrax, leprosy, tuberculosis, tetanus and bubonic plague. The most common fatal bacterial diseases are respiratory infections. Antibiotics are used to treat bacterial infections and are also used in farming, making antibiotic resistance a growing problem. Bacteria are important in sewage treatment and the breakdown of oil spills, the production of cheese and yogurt through fermentation, the recovery of gold, palladium, copper and other metals in the mining sector (biomining, bioleaching), as well as in biotechnology, and the manufacture of antibiotics and other chemicals.
Once regarded as plants constituting the class Schizomycetes ("fission fungi"), bacteria are now classified as prokaryotes. Unlike cells of animals and other eukaryotes, bacterial cells do not contain a nucleus and rarely harbour membrane-bound organelles. Although the term bacteria traditionally included all prokaryotes, the scientific classification changed after the discovery in the 1990s that prokaryotes consist of two very different groups of organisms that evolved from an ancient common ancestor. These evolutionary domains are called Bacteria and Archaea.[4]
Etymology

The word bacteria is the plural of the Neo-Latin bacterium, which is the Latinisation of the Ancient Greek βακτήριον (baktḗrion),[5] the diminutive of βακτηρία (baktēría), meaning "staff, cane",[6] because the first ones to be discovered were rod-shaped.[7][8]
Origin and early evolution

The ancestors of bacteria were unicellular microorganisms that were the first forms of life to appear on Earth, about 4 billion years ago.[10] For about 3 billion years, most organisms were microscopic, and bacteria and archaea were the dominant forms of life.[11][12][13] Although bacterial fossils exist, such as stromatolites, their lack of distinctive morphology prevents them from being used to examine the history of bacterial evolution, or to date the time of origin of a particular bacterial species. However, gene sequences can be used to reconstruct the bacterial phylogeny, and these studies indicate that bacteria diverged first from the archaeal/eukaryotic lineage.[14] The most recent common ancestor (MRCA) of bacteria and archaea was probably a hyperthermophile that lived about 2.5 billion–3.2 billion years ago.[15][16][17] The earliest life on land may have been bacteria some 3.22 billion years ago.[18]
Bacteria were also involved in the second great evolutionary divergence, that of the archaea and eukaryotes.[19][20] Here, eukaryotes resulted from the entering of ancient bacteria into endosymbiotic associations with the ancestors of eukaryotic cells, which were themselves possibly related to the Archaea.[21][22] This involved the engulfment by proto-eukaryotic cells of alphaproteobacterial symbionts to form either mitochondria or hydrogenosomes, which are still found in all known Eukarya (sometimes in highly reduced form, e.g. in ancient "amitochondrial" protozoa). Later, some eukaryotes that already contained mitochondria also engulfed cyanobacteria-like organisms, leading to the formation of chloroplasts in algae and plants. This is known as primary endosymbiosis.[23]
Habitat
Bacteria are ubiquitous, living in every possible habitat on the planet including soil, underwater, deep in Earth's crust and even such extreme environments as acidic hot springs and radioactive waste.[24][25] There are thought to be approximately 2×1030 bacteria on Earth,[26] forming a biomass that is only exceeded by plants.[27] They are abundant in lakes and oceans, in arctic ice, and geothermal springs[28] where they provide the nutrients needed to sustain life by converting dissolved compounds, such as hydrogen sulphide and methane, to energy.[29] They live on and in plants and animals. Most do not cause diseases, are beneficial to their environments, and are essential for life.[3][30] The soil is a rich source of bacteria and a few grams contain around a thousand million of them. They are all essential to soil ecology, breaking down toxic waste and recycling nutrients. They are even found in the atmosphere and one cubic metre of air holds around one hundred million bacterial cells. The oceans and seas harbour around 3 x 1026 bacteria which provide up to 50% of the oxygen humans breathe.[31] Only around 2% of bacterial species have been fully studied.[32]
| Habitat | Species | Reference |
|---|---|---|
| Cold (minus 15 °C Antarctica) | Cryptoendoliths | [33] |
| Hot (70–100 °C geysers) | Thermus aquaticus | [32] |
| Radiation, 5MRad | Deinococcus radiodurans | [33] |
| Saline, 47% salt (Dead Sea, Great Salt Lake) | several species | [32][33] |
| Acid pH 3 | several species | [24] |
| Alkaline pH 12.8 | betaproteobacteria | [33] |
| Space (6 years on a NASA satellite) | Bacillus subtilis | [33] |
| 3.2 km underground | several species | [33] |
| High pressure (Mariana Trench – 1200 atm) | Moritella, Shewanella and others | [33] |
Morphology
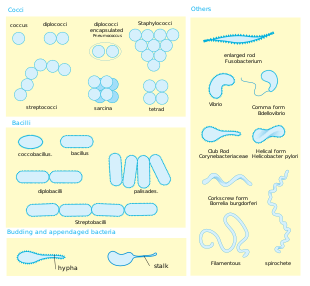
Size. Bacteria display a wide diversity of shapes and sizes. Bacterial cells are about one-tenth the size of eukaryotic cells and are typically 0.5–5.0 micrometres in length. However, a few species are visible to the unaided eye—for example, Thiomargarita namibiensis is up to half a millimetre long,[34] Epulopiscium fishelsoni reaches 0.7 mm,[35] and Thiomargarita magnifica can reach even 2 cm in length, which is 50 times larger than other known bacteria.[36][37] Among the smallest bacteria are members of the genus Mycoplasma, which measure only 0.3 micrometres, as small as the largest viruses.[38] Some bacteria may be even smaller, but these ultramicrobacteria are not well-studied.[39]
Shape. Most bacterial species are either spherical, called cocci (singular coccus, from Greek kókkos, grain, seed), or rod-shaped, called bacilli (sing. bacillus, from Latin baculus, stick).[40] Some bacteria, called vibrio, are shaped like slightly curved rods or comma-shaped; others can be spiral-shaped, called spirilla, or tightly coiled, called spirochaetes. A small number of other unusual shapes have been described, such as star-shaped bacteria.[41] This wide variety of shapes is determined by the bacterial cell wall and cytoskeleton and is important because it can influence the ability of bacteria to acquire nutrients, attach to surfaces, swim through liquids and escape predators.[42][43]

Multicellularity. Most bacterial species exist as single cells; others associate in characteristic patterns: Neisseria forms diploids (pairs), streptococci form chains, and staphylococci group together in "bunch of grapes" clusters. Bacteria can also group to form larger multicellular structures, such as the elongated filaments of Actinomycetota species, the aggregates of Myxobacteria species, and the complex hyphae of Streptomyces species.[45] These multicellular structures are often only seen in certain conditions. For example, when starved of amino acids, myxobacteria detect surrounding cells in a process known as quorum sensing, migrate towards each other, and aggregate to form fruiting bodies up to 500 micrometres long and containing approximately 100,000 bacterial cells.[46] In these fruiting bodies, the bacteria perform separate tasks; for example, about one in ten cells migrate to the top of a fruiting body and differentiate into a specialised dormant state called a myxospore, which is more resistant to drying and other adverse environmental conditions.[47]
Biofilms. Bacteria often attach to surfaces and form dense aggregations called biofilms[48] and larger formations known as microbial mats.[49] These biofilms and mats can range from a few micrometres in thickness to up to half a metre in depth, and may contain multiple species of bacteria, protists and archaea. Bacteria living in biofilms display a complex arrangement of cells and extracellular components, forming secondary structures, such as microcolonies, through which there are networks of channels to enable better diffusion of nutrients.[50][51] In natural environments, such as soil or the surfaces of plants, the majority of bacteria are bound to surfaces in biofilms.[52] Biofilms are also important in medicine, as these structures are often present during chronic bacterial infections or in infections of implanted medical devices, and bacteria protected within biofilms are much harder to kill than individual isolated bacteria.[53]
Cellular structure

Intracellular structures
The bacterial cell is surrounded by a cell membrane, which is made primarily of phospholipids. This membrane encloses the contents of the cell and acts as a barrier to hold nutrients, proteins and other essential components of the cytoplasm within the cell.[54] Unlike eukaryotic cells, bacteria usually lack large membrane-bound structures in their cytoplasm such as a nucleus, mitochondria, chloroplasts and the other organelles present in eukaryotic cells.[55] However, some bacteria have protein-bound organelles in the cytoplasm which compartmentalise aspects of bacterial metabolism,[56][57] such as the carboxysome.[58] Additionally, bacteria have a multi-component cytoskeleton to control the localisation of proteins and nucleic acids within the cell, and to manage the process of cell division.[59][60][61]
Many important biochemical reactions, such as energy generation, occur due to concentration gradients across membranes, creating a potential difference analogous to a battery. The general lack of internal membranes in bacteria means these reactions, such as electron transport, occur across the cell membrane between the cytoplasm and the outside of the cell or periplasm.[62] However, in many photosynthetic bacteria, the plasma membrane is highly folded and fills most of the cell with layers of light-gathering membrane.[63] These light-gathering complexes may even form lipid-enclosed structures called chlorosomes in green sulfur bacteria.[64]
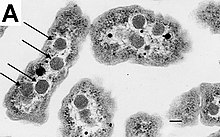
Bacteria do not have a membrane-bound nucleus, and their genetic material is typically a single circular bacterial chromosome of DNA located in the cytoplasm in an irregularly shaped body called the nucleoid.[65] The nucleoid contains the chromosome with its associated proteins and RNA. Like all other organisms, bacteria contain ribosomes for the production of proteins, but the structure of the bacterial ribosome is different from that of eukaryotes and archaea.[66]
Some bacteria produce intracellular nutrient storage granules, such as glycogen,[67] polyphosphate,[68] sulfur[69] or polyhydroxyalkanoates.[70] Bacteria such as the photosynthetic cyanobacteria, produce internal gas vacuoles, which they use to regulate their buoyancy, allowing them to move up or down into water layers with different light intensities and nutrient levels.[71]
Extracellular structures
Around the outside of the cell membrane is the cell wall. Bacterial cell walls are made of peptidoglycan (also called murein), which is made from polysaccharide chains cross-linked by peptides containing D-amino acids.[72] Bacterial cell walls are different from the cell walls of plants and fungi, which are made of cellulose and chitin, respectively.[73] The cell wall of bacteria is also distinct from that of achaea, which do not contain peptidoglycan. The cell wall is essential to the survival of many bacteria, and the antibiotic penicillin (produced by a fungus called Penicillium) is able to kill bacteria by inhibiting a step in the synthesis of peptidoglycan.[73]
There are broadly speaking two different types of cell wall in bacteria, that classify bacteria into Gram-positive bacteria and Gram-negative bacteria. The names originate from the reaction of cells to the Gram stain, a long-standing test for the classification of bacterial species.[74]
Gram-positive bacteria possess a thick cell wall containing many layers of peptidoglycan and teichoic acids. In contrast, Gram-negative bacteria have a relatively thin cell wall consisting of a few layers of peptidoglycan surrounded by a second lipid membrane containing lipopolysaccharides and lipoproteins. Most bacteria have the Gram-negative cell wall, and only members of the Bacillota group and actinomycetota (previously known as the low G+C and high G+C Gram-positive bacteria, respectively) have the alternative Gram-positive arrangement.[75] These differences in structure can produce differences in antibiotic susceptibility; for instance, vancomycin can kill only Gram-positive bacteria and is ineffective against Gram-negative pathogens, such as Haemophilus influenzae or Pseudomonas aeruginosa.[76] Some bacteria have cell wall structures that are neither classically Gram-positive or Gram-negative. This includes clinically important bacteria such as mycobacteria which have a thick peptidoglycan cell wall like a Gram-positive bacterium, but also a second outer layer of lipids.[77]
In many bacteria, an S-layer of rigidly arrayed protein molecules covers the outside of the cell.[78] This layer provides chemical and physical protection for the cell surface and can act as a macromolecular diffusion barrier. S-layers have diverse functions and are known to act as virulence factors in Campylobacter species and contain surface enzymes in Bacillus stearothermophilus.[79][80]
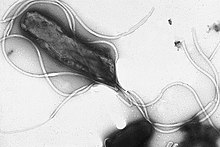
Flagella are rigid protein structures, about 20 nanometres in diameter and up to 20 micrometres in length, that are used for motility. Flagella are driven by the energy released by the transfer of ions down an electrochemical gradient across the cell membrane.[81]
Fimbriae (sometimes called "attachment pili") are fine filaments of protein, usually 2–10 nanometres in diameter and up to several micrometres in length. They are distributed over the surface of the cell, and resemble fine hairs when seen under the electron microscope.[82] Fimbriae are believed to be involved in attachment to solid surfaces or to other cells, and are essential for the virulence of some bacterial pathogens.[83] Pili (sing. pilus) are cellular appendages, slightly larger than fimbriae, that can transfer genetic material between bacterial cells in a process called conjugation where they are called conjugation pili or sex pili (see bacterial genetics, below).[84] They can also generate movement where they are called type IV pili.[85]
Glycocalyx is produced by many bacteria to surround their cells,[86] and varies in structural complexity: ranging from a disorganised slime layer of extracellular polymeric substances to a highly structured capsule. These structures can protect cells from engulfment by eukaryotic cells such as macrophages (part of the human immune system).[87] They can also act as antigens and be involved in cell recognition, as well as aiding attachment to surfaces and the formation of biofilms.[88]
The assembly of these extracellular structures is dependent on bacterial secretion systems. These transfer proteins from the cytoplasm into the periplasm or into the environment around the cell. Many types of secretion systems are known and these structures are often essential for the virulence of pathogens, so are intensively studied.[88]
Endospores
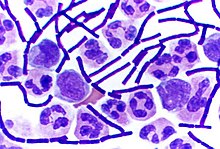
Some genera of Gram-positive bacteria, such as Bacillus, Clostridium, Sporohalobacter, Anaerobacter, and Heliobacterium, can form highly resistant, dormant structures called endospores.[90] Endospores develop within the cytoplasm of the cell; generally, a single endospore develops in each cell.[91] Each endospore contains a core of DNA and ribosomes surrounded by a cortex layer and protected by a multilayer rigid coat composed of peptidoglycan and a variety of proteins.[91]
Endospores show no detectable metabolism and can survive extreme physical and chemical stresses, such as high levels of UV light, gamma radiation, detergents, disinfectants, heat, freezing, pressure, and desiccation.[92] In this dormant state, these organisms may remain viable for millions of years.[93][94][95] Endospores even allow bacteria to survive exposure to the vacuum and radiation of outer space, leading to the possibility that bacteria could be distributed throughout the Universe by space dust, meteoroids, asteroids, comets, planetoids, or directed panspermia.[96][97]
Endospore-forming bacteria can cause disease; for example, anthrax can be contracted by the inhalation of Bacillus anthracis endospores, and contamination of deep puncture wounds with Clostridium tetani endospores causes tetanus, which, like botulism, is caused by a toxin released by the bacteria that grow from the spores.[98] Clostridioides difficile infection, a common problem in healthcare settings, is caused by spore-forming bacteria.[99]
Metabolism
Bacteria exhibit an extremely wide variety of metabolic types.[100] The distribution of metabolic traits within a group of bacteria has traditionally been used to define their taxonomy, but these traits often do not correspond with modern genetic classifications.[101] Bacterial metabolism is classified into nutritional groups on the basis of three major criteria: the source of energy, the electron donors used, and the source of carbon used for growth.[102]
Phototrophic bacteria derive energy from light using photosynthesis, while chemotrophic bacteria breaking down chemical compounds through oxidation,[103] driving metabolism by transferring electrons from a given electron donor to a terminal electron acceptor in a redox reaction. Chemotrophs are further divided by the types of compounds they use to transfer electrons. Bacteria that derive electrons from inorganic compounds such as hydrogen, carbon monoxide, or ammonia are called lithotrophs, while those that use organic compounds are called organotrophs.[103] Still, more specifically, aerobic organisms use oxygen as the terminal electron acceptor, while anaerobic organisms use other compounds such as nitrate, sulfate, or carbon dioxide.[103]
Many bacteria, called heterotrophs, derive their carbon from other organic carbon. Others, such as cyanobacteria and some purple bacteria, are autotrophic, meaning they obtain cellular carbon by fixing carbon dioxide.[104] In unusual circumstances, the gas methane can be used by methanotrophic bacteria as both a source of electrons and a substrate for carbon anabolism.[105]
| Nutritional type | Source of energy | Source of carbon | Examples |
|---|---|---|---|
| Phototrophs | Sunlight | Organic compounds (photoheterotrophs) or carbon fixation (photoautotrophs) | Cyanobacteria, Green sulfur bacteria, Chloroflexota, or Purple bacteria |
| Lithotrophs | Inorganic compounds | Organic compounds (lithoheterotrophs) or carbon fixation (lithoautotrophs) | Thermodesulfobacteriota, Hydrogenophilaceae, or Nitrospirota |
| Organotrophs | Organic compounds | Organic compounds (chemoheterotrophs) or carbon fixation (chemoautotrophs) | Bacillus, Clostridium, or Enterobacteriaceae |
In many ways, bacterial metabolism provides traits that are useful for ecological stability and for human society. For example, diazotrophs have the ability to fix nitrogen gas using the enzyme nitrogenase.[106] This trait, which can be found in bacteria of most metabolic types listed above,[107] leads to the ecologically important processes of denitrification, sulfate reduction, and acetogenesis, respectively.[108] Bacterial metabolic processes are important drivers in biological responses to pollution; for example, sulfate-reducing bacteria are largely responsible for the production of the highly toxic forms of mercury (methyl- and dimethylmercury) in the environment.[109] Nonrespiratory anaerobes use fermentation to generate energy and reducing power, secreting metabolic by-products (such as ethanol in brewing) as waste. Facultative anaerobes can switch between fermentation and different terminal electron acceptors depending on the environmental conditions in which they find themselves.[110]
Growth and reproduction



Unlike in multicellular organisms, increases in cell size (cell growth) and reproduction by cell division are tightly linked in unicellular organisms. Bacteria grow to a fixed size and then reproduce through binary fission, a form of asexual reproduction.[112] Under optimal conditions, bacteria can grow and divide extremely rapidly, and some bacterial populations can double as quickly as every 17 minutes.[113] In cell division, two identical clone daughter cells are produced. Some bacteria, while still reproducing asexually, form more complex reproductive structures that help disperse the newly formed daughter cells. Examples include fruiting body formation by myxobacteria and aerial hyphae formation by Streptomyces species, or budding. Budding involves a cell forming a protrusion that breaks away and produces a daughter cell.[114]
In the laboratory, bacteria are usually grown using solid or liquid media.[115] Solid growth media, such as agar plates, are used to isolate pure cultures of a bacterial strain. However, liquid growth media are used when the measurement of growth or large volumes of cells are required. Growth in stirred liquid media occurs as an even cell suspension, making the cultures easy to divide and transfer, although isolating single bacteria from liquid media is difficult. The use of selective media (media with specific nutrients added or deficient, or with antibiotics added) can help identify specific organisms.[116]
Most laboratory techniques for growing bacteria use high levels of nutrients to produce large amounts of cells cheaply and quickly.[115] However, in natural environments, nutrients are limited, meaning that bacteria cannot continue to reproduce indefinitely. This nutrient limitation has led the evolution of different growth strategies (see r/K selection theory). Some organisms can grow extremely rapidly when nutrients become available, such as the formation of algal and cyanobacterial blooms that often occur in lakes during the summer.[117] Other organisms have adaptations to harsh environments, such as the production of multiple antibiotics by Streptomyces that inhibit the growth of competing microorganisms.[118] In nature, many organisms live in communities (e.g., biofilms) that may allow for increased supply of nutrients and protection from environmental stresses.[52] These relationships can be essential for growth of a particular organism or group of organisms (syntrophy).[119]
Bacterial growth follows four phases. When a population of bacteria first enter a high-nutrient environment that allows growth, the cells need to adapt to their new environment. The first phase of growth is the lag phase, a period of slow growth when the cells are adapting to the high-nutrient environment and preparing for fast growth. The lag phase has high biosynthesis rates, as proteins necessary for rapid growth are produced.[120][121] The second phase of growth is the logarithmic phase, also known as the exponential phase. The log phase is marked by rapid exponential growth. The rate at which cells grow during this phase is known as the growth rate (k), and the time it takes the cells to double is known as the generation time (g). During log phase, nutrients are metabolised at maximum speed until one of the nutrients is depleted and starts limiting growth. The third phase of growth is the stationary phase and is caused by depleted nutrients. The cells reduce their metabolic activity and consume non-essential cellular proteins. The stationary phase is a transition from rapid growth to a stress response state and there is increased expression of genes involved in DNA repair, antioxidant metabolism and nutrient transport.[122] The final phase is the death phase where the bacteria run out of nutrients and die.[123]
Genetics
Most bacteria have a single circular chromosome that can range in size from only 160,000 base pairs in the endosymbiotic bacteria Carsonella ruddii,[125] to 12,200,000 base pairs (12.2 Mbp) in the soil-dwelling bacteria Sorangium cellulosum.[126] There are many exceptions to this; for example, some Streptomyces and Borrelia species contain a single linear chromosome,[127][128] while some Vibrio species contain more than one chromosome.[129] Some bacteria contain plasmids, small extra-chromosomal molecules of DNA that may contain genes for various useful functions such as antibiotic resistance, metabolic capabilities, or various virulence factors.[130]
Bacteria genomes usually encode a few hundred to a few thousand genes. The genes in bacterial genomes are usually a single continuous stretch of DNA. Although several different types of introns do exist in bacteria, these are much rarer than in eukaryotes.[131]
Bacteria, as asexual organisms, inherit an identical copy of the parent's genome and are clonal. However, all bacteria can evolve by selection on changes to their genetic material DNA caused by genetic recombination or mutations. Mutations arise from errors made during the replication of DNA or from exposure to mutagens. Mutation rates vary widely among different species of bacteria and even among different clones of a single species of bacteria.[132] Genetic changes in bacterial genomes emerge from either random mutation during replication or "stress-directed mutation", where genes involved in a particular growth-limiting process have an increased mutation rate.[133]
Some bacteria transfer genetic material between cells. This can occur in three main ways. First, bacteria can take up exogenous DNA from their environment in a process called transformation.[134] Many bacteria can naturally take up DNA from the environment, while others must be chemically altered in order to induce them to take up DNA.[135] The development of competence in nature is usually associated with stressful environmental conditions and seems to be an adaptation for facilitating repair of DNA damage in recipient cells.[136] Second, bacteriophages can integrate into the bacterial chromosome, introducing foreign DNA in a process known as transduction. Many types of bacteriophage exist; some infect and lyse their host bacteria, while others insert into the bacterial chromosome.[137] Bacteria resist phage infection through restriction modification systems that degrade foreign DNA[138] and a system that uses CRISPR sequences to retain fragments of the genomes of phage that the bacteria have come into contact with in the past, which allows them to block virus replication through a form of RNA interference.[139][140] Third, bacteria can transfer genetic material through direct cell contact via conjugation.[141]
In ordinary circumstances, transduction, conjugation, and transformation involve transfer of DNA between individual bacteria of the same species, but occasionally transfer may occur between individuals of different bacterial species, and this may have significant consequences, such as the transfer of antibiotic resistance.[142][143] In such cases, gene acquisition from other bacteria or the environment is called horizontal gene transfer and may be common under natural conditions.[144]
Behaviour
Movement

Many bacteria are motile (able to move themselves) and do so using a variety of mechanisms. The best studied of these are flagella, long filaments that are turned by a motor at the base to generate propeller-like movement.[145] The bacterial flagellum is made of about 20 proteins, with approximately another 30 proteins required for its regulation and assembly.[145] The flagellum is a rotating structure driven by a reversible motor at the base that uses the electrochemical gradient across the membrane for power.[146]

Bacteria can use flagella in different ways to generate different kinds of movement. Many bacteria (such as E. coli) have two distinct modes of movement: forward movement (swimming) and tumbling. The tumbling allows them to reorient and makes their movement a three-dimensional random walk.[147] Bacterial species differ in the number and arrangement of flagella on their surface; some have a single flagellum (monotrichous), a flagellum at each end (amphitrichous), clusters of flagella at the poles of the cell (lophotrichous), while others have flagella distributed over the entire surface of the cell (peritrichous). The flagella of a group of bacteria, the spirochaetes, are found between two membranes in the periplasmic space. They have a distinctive helical body that twists about as it moves.[145]
Two other types of bacterial motion are called twitching motility that relies on a structure called the type IV pilus,[148] and gliding motility, that uses other mechanisms. In twitching motility, the rod-like pilus extends out from the cell, binds some substrate, and then retracts, pulling the cell forward.[149]
Motile bacteria are attracted or repelled by certain stimuli in behaviours called taxes: these include chemotaxis, phototaxis, energy taxis, and magnetotaxis.[150][151][152] In one peculiar group, the myxobacteria, individual bacteria move together to form waves of cells that then differentiate to form fruiting bodies containing spores.[47] The myxobacteria move only when on solid surfaces, unlike E. coli, which is motile in liquid or solid media.[153]
Several Listeria and Shigella species move inside host cells by usurping the cytoskeleton, which is normally used to move organelles inside the cell. By promoting actin polymerisation at one pole of their cells, they can form a kind of tail that pushes them through the host cell's cytoplasm.[154]
Communication
A few bacteria have chemical systems that generate light. This bioluminescence often occurs in bacteria that live in association with fish, and the light probably serves to attract fish or other large animals.[155]
Bacteria often function as multicellular aggregates known as biofilms, exchanging a variety of molecular signals for intercell communication and engaging in coordinated multicellular behaviour.[156][157]
The communal benefits of multicellular cooperation include a cellular division of labour, accessing resources that cannot effectively be used by single cells, collectively defending against antagonists, and optimising population survival by differentiating into distinct cell types.[156] For example, bacteria in biofilms can have more than five hundred times increased resistance to antibacterial agents than individual "planktonic" bacteria of the same species.[157]
One type of intercellular communication by a molecular signal is called quorum sensing, which serves the purpose of determining whether the local population density is sufficient to support investment in processes that are only successful if large numbers of similar organisms behave similarly, such as excreting digestive enzymes or emitting light.[158][159] Quorum sensing enables bacteria to coordinate gene expression and to produce, release, and detect autoinducers or pheromones that accumulate with the growth in cell population.[160]
Classification and identification


Classification seeks to describe the diversity of bacterial species by naming and grouping organisms based on similarities. Bacteria can be classified on the basis of cell structure, cellular metabolism or on differences in cell components, such as DNA, fatty acids, pigments, antigens and quinones.[116] While these schemes allowed the identification and classification of bacterial strains, it was unclear whether these differences represented variation between distinct species or between strains of the same species. This uncertainty was due to the lack of distinctive structures in most bacteria, as well as lateral gene transfer between unrelated species.[162] Due to lateral gene transfer, some closely related bacteria can have very different morphologies and metabolisms. To overcome this uncertainty, modern bacterial classification emphasises molecular systematics, using genetic techniques such as guanine cytosine ratio determination, genome-genome hybridisation, as well as sequencing genes that have not undergone extensive lateral gene transfer, such as the rRNA gene.[163] Classification of bacteria is determined by publication in the International Journal of Systematic Bacteriology,[164] and Bergey's Manual of Systematic Bacteriology.[165] The International Committee on Systematic Bacteriology (ICSB) maintains international rules for the naming of bacteria and taxonomic categories and for the ranking of them in the International Code of Nomenclature of Bacteria.[166]
Historically, bacteria were considered a part of the Plantae, the Plant kingdom, and were called "Schizomycetes" (fission-fungi).[167] For this reason, collective bacteria and other microorganisms in a host are often called "flora".[168]The term "bacteria" was traditionally applied to all microscopic, single-cell prokaryotes. However, molecular systematics showed prokaryotic life to consist of two separate domains, originally called Eubacteria and Archaebacteria, but now called Bacteria and Archaea that evolved independently from an ancient common ancestor.[4] The archaea and eukaryotes are more closely related to each other than either is to the bacteria. These two domains, along with Eukarya, are the basis of the three-domain system, which is currently the most widely used classification system in microbiology.[169] However, due to the relatively recent introduction of molecular systematics and a rapid increase in the number of genome sequences that are available, bacterial classification remains a changing and expanding field.[170][171] For example, Cavalier-Smith argued that the Archaea and Eukaryotes evolved from Gram-positive bacteria.[172]
The identification of bacteria in the laboratory is particularly relevant in medicine, where the correct treatment is determined by the bacterial species causing an infection. Consequently, the need to identify human pathogens was a major impetus for the development of techniques to identify bacteria.[173]
The Gram stain, developed in 1884 by Hans Christian Gram, characterises bacteria based on the structural characteristics of their cell walls.[174][74] The thick layers of peptidoglycan in the "Gram-positive" cell wall stain purple, while the thin "Gram-negative" cell wall appears pink.[174] By combining morphology and Gram-staining, most bacteria can be classified as belonging to one of four groups (Gram-positive cocci, Gram-positive bacilli, Gram-negative cocci and Gram-negative bacilli). Some organisms are best identified by stains other than the Gram stain, particularly mycobacteria or Nocardia, which show acid fastness on Ziehl–Neelsen or similar stains.[175] Other organisms may need to be identified by their growth in special media, or by other techniques, such as serology.[176]
Culture techniques are designed to promote the growth and identify particular bacteria while restricting the growth of the other bacteria in the sample.[177] Often these techniques are designed for specific specimens; for example, a sputum sample will be treated to identify organisms that cause pneumonia, while stool specimens are cultured on selective media to identify organisms that cause diarrhea while preventing growth of non-pathogenic bacteria. Specimens that are normally sterile, such as blood, urine or spinal fluid, are cultured under conditions designed to grow all possible organisms.[116][178] Once a pathogenic organism has been isolated, it can be further characterised by its morphology, growth patterns (such as aerobic or anaerobic growth), patterns of hemolysis, and staining.[179]
As with bacterial classification, identification of bacteria is increasingly using molecular methods,[180] and mass spectroscopy.[181] Most bacteria have not been characterised and there are many species that cannot be grown in the laboratory.[182] Diagnostics using DNA-based tools, such as polymerase chain reaction, are increasingly popular due to their specificity and speed, compared to culture-based methods.[183] These methods also allow the detection and identification of "viable but nonculturable" cells that are metabolically active but non-dividing.[184] However, even using these improved methods, the total number of bacterial species is not known and cannot even be estimated with any certainty. Following present classification, there are a little less than 9,300 known species of prokaryotes, which includes bacteria and archaea;[185] but attempts to estimate the true number of bacterial diversity have ranged from 107 to 109 total species—and even these diverse estimates may be off by many orders of magnitude.[186][187]
Phyla
The following phyla have been validly published according to the Bacteriological Code:[188]
- Acidobacteriota
- Actinomycetota
- Aquificota
- Armatimonadota
- Atribacterota
- Bacillota
- Bacteroidota
- Balneolota
- Bdellovibrionota
- Caldisericota
- Calditrichota
- Campylobacterota
- Chlamydiota
- Chlorobiota
- Chloroflexota
- Chrysiogenota
- Coprothermobacterota
- Deferribacterota
- Deinococcota
- Dictyoglomota
- Elusimicrobiota
- Fibrobacterota
- Fusobacteriota
- Gemmatimonadota
- Ignavibacteriota
- Lentisphaerota
- Mycoplasmatota
- Myxococcota
- Nitrospinota
- Nitrospirota
- Planctomycetota
- Pseudomonadota
- Rhodothermota
- Spirochaetota
- Synergistota
- Thermodesulfobacteriota
- Thermomicrobiota
- Thermotogota
- Verrucomicrobiota
Interactions with other organisms
Despite their apparent simplicity, bacteria can form complex associations with other organisms. These symbiotic associations can be divided into parasitism, mutualism and commensalism.[190]
Commensals
The word "commensalism" is derived from the word "commensal", meaning "eating at the same table"[191] and all plants and animals are colonised by commensal bacteria. In humans and other animals, millions of them live on the skin, the airways, the gut and other orifices.[192][193]Referred to as "normal flora",[194] or "commensals",[195] these bacteria usually cause no harm but may occasionally invade other sites of the body and cause infection. Escherichia coli is a commensal in the human gut but can cause urinary tract infections.[196] Similarly, streptococci, which are part of the normal flora of the human mouth, can cause heart disease.[197]
Predators
Some species of bacteria kill and then consume other microorganisms; these species are called predatory bacteria.[198] These include organisms such as Myxococcus xanthus, which forms swarms of cells that kill and digest any bacteria they encounter.[199] Other bacterial predators either attach to their prey in order to digest them and absorb nutrients or invade another cell and multiply inside the cytosol.[200] These predatory bacteria are thought to have evolved from saprophages that consumed dead microorganisms, through adaptations that allowed them to entrap and kill other organisms.[201]
Mutualists
Certain bacteria form close spatial associations that are essential for their survival. One such mutualistic association, called interspecies hydrogen transfer, occurs between clusters of anaerobic bacteria that consume organic acids, such as butyric acid or propionic acid, and produce hydrogen, and methanogenic archaea that consume hydrogen.[202] The bacteria in this association are unable to consume the organic acids as this reaction produces hydrogen that accumulates in their surroundings. Only the intimate association with the hydrogen-consuming archaea keeps the hydrogen concentration low enough to allow the bacteria to grow.[203]
In soil, microorganisms that reside in the rhizosphere (a zone that includes the root surface and the soil that adheres to the root after gentle shaking) carry out nitrogen fixation, converting nitrogen gas to nitrogenous compounds.[204] This serves to provide an easily absorbable form of nitrogen for many plants, which cannot fix nitrogen themselves. Many other bacteria are found as symbionts in humans and other organisms. For example, the presence of over 1,000 bacterial species in the normal human gut flora of the intestines can contribute to gut immunity, synthesise vitamins, such as folic acid, vitamin K and biotin, convert sugars to lactic acid (see Lactobacillus), as well as fermenting complex undigestible carbohydrates.[205][206][207] The presence of this gut flora also inhibits the growth of potentially pathogenic bacteria (usually through competitive exclusion) and these beneficial bacteria are consequently sold as probiotic dietary supplements.[208]
Nearly all animal life is dependent on bacteria for survival as only bacteria and some archaea possess the genes and enzymes necessary to synthesise vitamin B12, also known as cobalamin, and provide it through the food chain. Vitamin B12 is a water-soluble vitamin that is involved in the metabolism of every cell of the human body. It is a cofactor in DNA synthesis and in both fatty acid and amino acid metabolism. It is particularly important in the normal functioning of the nervous system via its role in the synthesis of myelin.[209]
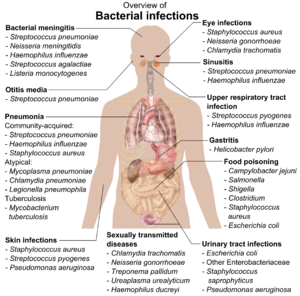
Pathogens

The body is continually exposed to many species of bacteria, including beneficial commensals, which grow on the skin and mucous membranes, and saprophytes, which grow mainly in the soil and in decaying matter. The blood and tissue fluids contain nutrients sufficient to sustain the growth of many bacteria. The body has defence mechanisms that enable it to resist microbial invasion of its tissues and give it a natural immunity or innate resistance against many microorganisms.[210] Unlike some viruses, bacteria evolve relatively slowly so many bacterial diseases also occur in other animals.[211]
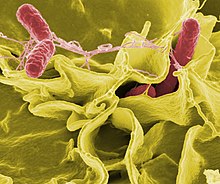
If bacteria form a parasitic association with other organisms, they are classed as pathogens.[212] Pathogenic bacteria are a major cause of human death and disease and cause infections such as tetanus (caused by Clostridium tetani), typhoid fever, diphtheria, syphilis, cholera, foodborne illness, leprosy (caused by Mycobacterium leprae) and tuberculosis (caused by Mycobacterium tuberculosis).[213] A pathogenic cause for a known medical disease may only be discovered many years later, as was the case with Helicobacter pylori and peptic ulcer disease.[214] Bacterial diseases are also important in agriculture, and bacteria cause leaf spot, fire blight and wilts in plants, as well as Johne's disease, mastitis, salmonella and anthrax in farm animals.[215]

Each species of pathogen has a characteristic spectrum of interactions with its human hosts. Some organisms, such as Staphylococcus or Streptococcus, can cause skin infections, pneumonia, meningitis and sepsis, a systemic inflammatory response producing shock, massive vasodilation and death.[216] Yet these organisms are also part of the normal human flora and usually exist on the skin or in the nose without causing any disease at all. Other organisms invariably cause disease in humans, such as Rickettsia, which are obligate intracellular parasites able to grow and reproduce only within the cells of other organisms. One species of Rickettsia causes typhus, while another causes Rocky Mountain spotted fever. Chlamydia, another phylum of obligate intracellular parasites, contains species that can cause pneumonia or urinary tract infection and may be involved in coronary heart disease.[217] Some species, such as Pseudomonas aeruginosa, Burkholderia cenocepacia, and Mycobacterium avium, are opportunistic pathogens and cause disease mainly in people who are immunosuppressed or have cystic fibrosis.[218][219] Some bacteria produce toxins, which cause diseases.[220] These are endotoxins, which come from broken bacterial cells, and exotoxins, which are produced by bacteria and released into the environment.[221] The bacterium Clostridium botulinum for example, produces a powerful exotoxin that cause respiratory paralysis, and Salmonellae produce an endotoxin that causes gastroenteritis.[221] Some exotoxins can be converted to toxoids, which are used as vaccines to prevent the disease.[222]
Bacterial infections may be treated with antibiotics, which are classified as bacteriocidal if they kill bacteria or bacteriostatic if they just prevent bacterial growth. There are many types of antibiotics, and each class inhibits a process that is different in the pathogen from that found in the host. An example of how antibiotics produce selective toxicity are chloramphenicol and puromycin, which inhibit the bacterial ribosome, but not the structurally different eukaryotic ribosome.[223] Antibiotics are used both in treating human disease and in intensive farming to promote animal growth, where they may be contributing to the rapid development of antibiotic resistance in bacterial populations.[224] Infections can be prevented by antiseptic measures such as sterilising the skin prior to piercing it with the needle of a syringe, and by proper care of indwelling catheters. Surgical and dental instruments are also sterilised to prevent contamination by bacteria. Disinfectants such as bleach are used to kill bacteria or other pathogens on surfaces to prevent contamination and further reduce the risk of infection.[225]
Significance in technology and industry
Bacteria, often lactic acid bacteria, such as Lactobacillus species and Lactococcus species, in combination with yeasts and moulds, have been used for thousands of years in the preparation of fermented foods, such as cheese, pickles, soy sauce, sauerkraut, vinegar, wine, and yogurt.[226][227]
The ability of bacteria to degrade a variety of organic compounds is remarkable and has been used in waste processing and bioremediation. Bacteria capable of digesting the hydrocarbons in petroleum are often used to clean up oil spills.[228] Fertiliser was added to some of the beaches in Prince William Sound in an attempt to promote the growth of these naturally occurring bacteria after the 1989 Exxon Valdez oil spill. These efforts were effective on beaches that were not too thickly covered in oil. Bacteria are also used for the bioremediation of industrial toxic wastes.[229] In the chemical industry, bacteria are most important in the production of enantiomerically pure chemicals for use as pharmaceuticals or agrichemicals.[230]
Bacteria can also be used in place of pesticides in biological pest control. This commonly involves Bacillus thuringiensis (also called BT), a Gram-positive, soil-dwelling bacterium. Subspecies of this bacteria are used as Lepidopteran-specific insecticides under trade names such as Dipel and Thuricide.[231] Because of their specificity, these pesticides are regarded as environmentally friendly, with little or no effect on humans, wildlife, pollinators, and most other beneficial insects.[232][233]
Because of their ability to quickly grow and the relative ease with which they can be manipulated, bacteria are the workhorses for the fields of molecular biology, genetics, and biochemistry. By making mutations in bacterial DNA and examining the resulting phenotypes, scientists can determine the function of genes, enzymes, and metabolic pathways in bacteria, then apply this knowledge to more complex organisms.[234] This aim of understanding the biochemistry of a cell reaches its most complex expression in the synthesis of huge amounts of enzyme kinetic and gene expression data into mathematical models of entire organisms. This is achievable in some well-studied bacteria, with models of Escherichia coli metabolism now being produced and tested.[235][236] This understanding of bacterial metabolism and genetics allows the use of biotechnology to bioengineer bacteria for the production of therapeutic proteins, such as insulin, growth factors, or antibodies.[237][238]
Из-за их важности для исследований в целом образцы бактериальных штаммов изолируются и сохраняются в центрах биологических ресурсов . Это обеспечивает доступность штамма для ученых во всем мире. [239]
История бактериологии

Бактерии впервые наблюдал голландский микроскопист Антони ван Левенгук в 1676 году с помощью однолинзового микроскопа собственной конструкции. Затем он опубликовал свои наблюдения в серии писем Лондонскому королевскому обществу . [240] Бактерии были самым замечательным микроскопическим открытием Левенгука. Их размер был на пределе того, что могли разрешить его простые линзы, и в один из самых поразительных перерывов в истории науки никто больше не видел их больше столетия. [241] Его наблюдения также включали простейших, которых он назвал анимулкулами , и его открытия были снова рассмотрены в свете более поздних открытий клеточной теории . [242]
Кристиан Готфрид Эренберг ввел слово «бактерия» в 1828 году. [243] Фактически, его бактерия представляла собой род, который содержал неспорообразующие палочковидные бактерии. [244] в отличие от Bacillus , рода спорообразующих палочковидных бактерий, определенного Эренбергом в 1835 году. [245]
Луи Пастер продемонстрировал в 1859 году, что рост микроорганизмов вызывает процесс брожения и что этот рост не обусловлен самопроизвольным зарождением ( дрожжи и плесени , обычно связанные с брожением, являются не бактериями, а скорее грибами ). Вместе со своим современником Робертом Кохом Пастер был одним из первых сторонников микробной теории болезней . [246] До них Игнац Земмельвейс и Джозеф Листер осознали важность дезинфекции рук в медицинской работе. Земмельвейс, который в 1840-х годах сформулировал свои правила мытья рук в больнице, еще до появления микробной теории, объяснял болезнь «разложением органических веществ животных». Его идеи были отвергнуты, а его книга на эту тему осуждена медицинским сообществом. Однако после Листера в 1870-х годах врачи начали дезинфицировать руки. [247]
Роберт Кох, пионер медицинской микробиологии, работал над холерой , сибирской язвой и туберкулезом . В своих исследованиях туберкулеза Кох наконец доказал микробную теорию, за что получил Нобелевскую премию в 1905 году. [248] В постулатах Коха он изложил критерии проверки того, является ли организм причиной заболевания , и эти постулаты используются до сих пор. [249]
Фердинанд Кон считается основателем бактериологии , изучающим бактерии с 1870 года. Кон был первым, кто классифицировал бактерии на основе их морфологии. [250] [251]
Хотя в девятнадцатом веке было известно, что бактерии являются причиной многих заболеваний, эффективных антибактериальных методов лечения не существовало. [252] В 1910 году Пауль Эрлих разработал первый антибиотик, заменив красители, избирательно окрашивающие бледную трепонему — спирохету , вызывающую сифилис , на соединения, избирательно убивающие возбудитель. [253] Эрлих, который был удостоен Нобелевской премии 1908 года за свою работу в области иммунологии , был пионером в использовании окрашивания для обнаружения и идентификации бактерий; его работа легла в основу окрашивания по Граму и окрашивания по Цилю-Нильсену . [254]
Важный шаг вперед в изучении бактерий был сделан в 1977 году, когда Карл Везе признал, что археи имеют отдельную от бактерий линию эволюционного происхождения. [255] Эта новая филогенетическая таксономия зависела от секвенирования и 16S рибосомальной РНК разделяла прокариотов на два эволюционных домена как часть трехдоменной системы . [4]
См. также
Ссылки
- ^ «31. Древняя жизнь: микроокаменелости вершинного кремня» . www.lpi.usra.edu . Проверено 12 марта 2022 г.
- ^ Отправитель Р., Фукс С., Майло Р. (19 августа 2016 г.). «Пересмотренные оценки количества клеток человека и бактерий в организме» . ПЛОС Биология . 14 (8): e1002533. дои : 10.1371/journal.pbio.1002533 . ISSN 1545-7885 . ПМЦ 4991899 . ПМИД 27541692 .
- ^ Перейти обратно: а б Маккатчеон Дж.П. (октябрь 2021 г.). «Геномика и клеточная биология внутриклеточных инфекций, полезных для хозяина» . Ежегодный обзор клеточной биологии и биологии развития . 37 (1): 115–142. doi : 10.1146/annurev-cellbio-120219-024122 . ПМИД 34242059 . S2CID 235786110 .
- ^ Перейти обратно: а б с Холл 2008 , с. 145.
- ^ βακτήριον . Лидделл, Генри Джордж ; Скотт, Роберт ; Греко-английский лексикон в проекте «Персей» .
- ^ бактерия Лидделла и Скотта .
- ^ Харпер Д. «Бактерии» . Интернет-словарь этимологии .
- ^ Перейти обратно: а б Краснер 2014 , с. 74.
- ^ Вёзе Ч.Р., Кандлер О., Уилис М.Л. (июнь 1990 г.). «На пути к естественной системе организмов: предложение по доменам архей, бактерий и эукариев» . Труды Национальной академии наук Соединенных Штатов Америки . 87 (12): 4576–79. Бибкод : 1990PNAS...87.4576W . дои : 10.1073/pnas.87.12.4576 . ПМК 54159 . ПМИД 2112744 .
- ^ Холл 2008 , с. 84.
- ^ Годой-Виторино Ф (июль 2019 г.). «Микробная экология человека и развивающаяся новая медицина» . Анналы трансляционной медицины . 7 (14): 342. doi : 10.21037/атм.2019.06.56 . ПМК 6694241 . ПМИД 31475212 .
- ^ Шопф JW (июль 1994 г.). «Разные темпы, разные судьбы: темп и способ эволюции менялись от докембрия к фанерозою» . Труды Национальной академии наук Соединенных Штатов Америки . 91 (15): 6735–42. Бибкод : 1994PNAS...91.6735S . дои : 10.1073/pnas.91.15.6735 . ПМЦ 44277 . ПМИД 8041691 .
- ^ Делонг Э.Ф., Пейс Н.Р. (август 2001 г.). «Экологическое разнообразие бактерий и архей». Систематическая биология . 50 (4): 470–78. CiteSeerX 10.1.1.321.8828 . дои : 10.1080/106351501750435040 . ПМИД 12116647 .
- ^ Браун-младший, Дулиттл В.Ф. (декабрь 1997 г.). «Археи и переход от прокариот к эукариотам» . Обзоры микробиологии и молекулярной биологии . 61 (4): 456–502. doi : 10.1128/mmbr.61.4.456-502.1997 . ПМК 232621 . ПМИД 9409149 .
- ^ Даум Б, Золото V (июнь 2018 г.). «Дергайся или плыви: к пониманию движения прокариот на основе схемы пилуса IV типа». Биологическая химия . 399 (7): 799–808. дои : 10.1515/hsz-2018-0157 . hdl : 10871/33366 . ПМИД 29894297 . S2CID 48352675 .
- ^ Ди Джулио М. (декабрь 2003 г.). «Универсальным предком и предком бактерий были гипертермофилы». Журнал молекулярной эволюции . 57 (6): 721–30. Бибкод : 2003JMolE..57..721D . дои : 10.1007/s00239-003-2522-6 . ПМИД 14745541 . S2CID 7041325 .
- ^ Баттистуцци Ф.У., Фейджао А., Хеджес С.Б. (ноябрь 2004 г.). «Геномная временная шкала эволюции прокариот: понимание происхождения метаногенеза, фототрофии и колонизации суши» . Эволюционная биология BMC . 4:44 . дои : 10.1186/1471-2148-4-44 . ПМЦ 533871 . ПМИД 15535883 .
- ^ Хоманн М., Сансьофре П., Ван Зуилен М., Хойбек С., Гонг Дж., Киллингсворт Б. и др. (23 июля 2018 г.). «Микробная жизнь и биогеохимический круговорот на суше 3220 миллионов лет назад» (PDF) . Природа Геонауки . 11 (9): 665–671. Бибкод : 2018NatGe..11..665H . дои : 10.1038/s41561-018-0190-9 . S2CID 134935568 .
- ^ Габальдон Т. (октябрь 2021 г.). «Происхождение и ранняя эволюция эукариотической клетки» . Ежегодный обзор микробиологии . 75 (1): 631–647. doi : 10.1146/annurev-micro-090817-062213 . ПМИД 34343017 . S2CID 236916203 . Архивировано из оригинала 19 августа 2022 года . Проверено 19 августа 2022 г.
- ^ Каллиер V (8 июня 2022 г.). «Митохондрии и происхождение эукариот» . Знающий журнал . doi : 10.1146/knowable-060822-2 . Проверено 19 августа 2022 г.
- ^ Пул А.М., Пенни Д. (январь 2007 г.). «Оценка гипотез происхождения эукариот». Биоэссе . 29 (1): 74–84. doi : 10.1002/bies.20516 . ПМИД 17187354 .
- ^ Дьялл С.Д., Браун М.Т., Джонсон П.Дж. (апрель 2004 г.). «Древние вторжения: от эндосимбионтов к органеллам». Наука . 304 (5668): 253–257. Бибкод : 2004Sci...304..253D . дои : 10.1126/science.1094884 . ПМИД 15073369 . S2CID 19424594 .
- ^ Стивенс Т.Г., Габр А., Калатрава В., Гроссман А.Р., Бхаттачарья Д. (сентябрь 2021 г.). «Почему первичный эндосимбиоз так редок?» . Новый фитолог . 231 (5): 1693–1699. дои : 10.1111/nph.17478 . ПМЦ 8711089 . ПМИД 34018613 .
- ^ Перейти обратно: а б Бейкер-Остин С., Допсон М. (апрель 2007 г.). «Жизнь в кислоте: гомеостаз pH у ацидофилов». Тенденции в микробиологии . 15 (4): 165–171. дои : 10.1016/j.tim.2007.02.005 . ПМИД 17331729 .
- ^ Чон С.В., Чхве Ю.Дж. (октябрь 2020 г.). «Экстремофильные микроорганизмы для очистки от токсичных загрязнителей окружающей среды» . Молекулы . 25 (21): 4916. doi : 10,3390/molecules25214916 . ПМЦ 7660605 . ПМИД 33114255 .
- ^ Флемминг ХК, Вюрц С. (апрель 2019 г.). «Бактерии и археи на Земле и их численность в биопленках». Обзоры природы. Микробиология . 17 (4): 247–260. дои : 10.1038/s41579-019-0158-9 . ПМИД 30760902 . S2CID 61155774 .
- ^ Бар-Он Ю.М., Филлипс Р., Майло Р. (июнь 2018 г.). «Распределение биомассы на Земле» . Труды Национальной академии наук Соединенных Штатов Америки . 115 (25): 6506–6511. Бибкод : 2018PNAS..115.6506B . дои : 10.1073/pnas.1711842115 . ПМК 6016768 . ПМИД 29784790 .
- ^ Уилис 2008 , с. 362.
- ^ Кушкевич И., Прохазка Ю., Гайдач М., Риттманн С.К., Витезова М. (июнь 2021 г.). «Молекулярная физиология анаэробных фототрофных пурпурных и зеленых серных бактерий» . Международный журнал молекулярных наук . 22 (12): 6398. doi : 10.3390/ijms22126398 . ПМЦ 8232776 . ПМИД 34203823 .
- ^ Уилис 2008 , с. 6.
- ^ Поммервилль 2014 , с. 3–6.
- ^ Перейти обратно: а б с Краснер 2014 , с. 38.
- ^ Перейти обратно: а б с д и ж г Поммервиль 2014 , с. 134.
- ^ Шульц Х.Н., Йоргенсен Б.Б. (2001). «Большие бактерии». Ежегодный обзор микробиологии . 55 : 105–137. дои : 10.1146/аннурев.микро.55.1.105 . ПМИД 11544351 . S2CID 18168018 .
- ^ Уильямс С (2011). «Кого ты называешь простым?». Новый учёный . 211 (2821): 38–41. дои : 10.1016/S0262-4079(11)61709-0 .
- ^ Волланд Дж.М., Гонсалес-Риццо С., Грос О., Тымл Т., Иванова Н., Шульц Ф., Гудо Д., Элизабет Н.Х., Нат Н., Удвари Д., Мальмстрем Р.Р. (18 февраля 2022 г.). «Бактерия длиной в сантиметр, ДНК которой разделена на мембраносвязанные органеллы» . bioRxiv (препринт). дои : 10.1101/2022.02.16.480423 . S2CID 246975579 .
- ^ Сандерсон К. (июнь 2022 г.). «Самая большая бактерия, когда-либо найденная, удивительно сложна». Природа . дои : 10.1038/d41586-022-01757-1 . ПМИД 35750919 . S2CID 250022076 .
- ^ Робертсон Дж., Гомерсолл М., Гилл П. (ноябрь 1975 г.). «Mycoplasma hominis: рост, размножение и выделение мелких жизнеспособных клеток» . Журнал бактериологии . 124 (2): 1007–1018. дои : 10.1128/JB.124.2.1007-1018.1975 . ПМК 235991 . ПМИД 1102522 .
- ^ Велимиров Б (2001). «Нанобактерии, ультрамикробактерии и голодающие формы: поиск мельчайших метаболизирующих бактерий» . Микробы и окружающая среда . 16 (2): 67–77. дои : 10.1264/jsme2.2001.67 .
- ^ Дюзенбери Д.Б. (2009). Жизнь в микромасштабе . Кембридж, Массачусетс: Издательство Гарвардского университета . стр. 20–25. ISBN 978-0-674-03116-6 .
- ^ Ян, округ Колумбия, Блэр К.М., Салама Н.Р. (март 2016 г.). «Оставаться в форме: влияние формы клеток на выживание бактерий в различных средах» . Обзоры микробиологии и молекулярной биологии . 80 (1): 187–203. дои : 10.1128/MMBR.00031-15 . ПМЦ 4771367 . ПМИД 26864431 .
- ^ Кабин М.Т., Джейкобс-Вагнер С. (август 2005 г.). «Форма бактериальной клетки». Обзоры природы. Микробиология . 3 (8): 601–10. дои : 10.1038/nrmicro1205 . ПМИД 16012516 . S2CID 23938989 .
- ^ Молодой К.Д. (сентябрь 2006 г.). «Селективное значение формы бактерий» . Обзоры микробиологии и молекулярной биологии . 70 (3): 660–703. дои : 10.1128/MMBR.00001-06 . ПМЦ 1594593 . ПМИД 16959965 .
- ^ Кроуфорд 2007 , с. xi.
- ^ Классен Д., Розен Д.Е., Койперс О.П., Согаард-Андерсен Л., ван Везель Г.П. (февраль 2014 г.). «Бактериальные решения проблемы многоклеточности: рассказ о биопленках, нитях и плодовых телах» . Обзоры природы. Микробиология . 12 (2): 115–24. дои : 10.1038/nrmicro3178 . hdl : 11370/0db66a9c-72ef-4e11-a75d-9d1e5827573d . ПМИД 24384602 . S2CID 20154495 .
- ^ Шимкец ЖЖ (1999). «Межклеточная передача сигналов во время развития плодовых тел Myxococcus xanthus». Ежегодный обзор микробиологии . 53 : 525–49. дои : 10.1146/аннурев.микро.53.1.525 . ПМИД 10547700 .
- ^ Перейти обратно: а б Кайзер Д. (2004). «Сигнализация в миксобактериях». Ежегодный обзор микробиологии . 58 : 75–98. дои : 10.1146/annurev.micro.58.030603.123620 . ПМИД 15487930 .
- ^ Уилис 2008 , с. 75.
- ^ Мандал А., Дутта А., Дас Р., Мукерджи Дж. (июнь 2021 г.). «Роль приливных микробных сообществ в секвестрации углекислого газа и удалении загрязняющих веществ: обзор». Бюллетень о загрязнении морской среды . 170 : 112626. Бибкод : 2021MarPB.17012626M . doi : 10.1016/j.marpolbul.2021.112626 . ПМИД 34153859 .
- ^ Донлан Р.М. (сентябрь 2002 г.). «Биопленки: микробная жизнь на поверхностях» . Новые инфекционные заболевания . 8 (9): 881–90. дои : 10.3201/eid0809.020063 . ПМЦ 2732559 . ПМИД 12194761 .
- ^ Бранда С.С., Вик С., Фридман Л., Колтер Р. (январь 2005 г.). «Биопленки: новый взгляд на матрицу». Тенденции в микробиологии . 13 (1): 20–26. дои : 10.1016/j.tim.2004.11.006 . ПМИД 15639628 .
- ^ Перейти обратно: а б Дэйви М.Э., О'Тул Дж.А. (декабрь 2000 г.). «Микробные биопленки: от экологии к молекулярной генетике» . Обзоры микробиологии и молекулярной биологии . 64 (4): 847–67. дои : 10.1128/MMBR.64.4.847-867.2000 . ПМК 99016 . ПМИД 11104821 .
- ^ Донлан Р.М., Костертон Дж.В. (апрель 2002 г.). «Биопленки: механизмы выживания клинически значимых микроорганизмов» . Обзоры клинической микробиологии . 15 (2): 167–93. doi : 10.1128/CMR.15.2.167-193.2002 . ПМК 118068 . ПМИД 11932229 .
- ^ Слончевски Дж.Л., Фостер Дж.В. (2013). Микробиология: развивающаяся наука (Третье изд.). Нью-Йорк: WW Нортон. п. 82. ИСБН 978-0-393-12367-8 .
- ^ Фейхоо-Сиота Л., Рама Х.Л., Санчес-Перес А., Вилла Т.Г. (июль 2017 г.). «Соображения о бактериальных нуклеоидах». Прикладная микробиология и биотехнология . 101 (14): 5591–602. дои : 10.1007/s00253-017-8381-7 . ПМИД 28664324 . S2CID 10173266 .
- ^ Бобик Т.А. (май 2006 г.). «Многогранные органеллы, разделяющие бактериальные метаболические процессы». Прикладная микробиология и биотехнология . 70 (5): 517–25. дои : 10.1007/s00253-005-0295-0 . ПМИД 16525780 . S2CID 8202321 .
- ^ Йейтс Т.О., Керфельд К.А., Хайнхорст С., Кэннон Г.К., Шайвли Дж.М. (сентябрь 2008 г.). «Белковые органеллы бактерий: карбоксисомы и родственные микрокомпарты». Обзоры природы. Микробиология . 6 (9): 681–91. дои : 10.1038/nrmicro1913 . ПМИД 18679172 . S2CID 22666203 .
- ^ Керфельд К.А., Савая М.Р., Танака С., Нгуен К.В., Филлипс М., Биби М., Йейтс Т.О. (август 2005 г.). «Белковые структуры, образующие оболочку примитивных бактериальных органелл». Наука . 309 (5736): 936–38. Бибкод : 2005Sci...309..936K . CiteSeerX 10.1.1.1026.896 . дои : 10.1126/science.1113397 . ПМИД 16081736 . S2CID 24561197 .
- ^ Гитай З. (март 2005 г.). «Новая биология бактериальных клеток: движущиеся части и субклеточная архитектура» . Клетка . 120 (5): 577–86. дои : 10.1016/j.cell.2005.02.026 . ПМИД 15766522 . S2CID 8894304 .
- ^ Ши Ю.Л., Ротфилд Л. (сентябрь 2006 г.). «Бактериальный цитоскелет» . Обзоры микробиологии и молекулярной биологии . 70 (3): 729–54. дои : 10.1128/MMBR.00017-06 . ПМЦ 1594594 . ПМИД 16959967 .
- ^ Норрис В., ден Блаувен Т., Кабин-Фламан А., Дой Р.Х., Харши Р., Яньер Л., Хименес-Санчес А., Джин Д.Д., Левин П.А., Милейковская Е., Мински А., Сайер М., Скарстад К. (март 2007 г.). «Функциональная систематика бактериальных гиперструктур» . Обзоры микробиологии и молекулярной биологии . 71 (1): 230–53. дои : 10.1128/MMBR.00035-06 . ПМЦ 1847379 . ПМИД 17347523 .
- ^ Поммервилль, 2014 , стр. 120–121.
- ^ Брайант Д.А., Фригаард Н.У. (ноябрь 2006 г.). «Освещение фотосинтеза и фототрофии прокариот». Тенденции в микробиологии . 14 (11): 488–96. дои : 10.1016/j.tim.2006.09.001 . ПМИД 16997562 .
- ^ Псенчик Дж., Иконен Т.П., Лауринмяки П., Меркель М.С., Батчер С.Дж., Серимаа Р.Э., Тума Р. (август 2004 г.). «Ламеллярная организация пигментов в хлоросомах — светособирающих комплексах зеленых фотосинтезирующих бактерий» . Биофизический журнал . 87 (2): 1165–72. Бибкод : 2004BpJ....87.1165P . дои : 10.1529/biophysj.104.040956 . ПМК 1304455 . ПМИД 15298919 .
- ^ Танбихлер М., Ван С.К., Шапиро Л. (октябрь 2005 г.). «Бактериальный нуклеоид: высокоорганизованная и динамичная структура» . Журнал клеточной биохимии . 96 (3): 506–21. дои : 10.1002/jcb.20519 . ПМИД 15988757 . S2CID 25355087 .
- ^ Поелсгаард Дж., Даутвейт С. (ноябрь 2005 г.). «Бактериальная рибосома как мишень для антибиотиков». Обзоры природы. Микробиология . 3 (11): 870–81. дои : 10.1038/nrmicro1265 . ПМИД 16261170 . S2CID 7521924 .
- ^ Йео М., Чейтер К. (март 2005 г.). «Взаимодействие метаболизма и дифференцировки гликогена дает представление о биологии развития Streptomyces coelicolor» . Микробиология . 151 (Часть 3): 855–61. дои : 10.1099/mic.0.27428-0 . ПМИД 15758231 . Архивировано из оригинала 29 сентября 2007 года.
- ^ Шиба Т., Цуцуми К., Исигэ К., Ногучи Т. (март 2000 г.). «Неорганический полифосфат и полифосфаткиназа: их новые биологические функции и применение» . Биохимия. Биохимия . 65 (3): 315–23. ПМИД 10739474 . Архивировано из оригинала 25 сентября 2006 года.
- ^ Брюн, округ Колумбия (июнь 1995 г.). «Выделение и характеристика белков глобул серы из Chromatium vinosum и Thiocapsaroseopersicina». Архив микробиологии . 163 (6): 391–99. Бибкод : 1995ArMic.163..391B . дои : 10.1007/BF00272127 . ПМИД 7575095 . S2CID 22279133 .
- ^ Кадури Д., Юркевич Е., Окон Ю., Кастро-Совински С. (2005). «Экологическое и сельскохозяйственное значение бактериальных полигидроксиалканоатов». Критические обзоры по микробиологии . 31 (2): 55–67. дои : 10.1080/10408410590899228 . ПМИД 15986831 . S2CID 4098268 .
- ^ Уолсби А.Е. (март 1994 г.). «Газовые пузырьки» . Микробиологические обзоры . 58 (1): 94–144. дои : 10.1128/ММБР.58.1.94-144.1994 . ПМК 372955 . ПМИД 8177173 .
- ^ ван Хейенорт Дж (март 2001 г.). «Образование гликановых цепей при синтезе бактериального пептидогликана» . Гликобиология . 11 (3): 25П–36П. дои : 10.1093/гликоб/11.3.25R . ПМИД 11320055 . S2CID 46066256 .
- ^ Перейти обратно: а б Кох А.Л. (октябрь 2003 г.). «Бактериальная стена как мишень для нападения: исследования прошлого, настоящего и будущего» . Обзоры клинической микробиологии . 16 (4): 673–87. doi : 10.1128/CMR.16.4.673-687.2003 . ПМК 207114 . ПМИД 14557293 .
- ^ Перейти обратно: а б Грамм ХК (1884 г.). «Об изолированном окрашивании шизомицетов в срезанных и сухих препаратах». Прогресс Мед . 2 : 185-89.
- ^ Гугенгольц П. (2002). «Изучение прокариотического разнообразия в эпоху генома» . Геномная биология . 3 (2): ОБЗОРЫ0003. doi : 10.1186/gb-2002-3-2-reviews0003 . ПМК 139013 . ПМИД 11864374 .
- ^ Уолш FM, Эмис С.Г. (октябрь 2004 г.). «Микробиология и механизмы лекарственной устойчивости полностью устойчивых патогенов» (PDF) . Современное мнение в микробиологии . 7 (5): 439–44. дои : 10.1016/j.mib.2004.08.007 . ПМИД 15451497 .
- ^ Олдервик Л.Дж., Харрисон Дж., Ллойд Г.С., Берч Х.Л. (март 2015 г.). «Клеточная стенка микобактерий – пептидогликан и арабиногалактан» . Перспективы Колд-Спринг-Харбора в медицине . 5 (8): а021113. doi : 10.1101/cshperspect.a021113 . ПМЦ 4526729 . ПМИД 25818664 .
- ^ Фэган Р.П., Фэйрвезер Н.Ф. (март 2014 г.). «Биогенез и функции бактериальных S-слоев» (PDF) . Обзоры природы. Микробиология . 12 (3): 211–22. дои : 10.1038/nrmicro3213 . ПМИД 24509785 . S2CID 24112697 .
- ^ Томпсон С.А. (декабрь 2002 г.). «Поверхностные слои Campylobacter (S-слои) и уклонение от иммунитета» . Анналы пародонтологии . 7 (1): 43–53. дои : 10.1902/анналы.2002.7.1.43 . ПМЦ 2763180 . ПМИД 16013216 .
- ^ Беверидж Т.Дж., Пауэлс П.Х., Сара М., Котиранта А., Лунатмаа К., Кари К., Керосуо Е., Хаапасало М., Эгельсир Э.М., Шохер И., Слейтр У.Б., Морелли Л., Каллегари М.Л., Номеллини Дж.Ф., Бингл В.Х., Смит Дж., Лейбовиц Е. , Лемэр М., Мирас И., Саламиту С., Беген П., Охайон Х., Гунон П., Матушек М., Коваль С.Ф. (июнь 1997 г.). «Функции S-слоев». Обзоры микробиологии FEMS . 20 (1–2): 99–149. дои : 10.1111/j.1574-6976.1997.tb00305.x . ПМИД 9276929 .
- ^ Кодзима С., Блер Д.Ф. (2004). Бактериальный жгутиковый мотор: структура и функция сложной молекулярной машины . Международный обзор цитологии. Том. 233. С. 93–134. дои : 10.1016/S0074-7696(04)33003-2 . ISBN 978-0-12-364637-8 . ПМИД 15037363 .
- ^ Уилис 2008 , с. 76.
- ^ Ченг Р.А., Видманн М. (2020). «Последние достижения в нашем понимании разнообразия и роли фимбрий шаперона-Ашера в содействии тропизму хозяина сальмонеллы и тканям» . Границы клеточной и инфекционной микробиологии . 10 : 628043. дои : 10.3389/fcimb.2020.628043 . ПМЦ 7886704 . ПМИД 33614531 .
- ^ Сильверман ПМ (февраль 1997 г.). «К структурной биологии бактериальной конъюгации» . Молекулярная микробиология . 23 (3): 423–29. дои : 10.1046/j.1365-2958.1997.2411604.x . ПМИД 9044277 . S2CID 24126399 .
- ^ Коста Т.Р., Фелисберто-Родригес К., Меир А., Превост М.С., Реджей А., Троктер М., Ваксман Г. (июнь 2015 г.). «Системы секреции грамотрицательных бактерий: структурные и механические сведения». Обзоры природы. Микробиология . 13 (6): 343–59. дои : 10.1038/nrmicro3456 . ПМИД 25978706 . S2CID 8664247 .
- ^ Луонг П., Дубе Д.Х. (июль 2021 г.). «Демонтаж бактериального гликокаликса: химические инструменты для исследования, возмущения и изображения бактериальных гликанов» . Биоорганическая и медицинская химия . 42 : 116268. doi : 10.1016/j.bmc.2021.116268 . ISSN 0968-0896 . ПМЦ 8276522 . ПМИД 34130219 .
- ^ Стоукс Р.В., Норрис-Джонс Р., Брукс Д.Е., Беверидж Т.Дж., Докси Д., Торсон Л.М. (октябрь 2004 г.). «Богатый гликанами внешний слой клеточной стенки Mycobacterium Tuberculosis действует как антифагоцитарная капсула, ограничивающая ассоциацию бактерии с макрофагами» . Инфекция и иммунитет . 72 (10): 5676–86. дои : 10.1128/IAI.72.10.5676-5686.2004 . ПМК 517526 . ПМИД 15385466 .
- ^ Перейти обратно: а б Калшойер Р., Паласиос А., Ансо И., Сифуэнте Дж., Ангита Дж., Джейкобс В.Р., Герен М.Е., Прадос-Росалес Р. (июль 2019 г.). «Капсула микобактерии туберкулеза: клеточная структура, имеющая ключевое значение в патогенезе» . Биохимический журнал . 476 (14): 1995–2016. дои : 10.1042/BCJ20190324 . ПМК 6698057 . ПМИД 31320388 .
- ^ Джерниган Дж.А., Стивенс Д.С., Эшфорд Д.А., Оменака К., Топиэль М.С., Гэлбрейт М., Таппер М., Фиск Т.Л., Заки С., Попович Т., Мейер Р.Ф., Куинн К.П., Харпер С.А., Фридкин С.К., Сейвар Дж.Дж., Шепард К.В., МакКоннелл М. , Гуарнер Дж., Ши В.Дж., Малецки Дж.М., Гербердинг Дж.Л., Хьюз Дж.М., Перкинс Б.А. (2001). «Ингаляционная форма сибирской язвы, связанная с биотерроризмом: первые 10 случаев зарегистрированы в США» . Новые инфекционные заболевания . 7 (6): 933–44. дои : 10.3201/eid0706.010604 . ПМК 2631903 . ПМИД 11747719 .
- ^ Николсон В.Л., Мунаката Н., Хорнек Г., Мелош Х.Дж., Сетлоу П. (сентябрь 2000 г.). «Устойчивость эндоспор Bacillus к экстремальным земным и внеземным средам» . Обзоры микробиологии и молекулярной биологии . 64 (3): 548–72. дои : 10.1128/MMBR.64.3.548-572.2000 . ПМК 99004 . ПМИД 10974126 .
- ^ Перейти обратно: а б Маккенни П.Т., Дрикс А., Айхенбергер П. (январь 2013 г.). «Эндоспора Bacillus subtilis: сборка и функции многослойной оболочки» . Обзоры природы. Микробиология . 11 (1): 33–44. дои : 10.1038/nrmicro2921 . ПМК 9910062 . ПМИД 23202530 . S2CID 205498395 .
- ^ Николсон В.Л., Фахардо-Кавасос П., Ребейл Р., Слиман Т.А., Ризенман П.Дж., Лоу Дж.Ф., Сюэ Ю (август 2002 г.). «Бактериальные эндоспоры и их значение в стрессоустойчивости». Антони ван Левенгук . 81 (1–4): 27–32. дои : 10.1023/A:1020561122764 . ПМИД 12448702 . S2CID 30639022 .
- ^ Вриланд Р.Х., Розенцвейг В.Д., Пауэрс Д.В. (октябрь 2000 г.). «Выделение галотолерантной бактерии возрастом 250 миллионов лет из первичного кристалла соли». Природа . 407 (6806): 897–900. Бибкод : 2000Natur.407..897V . дои : 10.1038/35038060 . ПМИД 11057666 . S2CID 9879073 .
- ^ Кано Р.Дж., Боруки М.К. (май 1995 г.). «Возрождение и идентификация бактериальных спор в доминиканском янтаре возрастом от 25 до 40 миллионов лет». Наука . 268 (5213): 1060–64. Бибкод : 1995Sci...268.1060C . дои : 10.1126/science.7538699 . ПМИД 7538699 .
- ^ «Ссора над древними бактериями» . Новости Би-би-си . 7 июня 2001 года . Проверено 26 апреля 2020 г. .
- ^ Николсон В.Л., Шуергер А.С., Сетлоу П. (апрель 2005 г.). «Солнечная УФ-среда и устойчивость бактериальных спор к УФ-излучению: соображения по транспортировке с Земли на Марс посредством естественных процессов и полета человека в космос». Мутационные исследования . 571 (1–2): 249–64. Бибкод : 2005MRFMM.571..249N . дои : 10.1016/j.mrfmmm.2004.10.012 . ПМИД 15748651 .
- ^ «Колонизировать галактику сложно. Почему бы вместо этого не послать бактерии?» . Экономист . 12 апреля 2018 г. ISSN 0013-0613 . Проверено 26 апреля 2020 г. .
- ^ Revitt-Mills SA, Видор С.Дж., Уоттс Т.Д., Лирас Д., Руд Дж.И., Адамс В. (май 2019 г.). «Плазмиды вирулентности патогенных клостридий» . Микробиологический спектр . 7 (3). doi : 10.1128/microbiolspec.GPP3-0034-2018 . ПМИД 31111816 . S2CID 160013108 .
- ^ Рейгадас Э., ван Прен Дж., Фальконе М., Фитцпатрик Ф., Верешильд М.Дж., Куйпер Э.Дж., Буза Э. (июль 2021 г.). «Как: профилактические мероприятия для предотвращения инфекции Clostridioides difficile» . Клиническая микробиология и инфекции . 27 (12): 1777–1783. дои : 10.1016/j.cmi.2021.06.037 . hdl : 1887/3249077 . ПМИД 34245901 .
- ^ Нилсон К.Х. (январь 1999 г.). «Микробиология после викингов: новые подходы, новые данные, новые идеи». Происхождение жизни и эволюция биосферы . 29 (1): 73–93. Бибкод : 1999ОЛЕВ...29...73Н . дои : 10.1023/А:1006515817767 . ПМИД 11536899 . S2CID 12289639 .
- ^ Сюй Дж (июнь 2006 г.). «Микробная экология в эпоху геномики и метагеномики: концепции, инструменты и последние достижения» . Молекулярная экология . 15 (7): 1713–31. дои : 10.1111/j.1365-294X.2006.02882.x . ПМИД 16689892 . S2CID 16374800 .
- ^ Зиллиг В. (декабрь 1991 г.). «Сравнительная биохимия архей и бактерий». Текущее мнение в области генетики и развития . 1 (4): 544–51. дои : 10.1016/S0959-437X(05)80206-0 . ПМИД 1822288 .
- ^ Перейти обратно: а б с Слончевски Дж.Л., Фостер Дж.В. Микробиология: развивающаяся наука (3-е изд.). WW Нортон и компания. стр. 491–44.
- ^ Хеллингверф К.Дж., Крилаард В., Хофф В.Д., Маттейс Х.К., Мур Л.Р., ван Роттердам Б.Дж. (1994). «Фотобиология бактерий» . Антони ван Левенгук (Представлена рукопись). 65 (4): 331–47. дои : 10.1007/BF00872217 . ПМИД 7832590 . S2CID 23438926 .
- ^ Далтон Х (июнь 2005 г.). «Лекция Левенгука 2000 года о естественной и неестественной истории метанокисляющих бактерий» . Философские труды Лондонского королевского общества. Серия Б, Биологические науки . 360 (1458): 1207–22. дои : 10.1098/rstb.2005.1657 . ПМК 1569495 . ПМИД 16147517 .
- ^ Имран А., Хаким С., Тарик М., Наваз М.С., Лараиб И., Гульзар У., Ханиф М.К., Сиддик М.Дж., Хаят М., Фраз А., Ахмад М. (2021). «Диазотрофы для снижения кризисов, вызванных азотным загрязнением: взгляд глубоко в корни» . Границы микробиологии . 12 : 637815. doi : 10.3389/fmicb.2021.637815 . ПМК 8180554 . ПМИД 34108945 .
- ^ Зер Дж.П., Дженкинс Б.Д., Шорт С.М., Стюард Г.Ф. (июль 2003 г.). «Разнообразие генов нитрогеназы и структура микробного сообщества: межсистемное сравнение» . Экологическая микробиология . 5 (7): 539–54. Бибкод : 2003EnvMi...5..539Z . дои : 10.1046/j.1462-2920.2003.00451.x . ПМИД 12823187 .
- ^ Косуги Й., Мацуура Н., Лян К., Ямамото-Икемото Р. (октябрь 2020 г.). «Очистка сточных вод с использованием процесса сульфатредукции, денитрификации Анаммокс и частичной нитрификации (SRDAPN)». Хемосфера . 256 : 127092. Бибкод : 2020Chmsp.25627092K . doi : 10.1016/j.chemSphere.2020.127092 . ПМИД 32559887 . S2CID 219476361 .
- ^ Морель Ф.М., Крапил А.М., Амиот М. (1998). «Химический круговорот и биоаккумуляция ртути». Ежегодный обзор экологии и систематики . 29 : 543–66. doi : 10.1146/annurev.ecolsys.29.1.543 . S2CID 86336987 .
- ^ Слесак И, Кула М, Слесак Х, Мишальски З, Стшалка К (август 2019 г.). «Как определить обязательный анаэробиоз? Эволюционный взгляд на систему антиоксидантного ответа и ранние этапы эволюции жизни на Земле» . Свободно-радикальная биология и медицина . 140 : 61–73. doi : 10.1016/j.freeradbiomed.2019.03.004 . ПМИД 30862543 .
- ^ Стюарт Э.Дж., Мэдден Р., Пол Дж., Таддей Ф. (февраль 2005 г.). «Старение и смерть организма, размножающегося морфологически симметричным делением» . ПЛОС Биология . 3 (2): е45. doi : 10.1371/journal.pbio.0030045 . ПМК 546039 . ПМИД 15685293 .
- ^ Кох А.Л. (2002). «Контроль бактериального клеточного цикла путем роста цитоплазмы». Критические обзоры по микробиологии . 28 (1): 61–77. дои : 10.1080/1040-840291046696 . ПМИД 12003041 . S2CID 11624182 .
- ^ Поммервилль 2014 , с. 138.
- ^ Поммервилль 2014 , с. 557.
- ^ Перейти обратно: а б Уилис 2008 , с. 42.
- ^ Перейти обратно: а б с Томсон Р.Б., Бертрам Х. (декабрь 2001 г.). «Лабораторная диагностика инфекций центральной нервной системы». Клиники инфекционных заболеваний Северной Америки . 15 (4): 1047–71. дои : 10.1016/S0891-5520(05)70186-0 . ПМИД 11780267 .
- ^ Паерл Х.В., Фултон Р.С., Мойсандер П.Х., Дайбл Дж. (апрель 2001 г.). «Вредное цветение пресноводных водорослей с акцентом на цианобактерии» . Научный мировой журнал . 1 : 76–113. дои : 10.1100/tsw.2001.16 . ПМК 6083932 . ПМИД 12805693 .
- ^ Чаллис Г.Л., Хопвуд Д.А. (ноябрь 2003 г.). «Синергия и непредвиденные обстоятельства как движущие силы эволюции производства множественных вторичных метаболитов видами Streptomyces» . Труды Национальной академии наук Соединенных Штатов Америки . 100 (Приложение 2): 14555–61. Бибкод : 2003PNAS..10014555C . дои : 10.1073/pnas.1934677100 . ПМК 304118 . ПМИД 12970466 .
- ^ Койман С.А., Огер П., Поджиале Дж.К., Кои Б.В. (август 2003 г.). «Количественные этапы симбиогенеза и эволюция гомеостаза». Биологические обзоры Кембриджского философского общества . 78 (3): 435–63. дои : 10.1017/S1464793102006127 . ПМИД 14558592 . S2CID 41072709 .
- ^ Бертран РЛ (2019). «Лаг-фаза — это динамичный, организованный, адаптивный и развивающийся период, который подготавливает бактерии к делению клеток» . Журнал бактериологии . 201 (7): e00697-18. дои : 10.1128/JB.00697-18 . ПМК 6416914 . ПМИД 30642990 .
- ^ Пратс К., Лопес Д., Жиро А., Феррер Дж., Валлс Дж. (август 2006 г.). «Индивидуальное моделирование бактериальных культур для изучения микроскопических причин лаг-фазы». Журнал теоретической биологии . 241 (4): 939–53. Бибкод : 2006JThBi.241..939P . дои : 10.1016/j.jtbi.2006.01.029 . ПМИД 16524598 .
- ^ Хекер М., Фёлькер Ю (2001). Общая реакция на стресс Bacillus subtilis и других бактерий . Достижения микробной физиологии. Том. 44. С. 35–91. дои : 10.1016/S0065-2911(01)44011-2 . ISBN 978-0-12-027744-5 . ПМИД 11407115 .
- ^ Слончевски Дж.Л., Фостер Дж.В. Микробиология: развивающаяся наука (3-е изд.). WW Нортон и компания. п. 143.
- ^ Леппянен М., Сундберг Л.Р., Лаанто Э., де Фрейтас Алмейда ГМ, Паппонен П., Маасилта И.Ю. (август 2017 г.). «Визуализация бактериальных колоний и взаимодействия фага и бактерии с субнанометровым разрешением с помощью гелий-ионной микроскопии» . Продвинутые биосистемы . 1 (8): e1700070. дои : 10.1002/adbi.201700070 . ПМИД 32646179 . S2CID 90960276 .
- ^ Накабати А., Ямасита А., Тох Х., Исикава Х., Данбар Х.Э., Моран Н.А., Хаттори М. (октябрь 2006 г.). «Геном бактериального эндосимбионта Carsonella длиной 160 тысяч оснований». Наука . 314 (5797): 267. doi : 10.1126/science.1134196 . ПМИД 17038615 . S2CID 44570539 .
- ^ Праделла С., Ханс А., Шпроер С., Райхенбах Х., Герт К., Бейер С. (декабрь 2002 г.). «Характеристика, размер генома и генетические манипуляции миксобактерии Sorangium cellulosum So ce56». Архив микробиологии . 178 (6): 484–92. Бибкод : 2002ArMic.178..484P . дои : 10.1007/s00203-002-0479-2 . ПМИД 12420170 . S2CID 21023021 .
- ^ Хиннебуш Дж., Тилли К. (декабрь 1993 г.). «Линейные плазмиды и хромосомы бактерий» . Молекулярная микробиология . 10 (5): 917–22. дои : 10.1111/j.1365-2958.1993.tb00963.x . ПМИД 7934868 . S2CID 23852021 .
- ^ Лин Ю.С., Кизер Х.М., Хопвуд Д.А., Чен К.В. (декабрь 1993 г.). «Хромосомная ДНК Streptomyces lividans 66 является линейной». Молекулярная микробиология . 10 (5): 923–33. дои : 10.1111/j.1365-2958.1993.tb00964.x . ПМИД 7934869 . S2CID 8536066 .
- ^ Вал М.Е., Солер-Бистю А., Бланд М.Дж., Мазель Д. (декабрь 2014 г.). «Управление многочастными геномами: модель холерного вибриона» . Современное мнение в микробиологии . 22 : 120–26. дои : 10.1016/j.mib.2014.10.003 . ПМИД 25460805 . S2CID 215743285 .
- ^ Кадо CI (октябрь 2014 г.). «Исторические события, породившие область плазмидной биологии» . Микробиологический спектр . 2 (5): 3. doi : 10.1128/microbiolspec.PLAS-0019-2013 . ПМИД 26104369 .
- ^ Белфорт М. , Рибан М.Э., Кутзи Т., Далгаард Дж.З. (июль 1995 г.). «Прокариотические интроны и интеины: множество форм и функций» . Журнал бактериологии . 177 (14): 3897–903. дои : 10.1128/jb.177.14.3897-3903.1995 . ПМК 177115 . ПМИД 7608058 .
- ^ Денамур Э., Матич I (май 2006 г.). «Эволюция частоты мутаций у бактерий» . Молекулярная микробиология . 60 (4): 820–27. дои : 10.1111/j.1365-2958.2006.05150.x . ПМИД 16677295 . S2CID 20713095 .
- ^ Райт Б.Е. (май 2004 г.). «Адаптивные мутации и эволюция, направленные на стресс» . Молекулярная микробиология . 52 (3): 643–50. дои : 10.1111/j.1365-2958.2004.04012.x . ПМИД 15101972 . S2CID 1071308 .
- ^ Чен И, Дубнау Д (март 2004 г.). «Поглощение ДНК во время бактериальной трансформации». Обзоры природы. Микробиология . 2 (3): 241–49. дои : 10.1038/nrmicro844 . ПМИД 15083159 . S2CID 205499369 .
- ^ Джонсборг О., Эльдхольм В., Ховарштайн Л.С. (декабрь 2007 г.). «Естественная генетическая трансформация: распространенность, механизмы и функции» . Исследования в области микробиологии . 158 (10): 767–78. дои : 10.1016/j.resmic.2007.09.004 . ПМИД 17997281 .
- ^ Бернштейн Х, Бернштейн С, Мишод Р.Э. (2012). «Репарация ДНК как основная адаптивная функция пола у бактерий и эукариот». Глава 1: стр. 1–49 в: Восстановление ДНК: новые исследования , Сакура Кимура и Сора Симидзу (ред.). Новая наука. Издательство, Хоппож, Нью-Йорк ISBN 978-1-62100-808-8 .
- ^ Брюссов Х., Канчая С., Хардт В.Д. (сентябрь 2004 г.). «Фаги и эволюция бактериальных патогенов: от геномных перестроек к лизогенной конверсии» . Обзоры микробиологии и молекулярной биологии . 68 (3): 560–602, оглавление. дои : 10.1128/MMBR.68.3.560-602.2004 . ПМК 515249 . ПМИД 15353570 .
- ^ Бикл Т.А., Крюгер Д.Х. (июнь 1993 г.). «Биология рестрикции ДНК» . Микробиологические обзоры . 57 (2): 434–50. дои : 10.1128/MMBR.57.2.434-450.1993 . ПМК 372918 . ПМИД 8336674 .
- ^ Баррангу Р., Фремо С., Дево Х., Ричардс М., Бояваль П., Муано С., Ромеро Д.А., Хорват П. (март 2007 г.). «CRISPR обеспечивает приобретенную устойчивость прокариот к вирусам». Наука . 315 (5819): 1709–12. Бибкод : 2007Sci...315.1709B . дои : 10.1126/science.1138140 . hdl : 20.500.11794/38902 . ПМИД 17379808 . S2CID 3888761 .
- ^ Браунс С.Дж., Джор М.М., Лундгрен М., Вестра Э.Р., Слейхуис Р.Дж., Снейдерс А.П., Дикман М.Дж., Макарова К.С., Кунин Е.В., ван дер Ост Дж. (август 2008 г.). «Маленькие РНК CRISPR обеспечивают противовирусную защиту прокариот» . Наука . 321 (5891): 960–64. Стартовый код : 2008Sci...321..960B . дои : 10.1126/science.1159689 . ПМЦ 5898235 . ПМИД 18703739 .
- ^ Кабесон Э, Риполь-Розада Х, Пенья А, де ла Крус Ф, Аречага I (январь 2015 г.). «На пути к интегрированной модели бактериальной конъюгации» . Обзоры микробиологии FEMS . 39 (1): 81–95. дои : 10.1111/1574-6976.12085 . ПМИД 25154632 . S2CID 34245150 .
- ^ Мишод Р.Э., Бернштейн Х., Недельку А.М. (май 2008 г.). «Адаптационное значение пола у микробных патогенов» (PDF) . Инфекция, генетика и эволюция . 8 (3): 267–85. Бибкод : 2008InfGE...8..267M . дои : 10.1016/j.meegid.2008.01.002 . ПМИД 18295550 . Архивировано (PDF) из оригинала 30 декабря 2016 года.
- ^ Гастингс П.Дж., Розенберг С.М., Slack A (сентябрь 2004 г.). «Боковая передача устойчивости к антибиотикам, вызванная антибиотиками». Тенденции в микробиологии . 12 (9): 401–14. дои : 10.1016/j.tim.2004.07.003 . ПМИД 15337159 .
- ^ Дэвисон Дж. (сентябрь 1999 г.). «Генетический обмен между бактериями в окружающей среде». Плазмида . 42 (2): 73–91. дои : 10.1006/plas.1999.1421 . ПМИД 10489325 .
- ^ Перейти обратно: а б с Ким К.В. (декабрь 2017 г.). «Электронно-микроскопические наблюдения поверхностных придатков прокариот». Журнал микробиологии (Сеул, Корея) . 55 (12): 919–26. дои : 10.1007/s12275-017-7369-4 . ПМИД 29214488 . S2CID 89872403 .
- ^ Макнаб РМ (декабрь 1999 г.). «Бактериальный жгутик: реверсивный вращающийся пропеллер и экспортный аппарат типа III» . Журнал бактериологии . 181 (23): 7149–53. дои : 10.1128/JB.181.23.7149-7153.1999 . ПМЦ 103673 . ПМИД 10572114 .
- ^ Ву М., Робертс Дж.В., Ким С., Кох Д.Л., ДеЛиза MP (июль 2006 г.). «Коллективная бактериальная динамика выявлена с помощью трехмерного метода отслеживания дефокусированных частиц в масштабе популяции» . Прикладная и экологическая микробиология . 72 (7): 4987–94. Бибкод : 2006ApEnM..72.4987W . дои : 10.1128/АЕМ.00158-06 . ПМЦ 1489374 . ПМИД 16820497 .
- ^ Мэттик Дж.С. (2002). «Пили IV типа и подергивания моторики». Ежегодный обзор микробиологии . 56 : 289–314. дои : 10.1146/annurev.micro.56.012302.160938 . ПМИД 12142488 .
- ^ Мерц А.Дж., Со М., член парламента Шитца (сентябрь 2000 г.). «Втягивание пилуса приводит к подергивающейся подвижности бактерий». Природа . 407 (6800): 98–102. Бибкод : 2000Natur.407...98M . дои : 10.1038/35024105 . ПМИД 10993081 . S2CID 4425775 .
- ^ Люкс Р., Ши В (июль 2004 г.). «Движения бактерий, управляемые хемотаксисом» . Критические обзоры по оральной биологии и медицине . 15 (4): 207–20. дои : 10.1177/154411130401500404 . ПМИД 15284186 .
- ^ Швайницер Т., Йозенханс К. (июль 2010 г.). «Бактериальные энергетические такси: глобальная стратегия?» . Архив микробиологии . 192 (7): 507–20. Бибкод : 2010ArMic.192..507S . дои : 10.1007/s00203-010-0575-7 . ПМЦ 2886117 . ПМИД 20411245 .
- ^ Франкель Р.Б., Базылински Д.А., Джонсон М.С., Тейлор Б.Л. (август 1997 г.). «Магнитоаэротаксис морских кокковидных бактерий» . Биофизический журнал . 73 (2): 994–1000. Бибкод : 1997BpJ....73..994F . дои : 10.1016/S0006-3495(97)78132-3 . ПМК 1180996 . ПМИД 9251816 .
- ^ Нан Б., Зусман Д.Р. (2011). «Раскрытие тайны скользящей подвижности миксобактерий» . Ежегодный обзор генетики . 45 : 21–39. doi : 10.1146/annurev-genet-110410-132547 . ПМЦ 3397683 . ПМИД 21910630 .
- ^ Гольдберг М.Б. (декабрь 2001 г.). «Актиновая подвижность внутриклеточных микробных патогенов» . Обзоры микробиологии и молекулярной биологии . 65 (4): 595–626, оглавление. дои : 10.1128/MMBR.65.4.595-626.2001 . ПМК 99042 . ПМИД 11729265 .
- ^ Кальканьиле М., Тредичи С.М., Тала А., Алифано П. (декабрь 2019 г.). «Бактериальные семиохимические вещества и взаимодействие трансцарства с насекомыми и растениями» . Насекомые . 10 (12): 441. doi : 10.3390/insects10120441 . ПМЦ 6955855 . ПМИД 31817999 .
- ^ Перейти обратно: а б Шапиро Дж. А. (1998). «Думая о бактериальных популяциях как о многоклеточных организмах» (PDF) . Ежегодный обзор микробиологии . 52 : 81–104. дои : 10.1146/аннурев.микро.52.1.81 . ПМИД 9891794 . Архивировано из оригинала (PDF) 17 июля 2011 года.
- ^ Перейти обратно: а б Костертон Дж.В., Левандовски З., Колдуэлл Д.Е., Корбер Д.Р., Лаппин-Скотт Х.М. (1995). «Микробные биопленки». Ежегодный обзор микробиологии . 49 : 711–45. дои : 10.1146/annurev.mi.49.100195.003431 . ПМИД 8561477 .
- ^ Миллер Д.П., Ламонт Р.Дж. (2019). «Сигнальные системы в бактериях полости рта». Иммунитет слизистой оболочки полости рта и микробиом . Достижения экспериментальной медицины и биологии. Том. 1197. стр. 27–43. дои : 10.1007/978-3-030-28524-1_3 . ISBN 978-3-030-28523-4 . ПМИД 31732932 . S2CID 208063186 .
- ^ Абисадо Р.Г., Беномар С., Клаус-младший, Дандекар А.А., Чендлер-младший (май 2018 г.). «Ощущение бактериального кворума и взаимодействие микробного сообщества» . мБио . 9 (3). дои : 10.1128/mBio.02331-17 . ПМЦ 5964356 . ПМИД 29789364 .
- ^ Миллер М.Б., Басслер Б.Л. (2001). «Кворум-чувство у бактерий». Ежегодный обзор микробиологии . 55 : 165–99. дои : 10.1146/annurev.micro.55.1.165 . ПМИД 11544353 . S2CID 1099089 .
- ^ Чжу К., Май У., Пфайффер В., Янссен С., Асникар Ф., Сандерс Дж.Г. и др. (декабрь 2019 г.). «Филогеномика 10 575 геномов выявила эволюционную близость между доменами бактерий и архей» . Природные коммуникации . 10 (1): 5477. Бибкод : 2019NatCo..10.5477Z . дои : 10.1038/s41467-019-13443-4 . ПМК 6889312 . ПМИД 31792218 .
- ^ Баучер И., Дуади С.Дж., Папке Р.Т., Уолш Д.А., Будро М.Е., Несбё К.Л., Кейс Р.Дж., Дулиттл В.Ф. (2003). «Боковой перенос генов и происхождение прокариотических групп». Ежегодный обзор генетики . 37 : 283–328. дои : 10.1146/annurev.genet.37.050503.084247 . ПМИД 14616063 .
- ^ Олсен Г.Дж., Вёзе Ч.Р., Овербик Р. (январь 1994 г.). «Ветры (эволюционных) перемен: вдохнуть новую жизнь в микробиологию» . Журнал бактериологии . 176 (1): 1–6. дои : 10.2172/205047 . ПМК 205007 . ПМИД 8282683 .
- ^ «Дом ИЖСЭМ» . Ijs.sgmjournals.org. 28 октября 2011 года. Архивировано из оригинала 19 октября 2011 года . Проверено 4 ноября 2011 г.
- ^ «Руководство Берджи» . Bergeys.org. Архивировано из оригинала 7 ноября 2011 года . Проверено 4 ноября 2011 г.
- ^ Хедлунд Б.П., Додсворт Дж.А., Стейли Дж.Т. (июнь 2015 г.). «Изменение ландшафта исследования микробного биоразнообразия и его значение для систематики». Систематическая и прикладная микробиология . 38 (4): 231–36. Бибкод : 2015СиАпМ..38..231H . дои : 10.1016/j.syapm.2015.03.003 . ПМИД 25921438 .
- ^ « Шизомицеты ». Медицинский словарь Мерриам-Вебстера. По состоянию на 3 августа 2021 г.
- ^ Браун М.М., Хорсвилл, Арканзас (ноябрь 2020 г.). «Staphylococcus epidermidis – друг или враг кожи?» . ПЛОС Патогены . 16 (11): e1009026. дои : 10.1371/journal.ppat.1009026 . ПМЦ 7660545 . ПМИД 33180890 .
- ^ Гупта РС (2000). «Естественные эволюционные отношения между прокариотами». Критические обзоры по микробиологии . 26 (2): 111–31. CiteSeerX 10.1.1.496.1356 . дои : 10.1080/10408410091154219 . ПМИД 10890353 . S2CID 30541897 .
- ^ Раппе М.С., Джованнони С.Дж. (2003). «Некультивируемое микробное большинство». Ежегодный обзор микробиологии . 57 : 369–94. дои : 10.1146/annurev.micro.57.030502.090759 . ПМИД 14527284 . S2CID 10781051 .
- ^ Дулиттл РФ (июнь 2005 г.). «Эволюционные аспекты полногеномной биологии». Современное мнение в области структурной биологии . 15 (3): 248–53. дои : 10.1016/j.sbi.2005.04.001 . ПМИД 15963888 .
- ^ Кавалер-Смит Т. (январь 2002 г.). «Неомуранское происхождение архебактерий, негибактериальный корень универсального дерева и бактериальная мегаклассификация» . Международный журнал систематической и эволюционной микробиологии . 52 (Часть 1): 7–76. дои : 10.1099/00207713-52-1-7 . ПМИД 11837318 .
- ^ Поммервилль 2014 , с. 15−31.
- ^ Перейти обратно: а б Краснер 2014 , с. 77.
- ^ Вудс Г.Л., Уокер Д.Х. (июль 1996 г.). «Обнаружение инфекции или инфекционных агентов с помощью цитологического и гистологического окрашивания» . Обзоры клинической микробиологии . 9 (3): 382–404. дои : 10.1128/CMR.9.3.382 . ПМК 172900 . ПМИД 8809467 .
- ^ Тан С., Орси Р.Х., Луо Х., Ге С., Чжан Г., Бейкер Р.К., Стивенсон А., Видманн М. (2019). «Оценка и сравнение методов молекулярного подтипирования и характеристики сальмонеллы» . Границы микробиологии . 10 : 1591. дои : 10.3389/fmicb.2019.01591 . ПМК 6639432 . ПМИД 31354679 .
- ^ Краснер 2014 , с. 87–89.
- ^ Член парламента Вайнштейна (март 1994 г.). «Клиническое значение культур крови». Клиники лабораторной медицины . 14 (1): 9–16. дои : 10.1016/S0272-2712(18)30390-1 . ПМИД 8181237 .
- ^ Райли Л.В. (ноябрь 2018 г.). «Лабораторные методы молекулярной эпидемиологии: бактериальные инфекции». Микробиологический спектр . 6 (6). doi : 10.1128/microbiolspec.AME-0004-2018 . ПМИД 30387415 . S2CID 54234977 .
- ^ Ленковски М., Нияковски К., Качмарек М., Сурдачка А. (март 2021 г.). «Техника петлевой изотермической амплификации в пародонтальной диагностике: систематический обзор» . Журнал клинической медицины . 10 (6): 1189. doi : 10.3390/jcm10061189 . ПМК 8000232 . ПМИД 33809163 .
- ^ Ализаде М., Юсефи Л., Пакдел Ф., Готаслу Р., Резаи М.А., Ходадади Э., Оскуэй М.А., Соруш Бархаги М.Х., Кафил Х.С. (2021). «Применение масс-спектроскопии MALDI-TOF в клинической микробиологии» . Достижения фармакологических и фармацевтических наук . 2021 : 9928238. doi : 10.1155/2021/9928238 . ПМЦ 8121603 . ПМИД 34041492 .
- ^ Дудек Н.К., Сан С.Л., Бурштейн Д. (2017). «Новое микробное разнообразие и функциональный потенциал микробиома полости рта морских млекопитающих» (PDF) . Современная биология . 27 (24): 3752–62. Бибкод : 2017CBio...27E3752D . дои : 10.1016/j.cub.2017.10.040 . ПМИД 29153320 . S2CID 43864355 .
- ^ Луи М., Луи Л., Симор А.Е. (август 2000 г.). «Роль технологии амплификации ДНК в диагностике инфекционных заболеваний» . CMAJ . 163 (3): 301–09. дои : 10.1016/s1381-1169(00)00220-x . ПМК 80298 . ПМИД 10951731 . Архивировано из оригинала 14 июня 2006 года.
- ^ Оливер Джей Ди (февраль 2005 г.). «Жизнеспособное, но некультивируемое состояние бактерий» . Журнал микробиологии . 43 Спецификация: 93–100. ПМИД 15765062 . Архивировано из оригинала 28 сентября 2007 года.
- ^ Эзеби JP (8 декабря 2011 г.). «Количество опубликованных имен» . Список названий прокариот, стоящих в номенклатуре . Архивировано из оригинала 19 января 2012 года . Проверено 10 декабря 2011 г.
- ^ Кертис Т.П., Слоан В.Т., Сканнелл Дж.В. (август 2002 г.). «Оценка прокариотического разнообразия и его пределы» . Труды Национальной академии наук Соединенных Штатов Америки . 99 (16): 10494–99. Бибкод : 2002PNAS...9910494C . дои : 10.1073/pnas.142680199 . ПМК 124953 . ПМИД 12097644 .
- ^ Шлосс П.Д., Хандельсман Дж. (декабрь 2004 г.). «Состояние микробной переписи» . Обзоры микробиологии и молекулярной биологии . 68 (4): 686–91. дои : 10.1128/MMBR.68.4.686-691.2004 . ПМК 539005 . ПМИД 15590780 .
- ^ Орен А., генеральный менеджер Гаррити (2021 г.). «Действительная публикация названий сорока двух типов прокариот» . Int J Syst Evol Microbiol . 71 (10): 5056. doi : 10.1099/ijsem.0.005056 . ПМИД 34694987 . S2CID 239887308 .
- ^ Фишер Б., Харви Р.П., Чамп ПК (2007). «Глава 33». Иллюстрированные обзоры Липпинкотта: микробиология (серия иллюстрированных обзоров Липпинкотта) . Хагерствон, доктор медицины: Липпинкотт Уильямс и Уилкинс. стр. 367–92. ISBN 978-0-7817-8215-9 .
- ^ Мушегян А.А., Эберт Д. (январь 2016 г.). «Переосмысление «мутуализма» в разнообразных сообществах симбионтов-хозяев». Биоэссе . 38 (1): 100–8. doi : 10.1002/bies.201500074 . ПМИД 26568407 . S2CID 31661712 .
- ^ Харпер Д. «Комменсализм» . Интернет-словарь этимологии .
- ^ Sears CL (октябрь 2005 г.). «Динамическое партнерство: прославление нашей кишечной флоры». Анаэроб . 11 (5): 247–51. дои : 10.1016/j.anaerobe.2005.05.001 . ПМИД 16701579 .
- ^ Хан Р., Петерсен ФК, Шекхар С. (2019). «Комменсальные бактерии: новый игрок в защите от респираторных патогенов» . Границы в иммунологии . 10 : 1203. дои : 10.3389/fimmu.2019.01203 . ПМК 6554327 . ПМИД 31214175 .
- ^ Роско Д.Л., Чоу А.В. (март 1988 г.). «Нормальная флора и иммунитет слизистых оболочек головы и шеи». Клиники инфекционных заболеваний Северной Америки . 2 (1): 1–19. дои : 10.1016/S0891-5520(20)30163-X . ПМИД 3074102 .
- ^ Шиао С.Л., Кершоу К.М., Лимон Дж.Дж., Ю С., Юн Дж., Ко Е.Ю., Гуарнерио Дж., Потдар А.А., Макговерн Д.П., Бозе С., Дар ТБ, Ноэ П., Ли Дж., Кубота Ю., Майми В.И., Дэвис М.Дж., Хенсон Р.М. , Чой Р.Ю., Ян В., Тан Дж., Гаргус М., Принс А.Д., Зумстег З.С., Андерхилл Д.М. (июль 2021 г.). «Комменсальные бактерии и грибы по-разному регулируют реакцию опухоли на лучевую терапию» . Раковая клетка . 39 (9): 1202–1213.e6. doi : 10.1016/j.ccell.2021.07.002 . ПМЦ 8830498 . ПМИД 34329585 .
- ^ Ляпичев К.А., Ивашкевич Ю., Чернов Ю., Чиненов Д., Шпот Е., Бессонов А.А., Дабая Б.С., Коноплев С. (2021). «MALT-лимфома мочевого пузыря демонстрирует резкое преобладание женщин, неравномерное географическое распространение и возможную инфекционную этиологию» . Исследования и отчеты в области урологии . 13 : 49–62. дои : 10.2147/RRU.S283366 . ПМЦ 7873029 . ПМИД 33575225 .
- ^ Элеян Л., Хан А.А., Мусоллари Г., Чандирамани А.С., Шейх С., Салха А., Тармахомед А., Харки А. (апрель 2021 г.). «Инфекционный эндокардит в детской популяции». Европейский журнал педиатрии . 180 (10): 3089–3100. дои : 10.1007/s00431-021-04062-7 . ПМИД 33852085 . S2CID 233225250 .
- ^ Мартин М.О. (сентябрь 2002 г.). «Хищные прокариоты: новые возможности для исследований». Журнал молекулярной микробиологии и биотехнологии . 4 (5): 467–77. ПМИД 12432957 .
- ^ Велисер Г.Дж., Стредвик К.Л. (август 2002 г.). «Экспериментальная социальная эволюция с Myxococcus xanthus». Антони ван Левенгук . 81 (1–4): 155–64. дои : 10.1023/А:1020546130033 . ПМИД 12448714 . S2CID 20018104 .
- ^ Бауэр А., Форххаммер К. (май 2021 г.). «Бактериальное хищничество цианобактерий» . Микробная физиология . 31 (2): 99–108. дои : 10.1159/000516427 . ISSN 2673-1665 . ПМИД 34010833 .
- ^ Велисер Г.Дж., Мендес-Соареш Х. (январь 2009 г.). «Бактериальные хищники» . Современная биология . 19 (2): R55–56. Бибкод : 2009CBio...19..R55V . дои : 10.1016/j.cub.2008.10.043 . ПМИД 19174136 . S2CID 5432036 .
- ^ Стамс А.Дж., де Бок Ф.А., Плугге СМ, ван Экерт М.Х., Дольфинг Дж., Шраа Г. (март 2006 г.). «Экзоклеточный перенос электронов в анаэробных микробных сообществах». Экологическая микробиология . 8 (3): 371–82. Бибкод : 2006EnvMi...8..371S . дои : 10.1111/j.1462-2920.2006.00989.x . ПМИД 16478444 .
- ^ Зинзер Э.Р. (август 2018 г.). «Перекрестная защита от перекиси водорода с помощью микробов-помощников: воздействие на цианобактерию Prochromococcus и других бенефициаров морских сообществ» . Отчеты по экологической микробиологии . 10 (4): 399–411. Бибкод : 2018EnvMR..10..399Z . дои : 10.1111/1758-2229.12625 . ПМИД 29411546 .
- ^ Бареа Х.М., Посо М.Дж., Азкон Р., Азкон-Агилар К. (июль 2005 г.). «Микробное сотрудничество в ризосфере» . Журнал экспериментальной ботаники . 56 (417): 1761–78. дои : 10.1093/jxb/eri197 . ПМИД 15911555 .
- ^ О'Хара А.М., Шанахан Ф. (июль 2006 г.). «Кишечная флора как забытый орган» . Отчеты ЭМБО . 7 (7): 688–93. дои : 10.1038/sj.embor.7400731 . ПМК 1500832 . ПМИД 16819463 .
- ^ Зотендал Э.Г., Воан Э.Э., де Вос В.М. (март 2006 г.). «Микробный мир внутри нас» . Молекулярная микробиология . 59 (6): 1639–50. дои : 10.1111/j.1365-2958.2006.05056.x . ПМИД 16553872 . S2CID 37602619 .
- ^ Горбач С.Л. (февраль 1990 г.). «Молочнокислые бактерии и здоровье человека». Анналы медицины . 22 (1): 37–41. дои : 10.3109/07853899009147239 . ПМИД 2109988 .
- ^ Салминен С.Дж., Геймонд М., Исолаури Э. (май 2005 г.). «Пробиотики, которые изменяют риск заболеваний» . Журнал питания . 135 (5): 1294–98. дои : 10.1093/jn/135.5.1294 . ПМИД 15867327 .
- ^ Ватанабэ Ф., Бито Т. (январь 2018 г.). «Источники витамина B12 и микробное взаимодействие» . Экспериментальная биология и медицина . 243 (2): 148–58. дои : 10.1177/1535370217746612 . ПМК 5788147 . ПМИД 29216732 .
- ^ Поммервилль, 2014 , стр. 16–21.
- ^ Кларк 2010 , с. 215.
- ^ Уилис 2008 , с. 44.
- ^ Кларк 2010 , стр. 30, 195, 233, 236.
- ^ Миллер А.К., Уильямс С.М. (июль 2021 г.). «Инфекция Helicobacter pylori оказывает как защитное, так и вредное воздействие на здоровье и болезни человека» . Гены и иммунитет . 22 (4): 218–226. дои : 10.1038/s41435-021-00146-4 . ПМЦ 8390445 . ПМИД 34244666 .
- ^ Шварц С., Энне VI, ван Дуйкерен Э. (октябрь 2016 г.). «40 лет ветеринарных работ в JAC – чему мы научились?» . Журнал антимикробной химиотерапии . 71 (10): 2681–90. дои : 10.1093/jac/dkw363 . ПМИД 27660260 .
- ^ Фиш Д.Н. (февраль 2002 г.). «Оптимальная противомикробная терапия сепсиса» . Американский журнал аптеки системы здравоохранения . 59 (Приложение 1): С13–19. дои : 10.1093/ajhp/59.suppl_1.S13 . ПМИД 11885408 .
- ^ Белланд Р.Дж., Уэллетт С.П., Гифферс Дж., Бирн Дж.И. (февраль 2004 г.). «Хламидия пневмония и атеросклероз» . Клеточная микробиология . 6 (2): 117–27. дои : 10.1046/j.1462-5822.2003.00352.x . ПМИД 14706098 . S2CID 45218449 .
- ^ Хейзе Э.Р. (февраль 1982 г.). «Заболевания, связанные с иммуносупрессией» . Перспективы гигиены окружающей среды . 43 : 9–19. дои : 10.2307/3429162 . JSTOR 3429162 . ПМЦ 1568899 . ПМИД 7037390 .
- ^ Сайман Л (2004). «Микробиология ранних заболеваний легких CF». Обзоры детских респираторных заболеваний . 5 (Приложение А): S367–69. дои : 10.1016/S1526-0542(04)90065-6 . ПМИД 14980298 .
- ^ Поммервилль 2014 , с. 118.
- ^ Перейти обратно: а б Поммервилль, 2014 г. , стр. 646–47.
- ^ Краснер 2014 , стр. 165, 369.
- ^ Йонат А., Башан А. (2004). «Рибосомальная кристаллография: инициация, образование пептидных связей и полимеризация аминокислот затрудняются антибиотиками» . Ежегодный обзор микробиологии . 58 : 233–51. дои : 10.1146/annurev.micro.58.030603.123822 . ПМИД 15487937 .
- ^ Хачатурян Г.Г. (ноябрь 1998 г.). «Использование антибиотиков в сельском хозяйстве, эволюция и перенос бактерий, устойчивых к антибиотикам» . CMAJ . 159 (9): 1129–36. ПМЦ 1229782 . ПМИД 9835883 .
- ^ Куо Дж (октябрь 2017 г.). «Процессы дезинфекции» . Исследования водной среды . 89 (10): 1206–44. Бибкод : 2017WaEnR..89.1206K . дои : 10.2175/106143017X15023776270278 . ПМИД 28954657 .
- ^ Джонсон М.Э., Люси Дж.А. (апрель 2006 г.). «Основные технологические достижения и тенденции в области сыра» . Журнал молочной науки . 89 (4): 1174–78. doi : 10.3168/jds.S0022-0302(06)72186-5 . ПМИД 16537950 .
- ^ Краснер 2014 , стр. 25–26.
- ^ Коэн Ю. (декабрь 2002 г.). «Биоремедиация нефти морскими микробными матами» . Международная микробиология . 5 (4): 189–93. дои : 10.1007/s10123-002-0089-5 . ПМИД 12497184 . S2CID 26039323 .
- ^ Невес Л.К., Миямура Т.Т., Мораес Д.А., Пенна Т.К., Конверти А. (2006). «Методы биофильтрации для удаления фенольных остатков». Прикладная биохимия и биотехнология . 129–132 (1–3): 130–52. дои : 10.1385/ABAB:129:1:130 . ПМИД 16915636 . S2CID 189905816 .
- ^ Лизе А., Фильо М.В. (декабрь 1999 г.). «Производство продуктов тонкой химии с использованием биокатализа». Современное мнение в области биотехнологии . 10 (6): 595–603. дои : 10.1016/S0958-1669(99)00040-3 . ПМИД 10600695 .
- ^ Аронсон А.И., Шай Ю. (февраль 2001 г.). «Почему инсектицидные токсины Bacillus thuringiensis так эффективны: уникальные особенности их действия» . Письма FEMS по микробиологии . 195 (1): 1–8. дои : 10.1111/j.1574-6968.2001.tb10489.x . ПМИД 11166987 .
- ^ Божик А (июль 2006 г.). «Восприимчивость взрослых Coccinella septempunctata (Coleoptera: Coccinellidae) к инсектицидам с различным механизмом действия». Наука борьбы с вредителями . 62 (7): 651–54. дои : 10.1002/ps.1221 . ПМИД 16649191 .
- ^ Чаттопадхьяй А., Бхатнагар Н.Б., Бхатнагар Р. (2004). «Бактериальные инсектицидные токсины». Критические обзоры по микробиологии . 30 (1): 33–54. дои : 10.1080/10408410490270712 . ПМИД 15116762 . S2CID 1580984 .
- ^ Серрес М.Х., Гопал С., Нахум Л.А., Лян П., Гаастерланд Т., Райли М. (2001). «Функциональное обновление генома Escherichia coli K-12» . Геномная биология . 2 (9): ИССЛЕДОВАНИЕ0035. doi : 10.1186/gb-2001-2-9-research0035 . ПМК 56896 . ПМИД 11574054 .
- ^ Алмаас Э., Ковач Б., Вичек Т., Олваи З.Н., Барабаши А.Л. (февраль 2004 г.). «Глобальная организация метаболических потоков у бактерии Escherichia coli». Природа . 427 (6977): 839–43. arXiv : q-bio/0403001 . Бибкод : 2004Natur.427..839A . дои : 10.1038/nature02289 . ПМИД 14985762 . S2CID 715721 .
- ^ Рид Дж.Л., Во Т.Д., Шиллинг Ч., Палссон Б.О. (2003). «Модель расширенного генома Escherichia coli K-12 (iJR904 GSM/GPR)» . Геномная биология . 4 (9): Р54. дои : 10.1186/gb-2003-4-9-r54 . ЧВК 193654 . ПМИД 12952533 .
- ^ Уолш Дж. (апрель 2005 г.). «Терапевтические инсулины и их крупнотоннажное производство». Прикладная микробиология и биотехнология . 67 (2): 151–59. дои : 10.1007/s00253-004-1809-x . ПМИД 15580495 . S2CID 5986035 .
- ^ Грауманн К., Премсталлер А (февраль 2006 г.). «Производство рекомбинантных терапевтических белков в микробных системах». Биотехнологический журнал . 1 (2): 164–86. дои : 10.1002/biot.200500051 . ПМИД 16892246 . S2CID 24702839 .
- ^ Рабш В., Хелм Р.А., Эйзенстарк А. (февраль 2004 г.). «Разнообразие типов фагов среди архивных культур Демерецкой коллекции штаммов серовара Salmonella enterica Typhimurium» . Прикладная и экологическая микробиология . 70 (2): 664–69. Бибкод : 2004ApEnM..70..664R . doi : 10.1128/aem.70.2.664-669.2004 . ПМЦ 348941 . ПМИД 14766539 .
- ^ Уилис 2008 .
- ^ Азимов I (1982). Биографическая энциклопедия науки и техники Азимова (2-е изд.). Гарден-Сити, Нью-Йорк: Doubleday and Company. п. 143.
- ^ Поммервилль 2014 , с. 7.
- ^ Эренберг К.Г. (1828). Символы Физио. Животные вечнопозвоночные . Берлин: Декас прима.
- ^ Порода RS, Conn HJ (май 1936 г.). «Статус общего термина Bacterium Ehrenberg 1828» . Журнал бактериологии . 31 (5): 517–18. дои : 10.1128/jb.31.5.517-518.1936 . ПМЦ 543738 . ПМИД 16559906 .
- ^ Эренберг К.Г. (1835). вклад в знание великой организации в направлении наименьшего пространства ( Третий на немецком языке). Берлин: Физические трактаты Королевской академии наук. стр. 143–336.
- ^ «Документы Пастера по теории микробов» . Юридический сайт юридического центра LSU в области медицины и общественного здравоохранения, исторические статьи об общественном здравоохранении. Архивировано из оригинала 18 декабря 2006 года . Проверено 23 ноября 2006 г.
- ^ « Раньше «мыть руки» было спорным медицинским советом» . История . 6 марта 2020 г. Архивировано из оригинала 11 августа 2023 г.
- ^ «Нобелевская премия по физиологии и медицине 1905 года» . Нобелевская премия.org. Архивировано из оригинала 10 декабря 2006 года . Проверено 22 ноября 2006 г.
- ^ О'Брайен С.Дж., Годерт Дж.Дж. (октябрь 1996 г.). «ВИЧ вызывает СПИД: постулаты Коха сбылись» . Современное мнение в иммунологии . 8 (5): 613–18. дои : 10.1016/S0952-7915(96)80075-6 . ПМИД 8902385 .
- ^ Чунг КТ. «Фердинанд Юлиус Кон (1828–1898): пионер бактериологии» (PDF) . Кафедра микробиологии и молекулярно-клеточных наук Университета Мемфиса. Архивировано (PDF) из оригинала 27 июля 2011 года.
- ^ Дрюс, Герхарт (1999). «Фердинанд Кон, основатель современной микробиологии» (PDF) . Новости АСМ . 65 (8): 547–52. Архивировано из оригинала (PDF) 13 июля 2017 года.
- ^ Терстон Эй Джей (декабрь 2000 г.). «О крови, воспалении и огнестрельных ранениях: история борьбы с сепсисом» . Австралийский и новозеландский журнал хирургии . 70 (12): 855–61. дои : 10.1046/j.1440-1622.2000.01983.x . ПМИД 11167573 .
- ^ Шварц Р.С. (март 2004 г.). «Волшебные пули Пауля Эрлиха». Медицинский журнал Новой Англии . 350 (11): 1079–80. дои : 10.1056/NEJMp048021 . ПМИД 15014180 .
- ^ «Жизнеописание Пауля Эрлиха» . Нобелевская премия.org. Архивировано из оригинала 28 ноября 2006 года . Проверено 26 ноября 2006 г.
- ^ Woese CR, Fox GE (ноябрь 1977 г.). «Филогенетическая структура прокариотического домена: первичные царства» . Труды Национальной академии наук Соединенных Штатов Америки . 74 (11): 5088–90. Бибкод : 1977PNAS...74.5088W . дои : 10.1073/pnas.74.11.5088 . ПМК 432104 . ПМИД 270744 .
Библиография
- Кларк Д. (2010). Микробы, гены и цивилизация: как эпидемии сформировали то, кем мы являемся сегодня . Река Аппер-Седл, Нью-Джерси: FT Press. ISBN 978-0-13-701996-0 . OCLC 473120711 .
- Кроуфорд Д. (2007). Смертельные спутники: как микробы сформировали нашу историю . Оксфорд, Нью-Йорк: Издательство Оксфордского университета. ISBN 978-0-19-956144-5 . OCLC 183198723 .
- Зал Б (2008 г.). Эволюция Стрикбергера: интеграция генов, организмов и популяций . Садбери, Массачусетс: Джонс и Бартлетт. ISBN 978-0-7637-0066-9 . OCLC 85814089 .
- Краснер Р. (2014). Микробная проблема: взгляд на общественное здравоохранение . Берлингтон, Массачусетс: Jones & Bartlett Learning. ISBN 978-1-4496-7375-8 . OCLC 794228026 .
- Поммервиль Дж. К. (2014). Основы микробиологии (10-е изд.). Бостон: Джонс и Бартлетт. ISBN 978-1-284-03968-9 .
- Уилис М (2008). Основы современной микробиологии . Садбери, Массачусетс: Издательство Jones and Bartlett. ISBN 978-0-7637-1075-0 . OCLC 67392796 .
