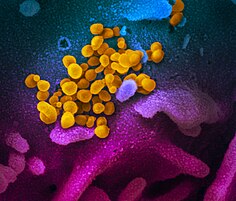
SARS-CoV-2
| Тяжелый острый респираторный синдром коронавирус 2 | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|

| |||||||||||
| Цветная трансмиссионная электронная микрофотография SARS-CoV-2 вирионов с видимыми коронками. | |||||||||||

| |||||||||||
Model of the external structure of the SARS-CoV-2 virion[1]
| |||||||||||
| Virus classification | |||||||||||
| (unranked): | Virus | ||||||||||
| Realm: | Riboviria | ||||||||||
| Kingdom: | Orthornavirae | ||||||||||
| Phylum: | Pisuviricota | ||||||||||
| Class: | Pisoniviricetes | ||||||||||
| Order: | Nidovirales | ||||||||||
| Family: | Coronaviridae | ||||||||||
| Genus: | Betacoronavirus | ||||||||||
| Subgenus: | Sarbecovirus | ||||||||||
| Species: | |||||||||||
| Virus: | Severe acute respiratory syndrome coronavirus 2
| ||||||||||
| Notable variants | |||||||||||
| Synonyms | |||||||||||
| |||||||||||
| Part of a series on the |
| COVID-19 pandemic |
|---|
 |
|
|
|
Тяжелый острый респираторный синдром, коронавирус 2 ( SARS‑CoV‑2 ) [ 2 ] Это штамм коронавируса , который вызывает COVID-19 , респираторное заболевание, ответственное за пандемию COVID-19 . [ 3 ] Ранее вирус имел предварительное название «новый коронавирус 2019 года» ( 2019-nCoV ). [ 4 ] [ 5 ] [ 6 ] [ 7 ] и его также называют человеческим коронавирусом 2019 ( HCoV-19 или hCoV-19 ). [ 8 ] [ 9 ] [ 10 ] [ 11 ] Впервые выявленная в городе Ухань , провинция Хубэй, Китай, Всемирная организация здравоохранения признала вспышку чрезвычайной ситуацией в области общественного здравоохранения, имеющей международное значение , с 30 января 2020 года по 5 мая 2023 года. [ 12 ] [ 13 ] [ 14 ] SARS‑CoV‑2 — вирус с одноцепочечной РНК с положительным смыслом. [ 15 ] это заразно для человека. [ 16 ]
SARS‑CoV‑2 — это штамм вида Betacoronavirus пандемического типа (SARSr-CoV), как и SARS-CoV-1 , вирус, вызвавший вспышку атипичной пневмонии в 2002–2004 годах . [ 2 ] [ 17 ] Существуют штаммы коронавируса, передающиеся от животных, более тесно связанные с SARS-CoV-2, наиболее известным родственником которых является коронавирус летучих мышей BANAL-52. SARS-CoV-2 имеет зоонозное происхождение; его близкое генетическое сходство с коронавирусами летучих мышей позволяет предположить, что он произошел от такого же вируса, передающегося летучими мышами . [18] Research is ongoing as to whether SARS‑CoV‑2 came directly from bats or indirectly through any intermediate hosts.[19] The virus shows little genetic diversity, indicating that the spillover event introducing SARS‑CoV‑2 to humans is likely to have occurred in late 2019.[20]
Epidemiological studies estimate that in the period between December 2019 and September 2020 each infection resulted in an average of 2.4–3.4 new infections when no members of the community were immune and no preventive measures were taken.[21] However, some subsequent variants have become more infectious.[22] The virus is airborne and primarily spreads between people through close contact and via aerosols and respiratory droplets that are exhaled when talking, breathing, or otherwise exhaling, as well as those produced from coughs and sneezes.[23][24] It enters human cells by binding to angiotensin-converting enzyme 2 (ACE2), a membrane protein that regulates the renin–angiotensin system.[25][26]
Terminology

During the initial outbreak in Wuhan, China, various names were used for the virus; some names used by different sources included "the coronavirus" or "Wuhan coronavirus".[27][28] In January 2020, the World Health Organization (WHO) recommended "2019 novel coronavirus" (2019-nCoV)[5][29] as the provisional name for the virus. This was in accordance with WHO's 2015 guidance[30] against using geographical locations, animal species, or groups of people in disease and virus names.[31][32]
On 11 February 2020, the International Committee on Taxonomy of Viruses adopted the official name "severe acute respiratory syndrome coronavirus 2" (SARS‑CoV‑2).[33] To avoid confusion with the disease SARS, the WHO sometimes refers to SARS‑CoV‑2 as "the COVID-19 virus" in public health communications[34][35] and the name HCoV-19 was included in some research articles.[8][9][10] Referring to COVID-19 as the "Wuhan virus" has been described as dangerous by WHO officials, and as xenophobic by many journalists and academics.[36][37][38]
Infection and transmission
This section has multiple issues. Please help improve it or discuss these issues on the talk page. (Learn how and when to remove these template messages)
|
Human-to-human transmission of SARS‑CoV‑2 was confirmed on 20 January 2020 during the COVID-19 pandemic.[16][39][40][41] Transmission was initially assumed to occur primarily via respiratory droplets from coughs and sneezes within a range of about 1.8 metres (6 ft).[42][43] Laser light scattering experiments suggest that speaking is an additional mode of transmission[44][45] and a far-reaching[46] one, indoors, with little air flow.[47][48] Other studies have suggested that the virus may be airborne as well, with aerosols potentially being able to transmit the virus.[49][50][51] During human-to-human transmission, between 200 and 800 infectious SARS‑CoV‑2 virions are thought to initiate a new infection.[52][53][54] If confirmed, aerosol transmission has biosafety implications because a major concern associated with the risk of working with emerging viruses in the laboratory is the generation of aerosols from various laboratory activities which are not immediately recognizable and may affect other scientific personnel.[55] Indirect contact via contaminated surfaces is another possible cause of infection.[56] Preliminary research indicates that the virus may remain viable on plastic (polypropylene) and stainless steel (AISI 304) for up to three days, but it does not survive on cardboard for more than one day or on copper for more than four hours.[10] The virus is inactivated by soap, which destabilizes its lipid bilayer.[57][58] Viral RNA has also been found in stool samples and semen from infected individuals.[59][60]
The degree to which the virus is infectious during the incubation period is uncertain, but research has indicated that the pharynx reaches peak viral load approximately four days after infection[61][62] or in the first week of symptoms and declines thereafter.[63] The duration of SARS-CoV-2 RNA shedding is generally between 3 and 46 days after symptom onset.[64]
A study by a team of researchers from the University of North Carolina found that the nasal cavity is seemingly the dominant initial site of infection, with subsequent aspiration-mediated virus-seeding into the lungs in SARS‑CoV‑2 pathogenesis.[65] They found that there was an infection gradient from high in proximal towards low in distal pulmonary epithelial cultures, with a focal infection in ciliated cells and type 2 pneumocytes in the airway and alveolar regions respectively.[65]
Studies have identified a range of animals—such as cats, ferrets, hamsters, non-human primates, minks, tree shrews, raccoon dogs, fruit bats, and rabbits—that are susceptible and permissive to SARS-CoV-2 infection.[66][67][68] Some institutions have advised that those infected with SARS‑CoV‑2 restrict their contact with animals.[69][70]
Asymptomatic and presymptomatic transmission
On 1 February 2020, the World Health Organization (WHO) indicated that "transmission from asymptomatic cases is likely not a major driver of transmission".[71] One meta-analysis found that 17% of infections are asymptomatic, and asymptomatic individuals were 42% less likely to transmit the virus.[72]
However, an epidemiological model of the beginning of the outbreak in China suggested that "pre-symptomatic shedding may be typical among documented infections" and that subclinical infections may have been the source of a majority of infections.[73] That may explain how out of 217 on board a cruise liner that docked at Montevideo, only 24 of 128 who tested positive for viral RNA showed symptoms.[74] Similarly, a study of ninety-four patients hospitalized in January and February 2020 estimated patients began shedding virus two to three days before symptoms appear and that "a substantial proportion of transmission probably occurred before first symptoms in the index case".[53] The authors later published a correction that showed that shedding began earlier than first estimated, four to five days before symptoms appear.[75]
Reinfection
There is uncertainty about reinfection and long-term immunity.[76] It is not known how common reinfection is, but reports have indicated that it is occurring with variable severity.[76]
The first reported case of reinfection was a 33-year-old man from Hong Kong who first tested positive on 26 March 2020, was discharged on 15 April 2020 after two negative tests, and tested positive again on 15 August 2020 (142 days later), which was confirmed by whole-genome sequencing showing that the viral genomes between the episodes belong to different clades.[77] The findings had the implications that herd immunity may not eliminate the virus if reinfection is not an uncommon occurrence and that vaccines may not be able to provide lifelong protection against the virus.[77]
Another case study described a 25-year-old man from Nevada who tested positive for SARS‑CoV‑2 on 18 April 2020 and on 5 June 2020 (separated by two negative tests). Since genomic analyses showed significant genetic differences between the SARS‑CoV‑2 variant sampled on those two dates, the case study authors determined this was a reinfection.[78] The man's second infection was symptomatically more severe than the first infection, but the mechanisms that could account for this are not known.[78]
Reservoir and origin

No natural reservoir for SARS-CoV-2 has been identified.[79] Prior to the emergence of SARS-CoV-2 as a pathogen infecting humans, there had been two previous zoonosis-based coronavirus epidemics, those caused by SARS-CoV-1 and MERS-CoV.[18]
The first known infections from SARS‑CoV‑2 were discovered in Wuhan, China.[80] The original source of viral transmission to humans remains unclear, as does whether the virus became pathogenic before or after the spillover event.[9][20][81] Because many of the early infectees were workers at the Huanan Seafood Market,[82][83] it has been suggested that the virus might have originated from the market.[9][84] However, other research indicates that visitors may have introduced the virus to the market, which then facilitated rapid expansion of the infections.[20][85] A March 2021 WHO-convened report stated that human spillover via an intermediate animal host was the most likely explanation, with direct spillover from bats next most likely. Introduction through the food supply chain and the Huanan Seafood Market was considered another possible, but less likely, explanation.[86] An analysis in November 2021, however, said that the earliest-known case had been misidentified and that the preponderance of early cases linked to the Huanan Market argued for it being the source.[87]
For a virus recently acquired through a cross-species transmission, rapid evolution is expected.[88] The mutation rate estimated from early cases of SARS-CoV-2 was of 6.54×10−4 per site per year.[86] Coronaviruses in general have high genetic plasticity,[89] but SARS-CoV-2's viral evolution is slowed by the RNA proofreading capability of its replication machinery.[90] For comparison, the viral mutation rate in vivo of SARS-CoV-2 has been found to be lower than that of influenza.[91]
Research into the natural reservoir of the virus that caused the 2002–2004 SARS outbreak has resulted in the discovery of many SARS-like bat coronaviruses, most originating in horseshoe bats. The closest match by far, published in Nature (journal) in February 2022, were viruses BANAL-52 (96.8% resemblance to SARS‑CoV‑2), BANAL-103 and BANAL-236, collected in three different species of bats in Feuang, Laos.[92][93][94] An earlier source published in February 2020 identified the virus RaTG13, collected in bats in Mojiang, Yunnan, China to be the closest to SARS‑CoV‑2, with 96.1% resemblance.[80][95] None of the above are its direct ancestor.[96]

Bats are considered the most likely natural reservoir of SARS‑CoV‑2.[86][97] Differences between the bat coronavirus and SARS‑CoV‑2 suggest that humans may have been infected via an intermediate host;[84] although the source of introduction into humans remains unknown.[98][79]
Although the role of pangolins as an intermediate host was initially posited (a study published in July 2020 suggested that pangolins are an intermediate host of SARS‑CoV‑2-like coronaviruses[99][100]), subsequent studies have not substantiated their contribution to the spillover.[86] Evidence against this hypothesis includes the fact that pangolin virus samples are too distant to SARS-CoV-2: isolates obtained from pangolins seized in Guangdong were only 92% identical in sequence to the SARS‑CoV‑2 genome (matches above 90 percent may sound high, but in genomic terms it is a wide evolutionary gap[101]). In addition, despite similarities in a few critical amino acids,[102] pangolin virus samples exhibit poor binding to the human ACE2 receptor.[103]
Phylogenetics and taxonomy
 Genomic organisation of isolate Wuhan-Hu-1, the earliest sequenced sample of SARS-CoV-2 | |
| NCBI genome ID | 86693 |
|---|---|
| Genome size | 29,903 bases |
| Year of completion | 2020 |
| Genome browser (UCSC) | |
SARS‑CoV‑2 belongs to the broad family of viruses known as coronaviruses.[28] It is a positive-sense single-stranded RNA (+ssRNA) virus, with a single linear RNA segment. Coronaviruses infect humans, other mammals, including livestock and companion animals, and avian species.[104] Human coronaviruses are capable of causing illnesses ranging from the common cold to more severe diseases such as Middle East respiratory syndrome (MERS, fatality rate ~34%). SARS-CoV-2 is the seventh known coronavirus to infect people, after 229E, NL63, OC43, HKU1, MERS-CoV, and the original SARS-CoV.[105]
Like the SARS-related coronavirus implicated in the 2003 SARS outbreak, SARS‑CoV‑2 is a member of the subgenus Sarbecovirus (beta-CoV lineage B).[106][107] Coronaviruses undergo frequent recombination.[108] The mechanism of recombination in unsegmented RNA viruses such as SARS-CoV-2 is generally by copy-choice replication, in which gene material switches from one RNA template molecule to another during replication.[109] The SARS-CoV-2 RNA sequence is approximately 30,000 bases in length,[110] relatively long for a coronavirus—which in turn carry the largest genomes among all RNA families.[111] Its genome consists nearly entirely of protein-coding sequences, a trait shared with other coronaviruses.[108]

A distinguishing feature of SARS‑CoV‑2 is its incorporation of a polybasic site cleaved by furin,[102][112] which appears to be an important element enhancing its virulence.[113] It was suggested that the acquisition of the furin-cleavage site in the SARS-CoV-2 S protein was essential for zoonotic transfer to humans.[114] The furin protease recognizes the canonical peptide sequence RX[R/K] R↓X where the cleavage site is indicated by a down arrow and X is any amino acid.[115][116] In SARS-CoV-2 the recognition site is formed by the incorporated 12 codon nucleotide sequence CCT CGG CGG GCA which corresponds to the amino acid sequence P RR A.[117] This sequence is upstream of an arginine and serine which forms the S1/S2 cleavage site (P RR A R↓S) of the spike protein.[118] Although such sites are a common naturally-occurring feature of other viruses within the Subfamily Orthocoronavirinae,[117] it appears in few other viruses from the Beta-CoV genus,[119] and it is unique among members of its subgenus for such a site.[102] The furin cleavage site PRRAR↓ is highly similar to that of the feline coronavirus, an alphacoronavirus 1 strain.[120]
Viral genetic sequence data can provide critical information about whether viruses separated by time and space are likely to be epidemiologically linked.[121] With a sufficient number of sequenced genomes, it is possible to reconstruct a phylogenetic tree of the mutation history of a family of viruses. By 12 January 2020, five genomes of SARS‑CoV‑2 had been isolated from Wuhan and reported by the Chinese Center for Disease Control and Prevention (CCDC) and other institutions;[110][122] the number of genomes increased to 42 by 30 January 2020.[123] A phylogenetic analysis of those samples showed they were "highly related with at most seven mutations relative to a common ancestor", implying that the first human infection occurred in November or December 2019.[123] Examination of the topology of the phylogenetic tree at the start of the pandemic also found high similarities between human isolates.[124] As of 21 August 2021,[update] 3,422 SARS‑CoV‑2 genomes, belonging to 19 strains, sampled on all continents except Antarctica were publicly available.[125]
On 11 February 2020, the International Committee on Taxonomy of Viruses announced that according to existing rules that compute hierarchical relationships among coronaviruses based on five conserved sequences of nucleic acids, the differences between what was then called 2019-nCoV and the virus from the 2003 SARS outbreak were insufficient to make them separate viral species. Therefore, they identified 2019-nCoV as a virus of Severe acute respiratory syndrome–related coronavirus.[126]
In July 2020, scientists reported that a more infectious SARS‑CoV‑2 variant with spike protein variant G614 has replaced D614 as the dominant form in the pandemic.[127][128]
Coronavirus genomes and subgenomes encode six open reading frames (ORFs).[129] In October 2020, researchers discovered a possible overlapping gene named ORF3d, in the SARS‑CoV‑2 genome. It is unknown if the protein produced by ORF3d has any function, but it provokes a strong immune response. ORF3d has been identified before, in a variant of coronavirus that infects pangolins.[130][131]
Phylogenetic tree
A phylogenetic tree based on whole-genome sequences of SARS-CoV-2 and related coronaviruses is:[132][133]
| SARS‑CoV‑2 related coronavirus |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
SARS-CoV-1, 79% to SARS-CoV-2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Variants
This section needs to be updated. (April 2023) |

There are many thousands of variants of SARS-CoV-2, which can be grouped into the much larger clades.[142] Several different clade nomenclatures have been proposed. Nextstrain divides the variants into five clades (19A, 19B, 20A, 20B, and 20C), while GISAID divides them into seven (L, O, V, S, G, GH, and GR).[143]
Several notable variants of SARS-CoV-2 emerged in late 2020. The World Health Organization has currently declared five variants of concern, which are as follows:[144]
- Alpha: Lineage B.1.1.7 emerged in the United Kingdom in September 2020, with evidence of increased transmissibility and virulence. Notable mutations include N501Y and P681H.
- An E484K mutation in some lineage B.1.1.7 virions has been noted and is also tracked by various public health agencies.
- Beta: Lineage B.1.351 emerged in South Africa in May 2020, with evidence of increased transmissibility and changes to antigenicity, with some public health officials raising alarms about its impact on the efficacy of some vaccines. Notable mutations include K417N, E484K and N501Y.
- Gamma: Lineage P.1 emerged in Brazil in November 2020, also with evidence of increased transmissibility and virulence, alongside changes to antigenicity. Similar concerns about vaccine efficacy have been raised. Notable mutations also include K417N, E484K and N501Y.
- Delta: Lineage B.1.617.2 emerged in India in October 2020. There is also evidence of increased transmissibility and changes to antigenicity.
- Omicron: Lineage B.1.1.529 emerged in Botswana in November 2021.
Other notable variants include 6 other WHO-designated variants under investigation and Cluster 5, which emerged among mink in Denmark and resulted in a mink euthanasia campaign rendering it virtually extinct.[145]
Virology
Virus structure

Each SARS-CoV-2 virion is 60–140 nanometres (2.4×10−6–5.5×10−6 in) in diameter;[105][83] its mass within the global human populace has been estimated as being between 0.1 and 10 kilograms.[146] Like other coronaviruses, SARS-CoV-2 has four structural proteins, known as the S (spike), E (envelope), M (membrane), and N (nucleocapsid) proteins; the N protein holds the RNA genome, and the S, E, and M proteins together create the viral envelope.[147] Coronavirus S proteins are glycoproteins and also type I membrane proteins (membranes containing a single transmembrane domain oriented on the extracellular side).[114] They are divided into two functional parts (S1 and S2).[104] In SARS-CoV-2, the spike protein, which has been imaged at the atomic level using cryogenic electron microscopy,[148][149] is the protein responsible for allowing the virus to attach to and fuse with the membrane of a host cell;[147] specifically, its S1 subunit catalyzes attachment, the S2 subunit fusion.[150]
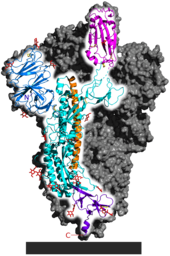
Genome
As of early 2022, about 7 million SARS-CoV-2 genomes had been sequenced and deposited into public databases and another 800,000 or so were added each month.[151] By September 2023, the GISAID EpiCoV database contained more than 16 million genome sequences.[152]
SARS-CoV-2 имеет линейный с положительным смыслом длиной около 30 000 оснований. геном с одноцепочечной РНК [104] Its genome has a bias against cytosine (C) and guanine (G) nucleotides, like other coronaviruses.[ 153 ] Геном имеет самый высокий состав U (32,2%), за ним следует A (29,9%) и аналогичный состав G (19,6%) и C (18,3%). [ 154 ] возникает Смещение нуклеотидов в результате мутации гуанинов и цитозинов на аденозины и урацилы соответственно. [ 155 ] мутация динуклеотидов CG Считается, что противовирусным белком цинковых пальцев . возникает, чтобы обойти защитный механизм клеток, связанный с [ 156 ] и снизить энергию для разъединения генома во время репликации и трансляции ( пара оснований аденозина и урацила через две водородные связи , цитозин и гуанин через три). [ 155 ] Истощение динуклеотидов CG в его геноме привело к тому, что вирус стал иметь заметную предвзятость в использовании кодонов . Например, шесть различных кодонов аргинина имеют относительное синонимическое использование кодонов : AGA (2,67), CGU (1,46), AGG (0,81), CGC (0,58), CGA (0,29) и CGG (0,19). [ 154 ] Аналогичная тенденция к смещению использования кодонов наблюдается и у других коронавирусов, связанных с атипичной пневмонией. [ 157 ]
Цикл репликации
Вирусные инфекции начинаются, когда вирусные частицы связываются с поверхностными клеточными рецепторами хозяина. [ 158 ] Эксперименты по моделированию белка шипового белка вируса вскоре показали, что SARS-CoV-2 обладает достаточным сродством к рецептору ангиотензинпревращающего фермента 2 (ACE2) на клетках человека, чтобы использовать его в качестве механизма проникновения в клетку . [ 159 ] К 22 января 2020 года группа в Китае, работающая с полным геномом вируса, и группа в США, использующая методы обратной генетики независимо и экспериментально, продемонстрировали, что ACE2 может действовать как рецептор SARS-CoV-2. [ 80 ] [ 160 ] [ 161 ] [ 162 ] Исследования показали, что SARS‑CoV‑2 имеет более высокое сродство к ACE2 человека, чем исходный вирус SARS. [ 148 ] [ 163 ] SARS-CoV-2 также может использовать базигин для облегчения проникновения в клетку. [ 164 ]
Начальное праймирование шипового белка трансмембранной протеазой серином 2 (TMPRSS2) необходимо для проникновения SARS-CoV-2. [ 25 ] Белок-хозяин нейропилин 1 (NRP1) может помочь вирусу проникнуть в клетку-хозяина с помощью ACE2. [ 165 ] После того, как вирион SARS-CoV-2 прикрепляется к клетке-мишени, клеточный TMPRSS2 разрезает шиповый белок вируса, обнажая слитый пептид в субъединице S2 и рецептор ACE2 хозяина. [ 150 ] После слияния вокруг вириона образуется эндосома , отделяющая его от остальной части клетки-хозяина. Вирион ускользает, когда pH эндосомы падает или когда катепсин протеаза хозяина , цистеиновая , расщепляет его. [ 150 ] Затем вирион высвобождает РНК в клетку и заставляет клетку производить и распространять копии вируса , которые заражают больше клеток. [ 166 ]
SARS-CoV-2 производит как минимум три фактора вирулентности , которые способствуют выделению новых вирионов из клеток-хозяев и подавляют иммунный ответ . [ 147 ] Включают ли они пониженную регуляцию ACE2, как это наблюдается у аналогичных коронавирусов, еще предстоит выяснить (по состоянию на май 2020 года). [ 167 ]
Лечение и разработка лекарств
Известно, что очень немногие лекарства эффективно ингибируют SARS‑CoV‑2. Было обнаружено, что маситиниб ингибирует основную протеазу SARS-CoV-2 , демонстрируя более чем 200-кратное снижение титров вируса в легких и носу мышей, однако он не одобрен для лечения COVID-19 у людей. [ 168 ] [ нужно обновить ] В декабре 2021 года Соединенные Штаты выдали разрешение на экстренное применение нирмарелвира /ритонавира для лечения вируса; [ 169 ] Европейский Союз , Великобритания и Канада, получив полное разрешение. Вскоре после этого этому примеру последовали [ 170 ] [ 171 ] [ 172 ] Одно исследование показало, что нирмарелвир/ритонавир снижает риск госпитализации и смерти на 88%. [ 173 ]
COVID Moonshot — это международный совместный открытый научный проект, начатый в марте 2020 года с целью разработки незапатентованного перорального противовирусного препарата для лечения SARS-CoV-2. [ 174 ]
Эпидемиология
Ретроспективные тесты, собранные в рамках китайской системы эпиднадзора, не выявили явных признаков значительной нераспознанной циркуляции SARS‑CoV‑2 в Ухане во второй половине 2019 года. [ 86 ]
Метаанализ от ноября 2020 года оценил базовую репродуктивную численность ( ) вируса составляет от 2,39 до 3,44. [ 21 ] Это означает, что каждое заражение вирусом, как ожидается, приведет к 2,39–3,44 новым инфекциям, если ни один член сообщества не имеет иммунитета и не профилактические меры принимаются . Число воспроизводства может быть выше в густонаселенных условиях, например, на круизных лайнерах . [ 175 ] Поведение человека влияет на значение R0, и, следовательно, оценки R0 различаются в разных странах, культурах и социальных нормах. Например, одно исследование выявило относительно низкие значения R0 (~3,5) в Швеции, Бельгии и Нидерландах, тогда как в Испании и США значения R0 были значительно выше (от 5,9 до 6,4 соответственно). [ 176 ]
| Вариант | Р0 | Источник |
|---|---|---|
| Эталонный/предковый штамм | ~2.8 | [ 177 ] |
| Альфа (Б.1.1.7) | (на 40-90% выше, чем в предыдущих вариантах) | [ 178 ] |
| Дельта (Б.1.617.2) | ~5 (3-8) | [ 179 ] |
В материковом Китае зарегистрировано около 96 000 подтвержденных случаев заражения. [ 180 ] Хотя доля инфекций, которые приводят к подтвержденным случаям или развитию диагностируемого заболевания, остается неясной, [ 181 ] По оценкам одной математической модели, 25 января 2020 года только в Ухане 75 815 человек были инфицированы, в то время как число подтвержденных случаев во всем мире составляло всего 2015. [ 182 ] До 24 февраля 2020 года более 95% всех смертей от COVID-19 в мире приходилось на провинцию Хубэй , где расположен Ухань. [ 183 ] [ 184 ] По состоянию на 10 марта 2023 года процент снизился до 0,047%. [ 180 ]
По состоянию на 10 марта 2023 года общее количество подтвержденных случаев заражения SARS‑CoV‑2 составило 676 609 955 человек. [ 180 ] Общее число смертей от вируса составило 6 881 955 человек. [ 180 ]
См. также
- 3C-подобная протеаза (NS5)
Ссылки
- ^ Solodovnikov A, Arkhipova V (29 July 2021). "Достоверно красиво: как мы сделали 3D-модель SARS-CoV-2" [Truly beautiful: how we made the SARS-CoV-2 3D model] (in Russian). N+1 . Archived from the original on 30 July 2021 . Retrieved 30 July 2021 .
- ^ Циммер С (26 февраля 2021 г.). «Тайная жизнь коронавируса. Маслянистый пузырь генов шириной 100 нанометров убил более двух миллионов человек и изменил мир. Ученые не совсем знают, что с этим делать» . Нью-Йорк Таймс . Архивировано из оригинала 27 февраля 2021 года . Проверено 28 февраля 2021 г.
- ^ Определения случаев эпиднадзора за заражением человека новым коронавирусом (нКоВ): временное руководство, версия 1, январь 2020 г. (Отчет). Всемирная организация здравоохранения. Январь 2020 г. HDL : 10665/330376 . ВОЗ/2019-nCoV/Эпиднадзор/v2020.1.
- ^ Перейти обратно: а б «Медицинские работники: часто задаваемые вопросы и ответы» . Центры США по контролю и профилактике заболеваний (CDC) . 11 февраля 2020 года. Архивировано из оригинала 14 февраля 2020 года . Проверено 15 февраля 2020 г.
- ^ «О новом коронавирусе (2019-nCoV)» . Центры США по контролю и профилактике заболеваний (CDC) . 11 февраля 2020 года. Архивировано из оригинала 11 февраля 2020 года . Проверено 25 февраля 2020 г.
- ^ Хармон А (4 марта 2020 г.). «Мы поговорили с шестью американцами, зараженными коронавирусом» . Нью-Йорк Таймс . Архивировано из оригинала 13 марта 2020 года . Проверено 16 марта 2020 г.
- ^ Перейти обратно: а б Вонг Джи, Би Ю.Х., Ван К.Х., Чен С.В., Чжан З.Г., Яо Ю.Г. (май 2020 г.). «Зоонозное происхождение человеческого коронавируса 2019 (HCoV-19/SARS-CoV-2): почему важна эта работа?» . Зоологические исследования . 41 (3): 213–219. дои : 10.24272/j.issn.2095-8137.2020.031 . ПМЦ 7231470 . ПМИД 32314559 .
- ^ Перейти обратно: а б с д Андерсен К.Г., Рамбо А., Липкин В.И., Холмс Э.К., Гарри РФ (апрель 2020 г.). «Проксимальное происхождение SARS-CoV-2» . Природная медицина . 26 (4): 450–452. дои : 10.1038/s41591-020-0820-9 . ПМК 7095063 . ПМИД 32284615 .
- ^ Перейти обратно: а б с ван Доремален Н., Бушмейкер Т., Моррис Д.Х., Холбрук М.Г., Гэмбл А., Уильямсон Б.Н., Тамин А., Харкорт Дж.Л., Торнбург Нью-Джерси, Гербер С.И., Ллойд-Смит Дж.О., де Вит Э., Мюнстер В.Дж. (апрель 2020 г.). «Аэрозольная и поверхностная стабильность SARS-CoV-2 по сравнению с SARS-CoV-1» . Медицинский журнал Новой Англии . 382 (16): 1564–1567. дои : 10.1056/NEJMc2004973 . ПМЦ 7121658 . ПМИД 32182409 .
- ^ «База данных hCoV-19» . Китайский национальный банк генов. Архивировано из оригинала 17 июня 2020 года . Проверено 2 июня 2020 г.
- ^ Заявление по поводу второго заседания Комитета по чрезвычайной ситуации Международных медико-санитарных правил (2005 г.) в связи со вспышкой нового коронавируса (2019-nCoV) . Всемирная организация здравоохранения (ВОЗ) . 30 января 2020 года. Архивировано из оригинала 31 января 2020 года . Проверено 30 января 2020 г.
- ^ Вступительное слово Генерального директора ВОЗ на брифинге для СМИ по COVID-19 – 11 марта 2020 г. Всемирная организация здравоохранения (ВОЗ) . 11 марта 2020 г. Архивировано из оригинала 11 марта 2020 г. . Проверено 12 марта 2020 г.
- ^ Ригби Дж., Сатия Б. (5 мая 2023 г.). «ВОЗ объявляет о прекращении глобальной чрезвычайной ситуации в области здравоохранения, вызванной COVID» . Рейтер . Проверено 6 мая 2023 г.
- ^ Мачи Дж., Херсковиц Дж., Сенан А.М., Дутта Д., Нат Б., Олейников М.Д., Бломберг В.Р., Мейгс Д.Д., Хасан М., Патель М., Клайн П., Чанг Р.К., Чанг Л., Гендельман Х.Э., Кевадия Б.Д. (сентябрь 2020 г.). «Естественное течение, патобиология и клинические проявления инфекций SARS-CoV-2» . Журнал нейроиммунной фармакологии . 15 (3): 359–386. дои : 10.1007/s11481-020-09944-5 . ПМЦ 7373339 . ПМИД 32696264 .
- ^ Перейти обратно: а б Чан Дж.Ф., Юань С., Кок К.Х., То К.К., Чу Х., Ян Дж., Син Ф., Лю Дж., Ип CC, Пун Р.В., Цой Х.В., Ло СК, Чан К.Х., Пун В.К., Чан В.М., Ип Дж.Д., Цай Дж.П. , Ченг В.К., Чен Х., Хуэй К.К., Юэнь К.Ю. (февраль 2020 г.). «Семейный кластер пневмонии, связанный с новым коронавирусом 2019 года, указывающий на передачу от человека к человеку: исследование семейного кластера» . Ланцет . 395 (10223): 514–523. дои : 10.1016/S0140-6736(20)30154-9 . ПМК 7159286 . ПМИД 31986261 .
- ^ «Новый коронавирус устойчив в течение нескольких часов на поверхностях» . Национальные институты здравоохранения (NIH) . NIH.gov. 17 марта 2020 г. Архивировано из оригинала 23 марта 2020 г. . Проверено 4 мая 2020 г.
- ^ Перейти обратно: а б Вьковски П., Крацель А., Штайнер С., Сталдер Х., Тиль В. (март 2021 г.). «Биология и репликация коронавируса: последствия для SARS-CoV-2» . Nat Rev Microbiol (обзор). 19 (3): 155–170. дои : 10.1038/s41579-020-00468-6 . ПМЦ 7592455 . ПМИД 33116300 .
- ^ Новый коронавирус (2019-nCoV): отчет о ситуации, 22 (Отчет). Всемирная организация здравоохранения . 11 февраля 2020 г. HDL : 10665/330991 .
- ^ Перейти обратно: а б с Коэн Дж. (январь 2020 г.). «Рынок морепродуктов в Ухане не может быть источником нового вируса, распространяющегося по всему миру». Наука . дои : 10.1126/science.abb0611 . S2CID 214574620 .
- ^ Перейти обратно: а б Биллах М.А., Миа М.М., Хан М.Н. (11 ноября 2020 г.). «Репродуктивное число коронавируса: систематический обзор и метаанализ, основанный на доказательствах глобального уровня» . ПЛОС ОДИН . 15 (11): e0242128. Бибкод : 2020PLoSO..1542128B . дои : 10.1371/journal.pone.0242128 . ПМЦ 7657547 . ПМИД 33175914 .
- ^ «Варианты COVID-19: в чем проблема?» . Клиника Мэйо . 27 августа 2022 г. Проверено 10 октября 2022 г.
- ^ « Как распространяется коронавирус. Архивировано 3 апреля 2020 года в Wayback Machine », Центры по контролю и профилактике заболеваний, дата обращения 14 мая 2021 года.
- ^ « Коронавирусное заболевание (COVID-19): как оно передается? Архивировано 15 октября 2020 г. в Wayback Machine », Всемирная организация здравоохранения.
- ^ Перейти обратно: а б Хоффманн М., Кляйне-Вебер Х., Шрёдер С., Крюгер Н., Херрлер Т., Эриксен С., Ширгенс Т.С., Херрлер Г., Ву Н.Х., Ниче А., Мюллер М.А., Дростен С., Полманн С. (апрель 2020 г.). «Проникновение в клетку SARS-CoV-2 зависит от ACE2 и TMPRSS2 и блокируется клинически проверенным ингибитором протеазы» . Клетка . 181 (2): 271–280.e8. doi : 10.1016/j.cell.2020.02.052 . ПМЦ 7102627 . ПМИД 32142651 .
- ^ Чжао П., Прайсман Дж.Л., Грант О.К., Цай Ю., Сяо Т., Розенбальм К.Е., Аоки К., Келлман Б.П., Бриджер Р., Баруш Д.Х., Бриндли М.А., Льюис Н.Е., Тимейер М., Чен Б., Вудс Р.Дж., Уэллс Л. (октябрь 2020 г.) ). «Вирусно-рецепторные взаимодействия гликозилированного спайка SARS-CoV-2 и человеческого рецептора ACE2» . Клетка-хозяин и микроб . 28 (4): 586–601.e6. дои : 10.1016/j.chom.2020.08.004 . ПМЦ 7443692 . ПМИД 32841605 .
- ^ Хуан П. (22 января 2020 г.). «Чем уханьский коронавирус сравнивается с MERS, SARS и простудой?» . ЭНЕРГЕТИЧЕСКИЙ ЯДЕРНЫЙ РЕАКТОР . Архивировано из оригинала 2 февраля 2020 года . Проверено 3 февраля 2020 г. .
- ^ Перейти обратно: а б Фокс Д. (январь 2020 г.). «Что нужно знать о новом коронавирусе». Природа . дои : 10.1038/d41586-020-00209-y . ПМИД 33483684 . S2CID 213064026 .
- ^ Всемирная организация здравоохранения (30 января 2020 г.). Новый коронавирус (2019-nCoV): отчет о ситуации, 10 (Отчет). Всемирная организация здравоохранения . hdl : 10665/330775 .
- ^ «Передовой опыт Всемирной организации здравоохранения по присвоению названий новым инфекционным заболеваниям человека» (PDF) . ВОЗ . Май 2015 г. Архивировано (PDF) из оригинала 12 февраля 2020 г.
- ^ «Новый коронавирус под названием Covid-19: ВОЗ» . СЕГОДНЯонлайн. Архивировано из оригинала 21 марта 2020 года . Проверено 11 февраля 2020 г.
- ^ «Коронавирус распространяет расизм против этнических китайцев и среди них» . Экономист . 17 февраля 2020 года. Архивировано из оригинала 17 февраля 2020 года . Проверено 17 февраля 2020 г.
- ^ «Наименование коронавирусной болезни (COVID-2019) и вируса, ее вызывающего» . Всемирная организация здравоохранения. Архивировано из оригинала 28 февраля 2020 года . Проверено 14 декабря 2020 г.
ICTV объявила «коронавирус тяжелого острого респираторного синдрома 2 (SARS-CoV-2)» в качестве названия нового вируса 11 февраля 2020 года. Это название было выбрано, поскольку вирус генетически связан с коронавирусом, ответственным за вспышку атипичной пневмонии в 2003 году. Несмотря на то, что эти два вируса связаны, они различны.
- ^ Хуэй М (18 марта 2020 г.). «Почему ВОЗ не называет коронавирус своим именем — SARS-CoV-2?» . Кварц . Архивировано из оригинала 25 марта 2020 года . Проверено 26 марта 2020 г. .
- ^ «Наименование коронавирусной болезни (COVID-2019) и вируса, ее вызывающего» . Всемирная организация здравоохранения. Архивировано из оригинала 28 февраля 2020 года . Проверено 14 декабря 2020 г.
С точки зрения информирования о рисках использование названия SARS может иметь непредвиденные последствия с точки зрения создания ненужного страха у некоторых групп населения. ... По этой и другим причинам ВОЗ начала называть этот вирус «вирусом, ответственным за COVID-19» или «вирусом COVID-19» при общении с общественностью. Ни одно из этих обозначений [ sic ] не предназначено для замены официального названия вируса, согласованного ICTV.
- ^ «Противодействие ненависти и предвзятости, связанным с COVID-19» . Национальная ассоциация образования. 5 июня 2020 года.
Это расизм и порождает ксенофобию», — заявил The Washington Post преподаватель азиатско-американских исследований Калифорнийского университета в Беркли Харви Донг . «Это очень опасная ситуация».
- ^ Гсталтер М (19 марта 2020 г.). «Сотрудник ВОЗ предостерегает от названия этого «китайского вируса», говорит, что «в этом нет никакой вины» » . Холм . Проверено 15 сентября 2022 г.
Райан не первый чиновник ВОЗ, выступающий против этой фразы. Генеральный директор Тедрос Адханом Гебрейесус заявил ранее в этом месяце, что этот термин «болезненно видеть» и «более опасен, чем сам вирус».
- ^ Говер А.Р., Харпер С.Б., Лэнгтон Л. (июль 2020 г.). «Преступления на почве ненависти против Азии во время пандемии COVID-19: изучение воспроизводства неравенства» . Американский журнал уголовного правосудия . 45 (4): 647–667. дои : 10.1007/s12103-020-09545-1 . ПМЦ 7364747 . ПМИД 32837171 .
- ^ Ли JY, Ю Z, Ван Q, Чжоу ZJ, Цю Y, Луо Р, Ge XY (март 2020 г.). «Эпидемия пневмонии, вызванной новым коронавирусом 2019 года (2019-nCoV), и понимание новых инфекционных заболеваний в будущем» . Микробы и инфекции . 22 (2): 80–85. doi : 10.1016/j.micinf.2020.02.002 . ПМК 7079563 . ПМИД 32087334 .
- ^ Кесслер Г. (17 апреля 2020 г.). «Ложное заявление Трампа о том, что ВОЗ заявила, что коронавирус «не заразен» » . Вашингтон Пост . Архивировано из оригинала 17 апреля 2020 года . Проверено 17 апреля 2020 г.
- ^ Куо Л (21 января 2020 г.). «Китай подтверждает передачу коронавируса от человека к человеку» . Хранитель . Архивировано из оригинала 22 марта 2020 года . Проверено 18 апреля 2020 г.
- ^ «Как распространяется COVID-19» . Центры США по контролю и профилактике заболеваний (CDC) . 27 января 2020 года. Архивировано из оригинала 28 января 2020 года . Проверено 29 января 2020 г.
- ^ Эдвардс Э. (25 января 2020 г.). «Как распространяется коронавирус?» . Новости Эн-Би-Си . Архивировано из оригинала 28 января 2020 года . Проверено 13 марта 2020 г.
- ^ Анфинруд П., Стадницкий В., Бакс CE, Бакс А (май 2020 г.). «Визуализация капель ротовой жидкости, генерируемых речью, с помощью рассеяния лазерного света» . Медицинский журнал Новой Англии . 382 (21): 2061–2063. дои : 10.1056/NEJMc2007800 . ПМК 7179962 . ПМИД 32294341 .
- ^ Стадницкий В., Бакс С.Э., Бакс А., Анфинруд П. (июнь 2020 г.). «Время жизни небольших речевых капель и их потенциальная роль в передаче SARS-CoV-2» . Труды Национальной академии наук Соединенных Штатов Америки . 117 (22): 11875–11877. Бибкод : 2020PNAS..11711875S . дои : 10.1073/pnas.2006874117 . ПМЦ 7275719 . ПМИД 32404416 .
- ^ Кломпас М., Бейкер М.А., Ри С. (август 2020 г.). «Передача SARS-CoV-2 воздушно-капельным путем: теоретические соображения и имеющиеся данные» . ДЖАМА . 324 (5): 441–442. дои : 10.1001/jama.2020.12458 . ПМИД 32749495 . S2CID 220500293 .
Исследователи продемонстрировали, что разговор и кашель образуют смесь капель и аэрозолей различных размеров, что эти выделения могут перемещаться вместе на расстояние до 27 футов, что SARS-CoV-2 может оставаться во взвешенном состоянии в воздухе и жизнеспособность в течение нескольких часов, что РНК SARS-CoV-2 можно выделить из проб воздуха в больницах и что плохая вентиляция продлевает время, в течение которого аэрозоли остаются в воздухе.
- ^ Реттнер Р. (21 января 2021 г.). «Разговор хуже, чем кашель, поскольку способствует распространению COVID-19 в помещении» . Живая наука . Проверено 10 октября 2022 г.
В одном смоделированном сценарии исследователи обнаружили, что после короткого кашля количество инфекционных частиц в воздухе быстро снижается через 1–7 минут; напротив, после 30-секундного разговора только через 30 минут количество инфекционных частиц упадет до аналогичного уровня; и большое количество частиц все еще оставалось во взвешенном состоянии через час. Другими словами, доза вирусных частиц, способных вызвать инфекцию, будет задерживаться в воздухе после речи гораздо дольше, чем после кашля. (В этом смоделированном сценарии за 0,5-секундный кашель попадало такое же количество капель, как и за 30-секундную речь.)
- ^ де Оливейра П.М., Мескита Л.К., Гкантонас С., Джусти А., Масторакос Э. (январь 2021 г.). «Эволюция спреев и аэрозолей из дыхательных путей: теоретические оценки для понимания передачи вируса» . Труды Королевского общества A: Математические, физические и технические науки . 477 (2245): 20200584. Бибкод : 2021RSPSA.47700584D . дои : 10.1098/rspa.2020.0584 . ПМЦ 7897643 . ПМИД 33633490 . S2CID 231643585 .
- ^ Мандавилли А (4 июля 2020 г.). «239 экспертов с одним большим утверждением: коронавирус передается воздушно-капельным путем. ВОЗ сопротивляется растущим доказательствам того, что вирусные частицы, плавающие в помещении, заразны, говорят некоторые ученые. Агентство утверждает, что исследования все еще неубедительны» . Нью-Йорк Таймс . Архивировано из оригинала 17 ноября 2020 года . Проверено 5 июля 2020 г.
- ^ Туфекчи З. (30 июля 2020 г.). «Надо поговорить о вентиляции» . Атлантика . Архивировано из оригинала 17 ноября 2020 года . Проверено 8 сентября 2020 г.
- ^ Льюис Д. (июль 2020 г.). «Все больше данных свидетельствуют о том, что коронавирус передается воздушно-капельным путем, но рекомендации по здравоохранению не догнали эту ситуацию» . Природа . 583 (7817): 510–513. Бибкод : 2020Natur.583..510L . дои : 10.1038/d41586-020-02058-1 . ПМИД 32647382 . S2CID 220470431 .
- ^ Попа А, Генгер Дж.В., Николсон М.Д., Пенц Т., Шмид Д., Аберле С.В., Агерер Б., Лерчер А., Эндлер Л., Колачо Х., Смит М., Шустер М., Грау М.Л., Мартинес-Хименес Ф., Пич О., Борена В., Павелка Е, Кесей З, Сенекович М, Лайне Дж, Аберле Дж. Х., Редльбергер-Фритц М., Кароли М, Зуфали А, Маричник С., Борковец М., Хуфнагль П., Наирц М., Вайс Г., Вольфингер М.Т., фон Лаер Д., Суперти- Фурга Г., Лопес-Бигас Н., Пуххаммер-Штокль Е., Аллербергер Ф., Михор Ф., Бок Дж., Бергталер А. (декабрь 2020 г.). «Геномная эпидемиология событий сверхраспространения в Австрии раскрывает мутационную динамику и свойства передачи SARS-CoV-2» . Наука трансляционной медицины . 12 (573): eabe2555. doi : 10.1126/scitranslmed.abe2555 . ПМЦ 7857414 . ПМИД 33229462 .
- ^ Перейти обратно: а б Хэ X, Лау Э.Х., Ву П, Дэн X, Ван J, Хао X, Лау YC, Вонг JY, Гуань Y, Тан X, Мо X, Чэнь Y, Ляо Б, Чен В, Ху Ф, Чжан Q, Чжун М , Ву Ю, Чжао Л., Чжан Ф., Коулинг Б.Дж., Ли Ф., Люн Г.М. (май 2020 г.). «Временная динамика выделения вируса и трансмиссивности COVID-19» . Природная медицина . 26 (5): 672–675. дои : 10.1038/s41591-020-0869-5 . ПМИД 32296168 .
- ^ Ватанабэ Т., Бартранд Т.А., Вейр М.Х., Омура Т., Хаас К.Н. (июль 2010 г.). «Разработка модели «доза-реакция» для коронавируса SARS» . Анализ рисков . 30 (7): 1129–38. Бибкод : 2010РискА..30.1129Вт . дои : 10.1111/j.1539-6924.2010.01427.x . ПМЦ 7169223 . ПМИД 20497390 .
- ^ Артика И.М., Маруф К.Н. (май 2017 г.). «Лабораторная биобезопасность при обращении с новыми вирусами» . Азиатско-Тихоокеанский журнал тропической биомедицины . 7 (5): 483–491. дои : 10.1016/j.apjtb.2017.01.020 . ПМК 7103938 . ПМИД 32289025 .
- ^ «Подготовка рабочего места к COVID-19» (PDF) . Всемирная организация здравоохранения . 27 февраля 2020 г. Архивировано (PDF) из оригинала 2 марта 2020 г. . Проверено 3 марта 2020 г.
- ^ Ён Э (20 марта 2020 г.). «Почему коронавирус оказался таким успешным» . Атлантика . Архивировано из оригинала 20 марта 2020 года . Проверено 20 марта 2020 г.
- ^ Гиббенс С. (18 марта 2020 г.). «Почему мыло предпочтительнее отбеливать при борьбе с коронавирусом» . Нэшнл Географик . Архивировано из оригинала 2 апреля 2020 года . Проверено 2 апреля 2020 г.
- ^ Холшу М.Л., ДеБолт С., Линдквист С., Лофи К.Х., Висман Дж., Брюс Х., Спиттерс С., Эриксон К., Вилкерсон С., Турал А., Диас Дж., Кон А., Фокс Л., Патель А., Гербер С.И., Ким Л., Тонг С. , Лу Х, Линдстрем С., Палланш М.А., Уэлдон В.К., Биггс Х.М., Уеки Т.М., Пиллаи С.К. (март 2020 г.). «Первый случай нового коронавируса 2019 года в США» . Медицинский журнал Новой Англии . 382 (10): 929–936. дои : 10.1056/NEJMoa2001191 . ПМК 7092802 . ПМИД 32004427 .
- ^ Ли Д., Цзинь М., Бао П., Чжао В., Чжан С. (май 2020 г.). «Клинические характеристики и результаты исследований спермы у мужчин с коронавирусным заболеванием 2019» . Открытая сеть JAMA . 3 (5): e208292. doi : 10.1001/jamanetworkopen.2020.8292 . ПМК 7206502 . ПМИД 32379329 .
- ^ Вёлфель Р., Корман В.М., Гуггемос В., Зайльмайер М., Занге С., Мюллер М.А., Нимейер Д., Джонс Т.К., Фоллмар П., Роте С., Хельшер М., Блейкер Т., Брюнинк С., Шнайдер Дж., Эманн Р., Цвирглмайер К., Дростен С. , Вендтнер С. (май 2020 г.). «Вирусологическая оценка госпитализированных больных с COVID-2019» . Природа . 581 (7809): 465-469. Нагрудный код : 2020Nature.581..465W . дои : 10.1038/s41586-020-2196-x . ПМИД 32235945 .
- ^ Купфершмидт К. (февраль 2020 г.). «Исследование, утверждающее, что новый коронавирус может передаваться людьми без симптомов, ошибочно». Наука . дои : 10.1126/science.abb1524 . S2CID 214094598 .
- ^ К К.К., Цанг ОТ, Люн В.С., Там А.Р., Ву TC, Лунг Д.К., Ип CC, Цай Дж.П., Чан Дж.М., Чик Т.С., Лау Д.П., Чой С.И., Чен Л.Л., Чан В.М., Чан К.Х., Ип Дж.Д., Нг AC , Пун Р.В., Луо КТ, Ченг В.К., Чан Дж.Ф., Хунг И.Ф., Чен З., Чен Х., Юэнь К.Ю. (май 2020 г.). «Временные профили вирусной нагрузки в образцах слюны из задней части ротоглотки и реакции антител в сыворотке крови во время заражения SARS-CoV-2: наблюдательное когортное исследование» . «Ланцет». Инфекционные болезни . 20 (5): 565–574. дои : 10.1016/S1473-3099(20)30196-1 . ПМЦ 7158907 . ПМИД 32213337 .
- ^ Аванзато В.А., Мэтсон М.Дж., Зайферт С.Н., Прайс Р., Уильямсон Б.Н., Анзик С.Л., Барбиан К., Джадсон С.Д., Фишер Э.Р., Мартенс С., Боуден Т.А., де Вит Е, Риедо FX, Мюнстер В.Дж. (декабрь 2020 г.). «Тематическое исследование: длительное выделение вируса SARS-CoV-2 от бессимптомного человека с ослабленным иммунитетом, больного раком» . Клетка . 183 (7): 1901–1912.е9. дои : 10.1016/j.cell.2020.10.049 . ПМЦ 7640888 . PMID 33248470 .
- ^ Перейти обратно: а б Хоу Ю., Окуда К., Эдвардс С.Э., Мартинес Д.Р., Асакура Т., Диннон К.Х., Като Т., Ли Р.Э., Йонт Б.Л., Масценик Т.М., Чен Г., Оливье К.Н., Гио А., Це Л.В., Лейст С.Р., Гралински Л.Е., Шефер А. , Данг Х., Гилмор Р., Накано С., Сан Л., Фулчер М.Л., Ливраги-Бутрико А., Найсли Н.И., Кэмерон М., Кэмерон С., Кельвин Д.Д., де Силва А., Марголис Д.М., Маркманн А., Бартельт Л., Зумвалт Р., Мартинес. Ф.Дж., Сальваторе С.П., Борчук А., Тата П.Р., Сонтаке В., Кимпл А., Джасперс И., О'Нил В.К., Рэнделл С.Х., Баучер Р.К., Барик Р.С. (июль 2020 г.). «Обратная генетика SARS-CoV-2 выявляет переменный градиент инфекции в дыхательных путях» . Клетка . 182 (2): 429–446.e14. doi : 10.1016/j.cell.2020.05.042 . ПМЦ 7250779 . ПМИД 32526206 .
- ^ Банерджи А., Моссман К., Бейкер М.Л. (февраль 2021 г.). «Зооантропонозный потенциал SARS-CoV-2 и последствия реинтродукции в популяцию людей» . Клетка-хозяин и микроб . 29 (2): 160–164. дои : 10.1016/j.chom.2021.01.004 . ПМЦ 7837285 . ПМИД 33539765 .
- ^ «Вопросы и ответы по COVID-19: МЭБ – Всемирная организация по охране здоровья животных» . www.oie.int . Архивировано из оригинала 31 марта 2020 года . Проверено 16 апреля 2020 г.
- ^ Гольдштейн Дж. (6 апреля 2020 г.). «Тигр из зоопарка Бронкса заболел коронавирусом» . Нью-Йорк Таймс . Архивировано из оригинала 9 апреля 2020 года . Проверено 10 апреля 2020 г.
- ^ «Заявление Министерства сельского хозяйства США о подтверждении Covid-19 у тигра в Нью-Йорке» . Министерство сельского хозяйства США . 5 апреля 2020 года. Архивировано из оригинала 15 апреля 2020 года . Проверено 16 апреля 2020 г.
- ^ «Если у вас есть животные — коронавирусная болезнь 2019 (COVID-19)» . Центры по контролю и профилактике заболеваний (CDC) . 13 апреля 2020 года. Архивировано из оригинала 1 апреля 2020 года . Проверено 16 апреля 2020 г.
- ^ Всемирная организация здравоохранения (1 февраля 2020 г.). Новый коронавирус (2019-nCoV): отчет о ситуации, 12 (Отчет). Всемирная организация здравоохранения . hdl : 10665/330777 .
- ^ Нограды Б (ноябрь 2020 г.). «Что говорят данные о бессимптомных инфекциях COVID» . Природа . 587 (7835): 534–535. Бибкод : 2020Natur.587..534N . дои : 10.1038/d41586-020-03141-3 . ПМИД 33214725 .
- ^ Ли Р., Пей С., Чен Б., Сун Ю, Чжан Т., Ян В., Шаман Дж. (май 2020 г.). «Значительная недокументированная инфекция способствует быстрому распространению нового коронавируса (SARS-CoV-2)» . Наука . 368 (6490): 489–493. Бибкод : 2020Sci...368..489L . дои : 10.1126/science.abb3221 . ПМЦ 7164387 . ПМИД 32179701 .
- ^ Daily Telegraph , четверг, 28 мая 2020 г., стр. 2, столбец 1, в котором упоминается медицинский журнал Thorax ; Thorax Статья , май 2020 г., COVID-19: по следам Эрнеста Шеклтона. Архивировано 30 мая 2020 г. в Wayback Machine.
- ^ Хэ X, Лау Э.Х., Ву П, Дэн X, Ван J, Хао X, Лау YC, Вонг JY, Гуань Y, Тан X, Мо X, Чэнь Y, Ляо Б, Чен В, Ху Ф, Чжан Q, Чжун М , Ву Ю, Чжао Л., Чжан Ф., Коулинг Б.Дж., Ли Ф., Люн Г.М. (сентябрь 2020 г.). «Поправка автора: временная динамика выделения вируса и передачи COVID-19» . Природная медицина . 26 (9): 1491–1493. дои : 10.1038/s41591-020-1016-z . ПМК 7413015 . ПМИД 32770170 .
- ^ Перейти обратно: а б Ледфорд Х (сентябрь 2020 г.). «Реинфекции коронавирусом: три вопроса, которые задают ученые» . Природа . 585 (7824): 168–169. дои : 10.1038/d41586-020-02506-y . ПМИД 32887957 . S2CID 221501940 .
- ^ Перейти обратно: а б К К.К., Хунг И.Ф., Ип Дж.Д., Чу А.В., Чан В.М., Там А.Р., Фонг Ч., Юань С., Цой Х.В., Нг А.С., Ли Л.Л., Ван П., Цо Э., То В.К., Цанг Д., Чан К.Х., Хуан Дж.Д. , Кок К.Х., Ченг В.К., Юэнь К.Ю. (август 2020 г.). «Реинфекция COVID-19 филогенетически отличным штаммом SARS-коронавируса-2, подтвержденная полногеномным секвенированием» . Клинические инфекционные болезни . 73 (9): e2946–e2951. дои : 10.1093/cid/ciaa1275 . ПМЦ 7499500 . ПМИД 32840608 . S2CID 221308584 .
- ^ Перейти обратно: а б Тиллетт Р.Л., Севински-младший, Хартли П.Д., Кервин Х., Кроуфорд Н., Горзальски А., Лавердюр С., Верма СК, Россетто СС, Джексон Д., Фаррелл М.Дж., Ван Хузер С., Пандори М. (январь 2021 г.). «Геномные доказательства повторного заражения SARS-CoV-2: тематическое исследование» . «Ланцет». Инфекционные болезни . 21 (1): 52–58. дои : 10.1016/S1473-3099(20)30764-7 . ПМК 7550103 . ПМИД 33058797 .
- ^ Перейти обратно: а б Холмс Э.К., Гольдштейн С.А., Расмуссен А.Л., Робертсон Д.Л., Критс-Кристоф А., Вертхайм Дж.О., Энтони С.Дж., Барклай В.С., Бони М.Ф., Доэрти ПК, Фаррар Дж. (август 2021 г.). «Происхождение SARS-CoV-2: критический обзор» . Клетка . 184 (19): 4848–4856. дои : 10.1016/j.cell.2021.08.017 . ПМЦ 8373617 . ПМИД 34480864 .
- ^ Перейти обратно: а б с Чжоу П, Ян XL, Ван XG, Ху Б, Чжан Л, Чжан В, Си ХР, Чжу Ю, Ли Б, Хуан CL, Чен HD, Чэнь Дж, Ло Ю, Го Х, Цзян Р.Д., Лю MQ, Чен Ю , Шэнь XR, Ван X, Чжэн XS, Чжао К, Чен QJ, Дэн Ф, Лю ЛЛ, Ян Б, Чжан FX, Ван Ю, Сяо ГФ, Ши ZL (март 2020 г.). «Вспышка пневмонии, связанная с новым коронавирусом вероятного происхождения от летучих мышей» . Природа . 579 (7798): 270–273. Бибкод : 2020Natur.579..270Z . дои : 10.1038/s41586-020-2012-7 . ПМК 7095418 . ПМИД 32015507 .
- ^ Эшнер К. (28 января 2020 г.). «Мы до сих пор не уверены, откуда на самом деле взялся уханьский коронавирус» . Популярная наука . Архивировано из оригинала 30 января 2020 года . Проверено 30 января 2020 г.
- ^ Хуан С, Ван Ю, Ли Х, Жэнь Л, Чжао Дж, Ху Ю, Чжан Л, Фань Г, Сюй Дж, Гу Икс, Чэн Цзы, Ю Т, Ся Дж, Вэй Ю, Ву В, Се Икс, Инь В , Ли Х, Лю М, Сяо Ю, Гао Х, Го Л, Се Дж, Ван Г, Цзян Р, Гао З, Цзинь Ц, Ван Дж, Цао Б (февраль 2020). «Клинические особенности пациентов, инфицированных новым коронавирусом 2019 года, в Ухане, Китай» . Ланцет . 395 (10223): 497–506. дои : 10.1016/S0140-6736(20)30183-5 . ПМЦ 7159299 . ПМИД 31986264 .
- ^ Перейти обратно: а б Чэнь Н, Чжоу М, Донг Икс, Цюй Дж, Гун Ф, Хан Ю, Цю Ю, Ван Дж, Лю Ю, Вэй Ю, Ся Дж, Ю Т, Чжан Икс, Чжан Л (февраль 2020 г.). «Эпидемиологические и клинические характеристики 99 случаев новой коронавирусной пневмонии 2019 года в Ухане, Китай: описательное исследование» . Ланцет . 395 (10223): 507–513. дои : 10.1016/S0140-6736(20)30211-7 . ПМК 7135076 . ПМИД 32007143 .
- ^ Перейти обратно: а б Сираноски Д. (март 2020 г.). «Тайна животного источника коронавируса углубляется» . Природа . 579 (7797): 18–19. Бибкод : 2020Natur.579...18C . дои : 10.1038/d41586-020-00548-w . ПМИД 32127703 .
- ^ Ю ВБ, Тан ГД, Чжан Л, Корлетт РТ (май 2020 г.). «Расшифровка эволюции и передачи нового коронавируса пневмонии (SARS-CoV-2 / HCoV-19) с использованием полногеномных данных» . Зоологические исследования . 41 (3): 247–257. дои : 10.24272/j.issn.2095-8137.2020.022 . ПМЦ 7231477 . ПМИД 32351056 .
- ^ Перейти обратно: а б с д и Совместная исследовательская группа ВОЗ и Китая (30 марта 2021 г.). Глобальное исследование происхождения SARS-CoV-2, организованное ВОЗ: Китайская часть . Женева, Швейцария: Всемирная организация здравоохранения . Проверено 31 мая 2023 г.
- ^ Воробей М. (декабрь 2021 г.). «Анализ ранних случаев COVID-19 в Ухане». Наука . 374 (6572): 1202–1204. Бибкод : 2021Sci...374.1202W . дои : 10.1126/science.abm4454 . ПМИД 34793199 . S2CID 244403410 .
- ^ Кан Л., Хе Дж., Шарп А.К., Ван Х, Браун А.М., Михалак П., Вегер-Лукарелли Дж. (август 2021 г.). «Избирательное изменение гена Spike привело к адаптации человека к SARS-CoV-2» . Клетка . 184 (17): 4392–4400.e4. дои : 10.1016/j.cell.2021.07.007 . ПМК 8260498 . ПМИД 34289344 .
- ^ Декаро Н., Лоруссо А (май 2020 г.). «Новый коронавирус человека (SARS-CoV-2): урок коронавирусов животных» . Ветеринарная микробиология . 244 : 108693. doi : 10.1016/j.vetmic.2020.108693 . ПМЦ 7195271 . ПМИД 32402329 .
- ^ Робсон Ф., Хан К.С., Ле ТК, Пэрис С., Демирбаг С., Барфусс П., Рокки П., Нг В.Л. (август 2020 г.). «Корректировка РНК коронавируса: молекулярная основа и терапевтическое нацеливание [опубликованная поправка появляется в Mol Cell. 17 декабря 2020 г.;80(6):1136–1138]» . Молекулярная клетка . 79 (5): 710–727. doi : 10.1016/j.molcel.2020.07.027 . ПМЦ 7402271 . ПМИД 32853546 .
- ^ Тао К., Цзоу П.Л., Нухин Дж., Гупта Р.К., де Оливейра Т., Косаковский пруд С.Л., Фера Д., Шафер Р.В. (декабрь 2021 г.). «Биологическое и клиническое значение новых вариантов SARS-CoV-2» . Обзоры природы Генетика . 22 (12): 757–773. дои : 10.1038/s41576-021-00408-x . ПМЦ 8447121 . ПМИД 34535792 .
- ^ Теммам С., Вонгфайлот К., Салазар Э.Б., Мунье С., Бономи М., Реньо Б., Дуангбубфа Б., Карами Ю., Кретьен Д., Санамсай Д., Каяфет В. (февраль 2022 г.). «Коронавирусы летучих мышей, родственные SARS-CoV-2 и инфекционные для клеток человека» . Природа . 604 (7905): 330–336. Бибкод : 2022Natur.604..330T . дои : 10.1038/s41586-022-04532-4 . ПМИД 35172323 . S2CID 246902858 .
- ^ Маллапати С (24 сентября 2021 г.). «Ближайшие известные родственники вируса, вызывающего COVID-19, обнаружены в Лаосе» . Природа . 597 (7878): 603. Бибкод : 2021Natur.597..603M . дои : 10.1038/d41586-021-02596-2 . ПМИД 34561634 . S2CID 237626322 .
- ^ «Недавно обнаруженные вирусы летучих мышей намекают на происхождение Covid» . Нью-Йорк Таймс . 14 октября 2021 г.
- ^ «Изолят RaTG13 коронавируса летучих мышей, полный геном» . Национальный центр биотехнологической информации (NCBI) . 10 февраля 2020 года. Архивировано из оригинала 15 мая 2020 года . Проверено 5 марта 2020 г.
- ^ «Аргумент о бритве Оккама» не изменился в пользу утечки из лаборатории» . Snopes.com . Сноупс. 16 июля 2021 г. Проверено 18 июля 2021 г.
- ^ Лу Р, Чжао X, Ли Дж, Ню П, Ян Б, У Х, Ван В, Сун Х, Хуан Б, Чжу Н, Би Ю, Ма Х, Чжан Ф, Ван Л, Ху Т, Чжоу Х, Ху Z , Чжоу В., Чжао Л., Чэнь Дж., Мэн Ю., Ван Дж., Линь Ю., Юань Дж., Се З., Ма Дж., Лю У.Дж., Ван Д., Сюй В., Холмс Э.К., Гао Г.Ф., У Г, Чен В, Ши В, Тан В (февраль 2020 г.). «Геномная характеристика и эпидемиология нового коронавируса 2019 года: значение для происхождения вируса и связывания с рецепторами» . Ланцет . 395 (10224): 565–574. дои : 10.1016/S0140-6736(20)30251-8 . ПМК 7159086 . ПМИД 32007145 .
- ^ О'Киф Дж., Фриман С., Никол А. (21 марта 2021 г.). Основные сведения о передаче SARS-CoV-2 . Ванкувер, Британская Колумбия: Национальный сотрудничающий центр по гигиене окружающей среды (NCCEH). ISBN 978-1-988234-54-0 . Архивировано из оригинала 12 мая 2021 года . Проверено 12 мая 2021 г.
- ^ Сяо К, Чжай Дж, Фэн Ю, Чжоу Н, Чжан X, Цзоу JJ, Ли Н, Го Ю, Ли Х, Шэнь Икс, Чжан Цз, Шу Ф, Хуан В, Ли Ю, Чжан Цз, Чэнь РА, Ву Юй ., Пэн С.М., Хуан М., Се В.Дж., Цай К.Х., Хоу Ф.Х., Чэнь В., Сяо Л., Шэнь Ю. (июль 2020 г.). «Выделение коронавируса, связанного с SARS-CoV-2, от малайских панголинов» . Природа . 583 (7815): 286–289. Бибкод : 2020Natur.583..286X . дои : 10.1038/s41586-020-2313-x . ПМИД 32380510 . S2CID 218557880 .
- ^ Чжао Дж., Цуй В., Тянь Б.П. (2020). «Потенциальные промежуточные хозяева SARS-CoV-2» . Границы микробиологии . 11 : 580137. doi : 10.3389/fmicb.2020.580137 . ПМЦ 7554366 . ПМИД 33101254 .
- ^ «Почему так сложно отследить происхождение COVID-19» . Наука . Нэшнл Географик. 10 сентября 2021 г.
- ^ Перейти обратно: а б с Ху Б, Го Х, Чжоу П, Ши ЗЛ (март 2021 г.). «Характеристика SARS-CoV-2 и COVID-19» . Обзоры природы. Микробиология . 19 (3): 141–154. дои : 10.1038/s41579-020-00459-7 . ПМЦ 7537588 . ПМИД 33024307 .
- ^ Джованетти М., Бенедетти Ф., Кампизи Г., Чиккоцци А., Фабрис С., Чеккарелли Г., Тамбоне В., Карузо А., Анджелетти С., Зелла Д., Чиккоцци М. (январь 2021 г.). «Модели эволюции SARS-CoV-2: краткий обзор вариантов его генома» . Связь с биохимическими и биофизическими исследованиями . 538 :88–91. дои : 10.1016/j.bbrc.2020.10.102 . ПМЦ 7836704 . ПМИД 33199021 . S2CID 226988090 .
- ^ Перейти обратно: а б с Вьковски П., Крацель А., Штайнер С., Сталдер Х., Тиль В. (март 2021 г.). «Биология и репликация коронавируса: последствия для SARS-CoV-2» . Обзоры природы. Микробиология . 19 (3): 155–170. дои : 10.1038/s41579-020-00468-6 . ПМЦ 7592455 . ПМИД 33116300 .
- ^ Перейти обратно: а б Чжу Н, Чжан Д, Ван В, Ли Х, Ян Б, Сун Дж, Чжао Икс, Хуан Б, Ши В, Лу Р, Ню П, Чжан Ф, Ма Икс, Ван Д, Сюй В, Ву Г, Гао ГФ , Тан В. (февраль 2020 г.). «Новый коронавирус от пациентов с пневмонией в Китае, 2019 г.» . Медицинский журнал Новой Англии . 382 (8): 727–733. дои : 10.1056/NEJMoa2001017 . ПМК 7092803 . ПМИД 31978945 .
- ^ «Филогения SARS-подобных бетакоронавирусов» . следующий штамм . Архивировано из оригинала 20 января 2020 года . Проверено 18 января 2020 г.
- ^ Вонг AC, Ли X, Лау СК, Ву ПК (февраль 2019 г.). «Глобальная эпидемиология коронавирусов летучих мышей» . Вирусы . 11 (2): 174. дои : 10.3390/v11020174 . ПМК 6409556 . ПМИД 30791586 .
- ^ Перейти обратно: а б Сингх Д., Йи С.В. (апрель 2021 г.). «О происхождении и эволюции SARS-CoV-2» . Экспериментальная и молекулярная медицина . 53 (4): 537–547. дои : 10.1038/s12276-021-00604-z . ПМК 8050477 . ПМИД 33864026 .
- ^ [ PubMed ] Джексон Б., Бони М.Ф., Булл М.Дж., Коллеран А., Колкухун Р.М., Дарби А.С., Холденби С., Хилл В., Лукачи А., Маккроун Дж.Т., Николлс С.М., О'Тул А., Паккиарини Н., Поплавски Р., Шер Э., Тодд Ф., Вебстер Х.Дж., Уайтхед М., Вежбицки С., Ломан Н.Дж., Коннор Т.Р., Робертсон Д.Л., Пайбус О.Г., Рамбо А. (сентябрь 2021 г.). «Поколение и передача межлинейных рекомбинантов при пандемии SARS-CoV-2» . Ячейка 184 (20):5179–5188.e8. doi : 10.1016/j.cell.2021.08.014 . ПМЦ 8367733 . ПМИД 34499854 . S2CID 237099659 .
- ^ Перейти обратно: а б «КоВ2020» . GISAID ЭпифлюДБ . Архивировано из оригинала 12 января 2020 года . Проверено 12 января 2020 г.
- ^ Ким Д., Ли Дж.И., Ян Дж.С., Ким Дж.В., Ким В.Н., Чанг Х. (май 2020 г.). «Архитектура транскриптома SARS-CoV-2» . Клетка . 181 (4): 914–921.e10. дои : 10.1016/j.cell.2020.04.011 . ПМЦ 7179501 . ПМИД 32330414 .
- ^ Хоссейн М.Г., Тан Юд, Актер С., Чжэн С. (май 2022 г.). «Роль сайта расщепления многоосновного фурина шиповидного белка в репликации, патогенезе SARS-CoV-2, иммунных реакциях хозяина и вакцинации». Журнал медицинской вирусологии . 94 (5): 1815–1820. дои : 10.1002/jmv.27539 . ПМИД 34936124 . S2CID 245430230 .
- ^ К.К., Шридхару С., Чиу К.Х., Хунгу Д.Л., Ли X, Хунгу И.Ф., Таму А.Р., Чунг Т.В., Чану Дж.Ф., Чжану А.Дж., Ченгу В.К., Юэню Кюй (декабрь 2021 г.). «Уроки, извлеченные через год после появления SARS-CoV-2, приведшего к пандемии COVID-19» . Новые микробы и инфекции . 10 (1): 507–535. дои : 10.1080/22221751.2021.1898291 . ПМК 8006950 . ПМИД 33666147 .
- ^ Перейти обратно: а б Джексон CB, Фарзан М, Чен Б, Чой Х (январь 2022 г.). «Механизмы проникновения SARS-CoV-2 в клетки» . Nature Reviews Молекулярно-клеточная биология . 23 (1): 3–20. дои : 10.1038/s41580-021-00418-x . ПМЦ 8491763 . ПМИД 34611326 .
- ^ Браун Э., Заутер Д. (2019). «Фурин-опосредованный процессинг белков при инфекционных заболеваниях и раке» . Клиническая и трансляционная иммунология . 8 (8): е1073. дои : 10.1002/cti2.1073 . ПМК 6682551 . ПМИД 31406574 .
- ^ Ванкадари Н. (август 2020 г.). «Структура связывания фуриновой протеазы с шиповым гликопротеином SARS-CoV-2 и значение для потенциальных мишеней и вирулентности» . Журнал физической химии . 11 (16): 6655–6663. doi : 10.1021/acs.jpclett.0c01698 . ПМК 7409919 . ПМИД 32787225 .
- ^ Перейти обратно: а б Кутар Б, Валле С, де Ламбаллери Х, Канард Б, Сейда Н.Г., Декроли Э (апрель 2020 г.). «Шипучий гликопротеин нового коронавируса 2019-nCoV содержит фуриноподобный сайт расщепления, отсутствующий в CoV той же клады» . Противовирусные исследования . 176 (7): 104742. Бибкод : 2020CBio...30E1346Z . дои : 10.1016/j.cub.2020.03.022 . ПМК 7114094 . ПМИД 32057769 .
- ^ Чжан Т, У Ц, Чжан Цз (апрель 2020 г.). «Вероятное происхождение SARS-CoV-2 от панголинов, связанное со вспышкой COVID-19» . Современная биология . 30 (7): 1346–1351.e2. Бибкод : 2020CBio...30E1346Z . дои : 10.1016/j.cub.2020.03.022 . ПМК 7156161 . ПМИД 32197085 .
- ^ У Ю, Чжао С (декабрь 2020 г.). «Участки расщепления фурина естественным образом встречаются у коронавирусов» . Исследования стволовых клеток . 50 : 102115. doi : 10.1016/j.scr.2020.102115 . ПМЦ 7836551 . ПМИД 33340798 .
- ^ Воробей М., Пекар Дж., Ларсен Б.Б., Нельсон М.И., Хилл В., Джой Дж.Б., Рамбо А., Сушард М.А., Вертхайм Дж.О., Леми П. (октябрь 2020 г.). «Появление SARS-CoV-2 в Европе и Северной Америке» . Наука . 370 (6516): 564–570. дои : 10.1126/science.abc8169 . ПМК 7810038 . ПМИД 32912998 .
- ^ «Первоначальный выпуск генома нового коронавируса» . Вирусологический . 11 января 2020 года. Архивировано из оригинала 12 января 2020 года . Проверено 12 января 2020 г.
- ^ Перейти обратно: а б Бедфорд Т., Неер Р., Хэдфилд Н., Ходкрофт Э., Ильцисин М., Мюллер Н. «Геномный анализ распространения nCoV: отчет о ситуации от 30 января 2020 г.» . nextstrain.org . Архивировано из оригинала 15 марта 2020 года . Проверено 18 марта 2020 г.
- ^ Сунь Дж., Хэ В.Т., Ван Л., Лай А., Цзи Икс, Чжай Икс, Ли Г, Сушард М.А., Тянь Дж., Чжоу Дж., Вейт М., Су С. (май 2020 г.). «COVID-19: эпидемиология, эволюция и междисциплинарные перспективы» . Тенденции молекулярной медицины . 26 (5): 483–495. doi : 10.1016/j.molmed.2020.02.008 . ПМЦ 7118693 . ПМИД 32359479 .
- ^ «Геномная эпидемиология нового коронавируса – глобальная выборка» . Следующий штамм . 25 октября 2021 года. Архивировано из оригинала 20 апреля 2020 года . Проверено 26 октября 2021 г.
- ^ Группа по изучению Coronaviridae Международного комитета по таксономии вирусов (апрель 2020 г.). «Вид коронавируса, связанного с тяжелым острым респираторным синдромом: классификация 2019-nCoV и присвоение ему названия SARS-CoV-2» . Природная микробиология . 5 (4): 536–544. дои : 10.1038/s41564-020-0695-z . ПМК 7095448 . ПМИД 32123347 .
- ^ «Новый, более заразный штамм COVID-19 теперь доминирует в глобальных случаях заражения вирусом: исследование» . www.medicalxpress.com . Архивировано из оригинала 17 ноября 2020 года . Проверено 16 августа 2020 г. .
- ^ Корбер Б., Фишер В.М., Гнанакаран С., Юн Х., Тейлер Дж., Абфальтерер В., Хенгартнер Н., Георгий Э.Э., Бхаттачарья Т., Фоли Б., Хасти К.М., Паркер М.Д., Партридж Д.Г., Эванс К.М., Фриман Т.М., де Сильва Т.И., МакДэнал C, Перес Л.Г., Тан Х., Moon-Walker A, Уилан С.П., ЛаБранш CC, Сапфир Э.О., Монтефиори, округ Колумбия (август 2020 г.). «Отслеживание изменений в пике SARS-CoV-2: доказательства того, что D614G увеличивает инфекционность вируса COVID-19» . Клетка . 182 (4): 812–827.e19. doi : 10.1016/j.cell.2020.06.043 . ПМЦ 7332439 . ПМИД 32697968 .
- ^ Дхама К., Хан С., Тивари Р., Сиркар С., Бхат С., Малик Ю.С., Сингх К.П., Чайкумпа В., Бонилья-Алдана Д.К., Родригес-Моралес А.Дж. (сентябрь 2020 г.). «Коронавирусная болезнь 2019-COVID-19» . Обзоры клинической микробиологии . 33 (4). дои : 10.1128/CMR.00028-20 . ПМЦ 7405836 . ПМИД 32580969 .
- ^ Докрилл П (11 ноября 2020 г.). «Ученые только что обнаружили загадочно скрытый «ген внутри гена» SARS-CoV-2» . НаукаАлерт . Архивировано из оригинала 17 ноября 2020 года . Проверено 11 ноября 2020 г.
- ^ Нельсон К.В., Ардерн З., Голдберг Т.Л., Мэн С., Куо Ч., Людвиг С., Колокотронис С.О., Вэй Х (октябрь 2020 г.). «Динамически развивающийся новый перекрывающийся ген как фактор пандемии SARS-CoV-2» . электронная жизнь . 9 . doi : 10.7554/eLife.59633 . ПМЦ 7655111 . ПМИД 33001029 .
- ^ Перейти обратно: а б Чжоу Х., Цзи Дж., Чен Х., Би Ю., Ли Дж., Ван Ц. и др. (август 2021 г.). «Идентификация новых коронавирусов летучих мышей проливает свет на эволюционное происхождение SARS-CoV-2 и родственных вирусов» . Клетка . 184 (17): 4380–4391.e14. doi : 10.1016/j.cell.2021.06.008 . ПМЦ 8188299 . ПМИД 34147139 .
- ^ Перейти обратно: а б Вачараплуесади С., Тан К.В., Маниорн П., Дуэнкэ П., Чжу Ф., Джойджинда Ю. и др. (февраль 2021 г.). «Доказательства наличия коронавирусов, связанных с SARS-CoV-2, циркулирующих у летучих мышей и панголинов в Юго-Восточной Азии » Природные коммуникации . 12 (1): 972. Бибкод : 2021NatCo..12..972W . дои : 10.1038/ s41467-021-21240-1 ПМЦ 7873279 . ПМИД 33563978 .
- ^ Мураками С., Китамура Т., Сузуки Дж., Сато Р., Аой Т., Фуджи М. и др. (декабрь 2020 г.). «Обнаружение и характеристика сарбековируса летучих мышей, филогенетически родственного SARS-CoV-2, Япония» . Новые инфекционные заболевания . 26 (12): 3025–3029. дои : 10.3201/eid2612.203386 . ПМК 7706965 . ПМИД 33219796 .
- ^ Перейти обратно: а б Чжоу Х, Чен X, Ху Т, Ли Дж, Сун Х, Лю Ю и др. (июнь 2020 г.). «Новый коронавирус летучих мышей, тесно связанный с SARS-CoV-2, содержит естественные вставки в сайте расщепления S1/S2 белка-шипа» . Современная биология . 30 (11): 2196–2203.e3. дои : 10.1016/j.cub.2020.05.023 . ПМЦ 7211627 . ПМИД 32416074 .
- ^ Лам Т.Т., Цзя Н., Чжан Ю.В., Шум М.Х., Цзян Дж.Ф., Чжу Х.К. и др. (июль 2020 г.). «Идентификация коронавирусов, связанных с SARS-CoV-2, у малайских панголинов». Природа . 583 (7815): 282–285. Бибкод : 2020Natur.583..282L . дои : 10.1038/s41586-020-2169-0 . ПМИД 32218527 . S2CID 214683303 .
- ^ Сяо К., Чжай Дж., Фэн Ю., Чжоу Н., Чжан Х., Цзоу Дж.Дж. и др. (июль 2020 г.). «Выделение коронавируса, связанного с SARS-CoV-2, от малайских панголинов» Природа 583 (7815): 286–289. Бибкод : 2020Nature.583..286X . дои : 10.1038/s41586-020-2313-x . ПМИД 32380510 . S2CID 256822274 .
- ^ Перейти обратно: а б Делон Д., Хул В., Карлссон Е.А., Хассанин А., Оу Т.П., Байдалюк А. и др. (ноябрь 2021 г.). «Новый коронавирус, связанный с SARS-CoV-2, у летучих мышей из Камбоджи» . Природные коммуникации . 12 (1): 6563. Бибкод : 2021NatCo..12.6563D . дои : 10.1038/s41467-021-26809-4 . ПМЦ 8578604 . ПМИД 34753934 .
- ^ Чжоу Х, Чен X, Ху Т, Ли Дж, Сун Х, Лю Ю и др. (июнь 2020 г.). «Новый коронавирус летучих мышей, тесно связанный с SARS-CoV-2, содержит естественные вставки в сайте расщепления S1/S2 белка-шипа» . Современная биология . 30 (11): 2196–2203.e3. дои : 10.1016/j.cub.2020.05.023 . ПМЦ 7211627 . ПМИД 32416074 .
- ^ Чжоу П., Ян XL, Ван XG, Ху Б, Чжан Л., Чжан В. и др. (март 2020 г.). «Вспышка пневмонии, связанная с новым коронавирусом вероятного происхождения от летучих мышей» . Природа . 579 (7798): 270–273. Бибкод : 2020Natur.579..270Z . дои : 10.1038/s41586-020-2012-7 . ПМК 7095418 . ПМИД 32015507 .
- ^ Теммам С., Вонгфайлот К., Бакеро Е., Мунье С., Бономи М., Рено Б. и др. (апрель 2022 г.). «Коронавирусы летучих мышей, родственные SARS-CoV-2 и заразные для клеток человека». Природа . 604 (7905): 330–336. Бибкод : 2022Natur.604..330T . дои : 10.1038/s41586-022-04532-4 . ПМИД 35172323 . S2CID 246902858 .
- ^ Кояма Т., Платт Д., Парида Л. (июль 2020 г.). «Вариантный анализ геномов SARS-CoV-2» . Бюллетень Всемирной организации здравоохранения . 98 (7): 495–504. дои : 10.2471/BLT.20.253591 . ПМЦ 7375210 . ПМИД 32742035 .
Всего мы обнаружили 65776 вариантов, из них 5775 различных.
- ^ Алм Э., Броберг Э.К., Коннор Т., Ходкрофт Э.Б., Комиссаров А.Б., Маурер-Стро С., Мелиду А., Неер Р.А., О'Тул А., Переяслов Д. (август 2020 г.). с января по июнь». «Географическое и временное распределение клад SARS-CoV-2 в Европейском регионе ВОЗ , Евронаблюдение . 25 (32). дои : 10.2807/1560-7917.ES.2020.25.32.2001410 . ПМЦ 7427299 . ПМИД 32794443 .
- ^ Всемирная организация здравоохранения (27 ноября 2021 г.). «Отслеживание вариантов SARS-CoV-2» . Всемирная организация здравоохранения . Архивировано из оригинала 6 июня 2021 года . Проверено 28 ноября 2021 г.
- ^ «Вариантный штамм SARS-CoV-2, связанный с норками – Дания» . ВОЗ . 3 декабря 2020 года. Архивировано из оригинала 31 декабря 2020 года . Проверено 30 декабря 2020 г.
- ^ Отправитель Р., Бар-Он Ю.М., Глейзер С., Бернштейн Б., Фламхольц А., Филлипс Р., Майло Р. (апрель 2021 г.). «Общее количество и масса вирионов SARS-CoV-2» . MedRxiv: Сервер препринтов для медицинских наук . дои : 10.1101/2020.11.16.20232009 . ПМЦ 7685332 . ПМИД 33236021 .
- ^ Перейти обратно: а б с Ву С, Лю Ю, Ян Ю, Чжан П, Чжун В, Ван Ю, Ван Ц, Сюй Ю, Ли М, Ли Икс, Чжэн М, Чен Л, Ли Х (май 2020 г.). «Анализ терапевтических мишеней для SARS-CoV-2 и открытие потенциальных лекарств вычислительными методами» . Акта Фармасьютика Синика Б. 10 (5): 766–788. дои : 10.1016/j.apsb.2020.02.008 . ПМК 7102550 . ПМИД 32292689 .
- ^ Перейти обратно: а б Wrapp D, Wang N, Corbett KS, Goldsmith JA, Hsieh CL, Abiona O, Graham BS, McLellan JS (март 2020 г.). «Крио-ЭМ структура пика 2019-nCoV в префузионной конформации» . Наука . 367 (6483): 1260–1263. Бибкод : 2020Sci...367.1260W . дои : 10.1126/science.abb2507 . ПМЦ 7164637 . ПМИД 32075877 .
- ^ Мандельбаум РФ (19 февраля 2020 г.). «Ученые создают на атомном уровне картину потенциальной ахиллесовой пяты нового коронавируса» . Гизмодо . Архивировано из оригинала 8 марта 2020 года . Проверено 13 марта 2020 г.
- ^ Сохансандж Б.А., Розен Г.Л. (26 апреля 2022 г.). Галья М.М. (ред.). «Сопоставление данных для глубокого понимания: максимально эффективно использовать поток последовательностей генома SARS-CoV-2» . mSystems . 7 (2): e00035–22. дои : 10.1128/msystems.00035-22 . ISSN 2379-5077 . ПМЦ 9040592 . ПМИД 35311562 .
- ^ «ГИСАИД-gisaid.org» . сайт gisaid.org . Проверено 16 сентября 2023 г.
- ^ Кандил М., Ибрагим А., Файез М., Аль-Назави М. (июнь 2020 г.). «От SARS и MERS CoV к SARS-CoV-2: движение к более предвзятому использованию кодонов в вирусных структурных и неструктурных генах» . Журнал медицинской вирусологии . 92 (6): 660–666. дои : 10.1002/jmv.25754 . ПМЦ 7228358 . ПМИД 32159237 .
- ^ Перейти обратно: а б Хоу В (сентябрь 2020 г.). «Характеристика характера использования кодонов при SARS-CoV-2» . Вирусологический журнал . 17 (1): 138. дои : 10.1186/s12985-020-01395-x . ПМЦ 7487440 . ПМИД 32928234 .
- ^ Перейти обратно: а б Ван Ю, Мао Дж. М., Ван Г. Д., Луо З. П., Ян Л., Яо Ц, Чен КП (июль 2020 г.). «Человеческий SARS-CoV-2 эволюционировал, чтобы уменьшить количество динуклеотидов CG в открытых рамках считывания» . Научные отчеты . 10 (1): 12331. Бибкод : 2020NatSR..1012331W . дои : 10.1038/s41598-020-69342-y . ПМЦ 7378049 . ПМИД 32704018 .
- ^ Райс А.М., Кастильо Моралес А., Хо А.Т., Мордштейн С., Мюльхаузен С., Уотсон С., Кано Л., Янг Б., Кудла Г., Херст Л.Д. (январь 2021 г.). «Доказательства сильной мутационной предвзятости в сторону содержания U в SARS-CoV-2 и отбора против него: последствия для разработки вакцины» . Молекулярная биология и эволюция . 38 (1): 67–83. дои : 10.1093/molbev/msaa188 . ПМЦ 7454790 . ПМИД 32687176 .
- ^ Гу Х, Чу Д.К., Пейрис М., Пун Л.Л. (январь 2020 г.). «Многомерный анализ использования кодонов SARS-CoV-2 и других бетакоронавирусов» . Эволюция вирусов . 6 (1): veaa032. дои : 10.1093/ve/veaa032 . ПМЦ 7223271 . ПМИД 32431949 .
- ^ Ван Ц, Чжан Ю, Ву Л, Ню С, Сун С, Чжан Цз, Лу Г, Цяо С, Ху Ю, Юэнь Кюй, Ван Ц, Чжоу Х, Ян Дж, Ци Дж (май 2020 г.). «Структурная и функциональная основа проникновения SARS-CoV-2 с использованием человеческого ACE2» . Ячейка 181 (4): 894–904.e9. дои : 10.1016/j.cell.2020.03.045 . ПМЦ 7144619 . PMID 32275855 .
- ^ Сюй X, Чен П, Ван Дж, Фэн Дж, Чжоу Х, Ли X, Чжун В, Хао П (март 2020 г.). «Эволюция нового коронавируса в результате продолжающейся вспышки в Ухане и моделирование его шипового белка для риска передачи человеку» . Наука Китай Науки о жизни . 63 (3): 457–460. дои : 10.1007/s11427-020-1637-5 . ПМК 7089049 . ПМИД 32009228 .
- ^ Летко М., Марци А., Мюнстер В. (апрель 2020 г.). «Функциональная оценка проникновения в клетки и использования рецепторов для SARS-CoV-2 и других бетакоронавирусов линии B» . Природная микробиология . 5 (4): 562–569. дои : 10.1038/s41564-020-0688-y . ПМК 7095430 . ПМИД 32094589 .
- ^ Летко М., Марци А., Мюнстер В. (апрель 2020 г.). «Функциональная оценка проникновения в клетки и использования рецепторов для SARS-CoV-2 и других бетакоронавирусов линии B» . Природная микробиология . 5 (4): 562–569. дои : 10.1038/s41564-020-0688-y . ПМК 7095430 . ПМИД 32094589 .
- ^ Эль Сахли ХМ. «Геномная характеристика нового коронавируса 2019 года» . Медицинский журнал Новой Англии . Архивировано из оригинала 17 февраля 2020 года . Проверено 9 февраля 2020 г.
- ^ «Новая структура коронавируса указывает на цели для вакцин и методов лечения» . Национальные институты здравоохранения (NIH) . 2 марта 2020 года. Архивировано из оригинала 1 апреля 2020 года . Проверено 3 апреля 2020 г.
- ^ Ван К., Чен В., Чжан З., Дэн Ю., Лянь JQ, Ду П., Вэй Д., Чжан Ю., Сунь XX, Гун Л., Ян Икс, Хэ Л., Чжан Л., Ян З., Гэн Дж.Дж., Чэнь Р., Чжан Х. , Ван Б, Чжу Ю.М., Нан Г., Цзян Дж.Л., Ли Л., Ву Дж., Линь П., Хуан В., Се Л., Чжэн Чж., Чжан К., Мяо Дж.Л., Цуй HY, Хуан М, Чжан Дж, Фу Л, Ян XM, Чжао Z, Сунь С, Гу Х, Ван Z, Ван CF, Лу Ю, Лю YY, Ван QY, Бянь Х, Чжу П, Чэнь ZN (декабрь 2020 г.) . «Белок-шип CD147 — это новый путь заражения SARS-CoV-2 клеток-хозяев» . Сигнальная трансдукция и таргетная терапия . 5 (1): 283. bioRxiv 10.1101/2020.14.03.988345 . дои : 10.1038/s41392-020-00426-x . ПМЦ 7714896 . ПМИД 33277466 . S2CID 214725955 .
- ^ Саморано Куэрво Н., Гранво Н. (ноябрь 2020 г.). «ACE2: Доказательства роли входного рецептора для SARS-CoV-2 и последствия для сопутствующих заболеваний» . электронная жизнь . 9 . doi : 10.7554/eLife.61390 . ПМЦ 7652413 . ПМИД 33164751 .
- ^ «Анатомия убийцы: понимание SARS-CoV-2 и лекарств, которые могут уменьшить его силу» . Экономист . 12 марта 2020 г. Архивировано из оригинала 14 марта 2020 г. . Проверено 14 марта 2020 г.
- ^ Бичинг, Нью-Джерси, Флетчер Т.Е., Фаулер Р. (22 мая 2020 г.). «Передовой опыт BMJ: Коронавирусное заболевание 2019 (COVID-19)» (PDF) . БМЖ . Архивировано (PDF) из оригинала 13 июня 2020 года . Проверено 25 мая 2020 г.
- ^ Дрейман Н., ДеМарко Дж.К., Джонс К.А., Азизи С.А., Фроггатт Х.М., Тан К., Мальцева Н.И., Чен С., Николаеску В., Дворкин С., Ферлонг К., Катаят Р.С., Фирпо М.Р., Мастродоменико В., Брюс Э.А., Шмидт М.М., Еджейчак Р. , Муньос-Алия Ма , Шустер Б , Наир В , Хан К.Ю , О'Брайен А , Томациду А , Мейер Б , Виньюцци М , Миссиакас Д , Боттен Дж.В. , Брук С.Б. , Ли Х , Бейкер СК , Маунс BC , Хитон Н.С. Северсон В.Е., Палмер К.Е., Дикинсон BC, Йоахимиак А., Рэндалл Дж., Тэй С. (август 2021 г.). «Маситиниб — это широкий ингибитор 3CL коронавируса, который блокирует репликацию SARS-CoV- 2. Наука 373 (6557): 931–936. Бибкод : 2021Научный... 373..931D дои : 10.1126/science.abg5827 . ПМЦ 8809056 . ПМИД 34285133 .
- ^ Информационный бюллетень для медицинских работников: Разрешение на экстренное использование Паксловида (PDF) . Пфайзер . 22 декабря 2021 года. Архивировано из оригинала 23 декабря 2021 года.
- ^ «Пахловид ЭПАР» . Европейское агентство лекарственных средств (EMA) . 24 января 2022 г. Проверено 3 февраля 2022 г. Текст скопирован из источника, права на который принадлежат Европейскому агентству по лекарственным средствам. Воспроизведение разрешено при условии указания источника.
- ^ Пероральный противовирусный препарат от COVID-19, Паксловид, одобрен регулирующим органом Великобритании . Агентство по регулированию лекарственных средств и товаров медицинского назначения. 31 декабря 2021 г.
- ^ Министерство здравоохранения Канады разрешило применение Паксловида для пациентов с легкой и умеренной формой COVID-19 с высоким риском развития серьезного заболевания . Здоровье Канады . 17 января 2022 г. Проверено 24 апреля 2022 г.
- ^ FDA разрешает первый пероральный противовирусный препарат для лечения COVID-19 . США Управление по санитарному надзору за качеством пищевых продуктов и медикаментов (FDA) . 22 декабря 2021 г. Проверено 22 декабря 2021 г.
 В данную статью включен текст из этого источника, находящегося в свободном доступе .
В данную статью включен текст из этого источника, находящегося в свободном доступе .
- ^ Уиппл Т. (23 октября 2021 г.). «Moonshot — это ключ к работе по борьбе с Covid-19, в которой нуждается страна» . Таймс . Проверено 5 ноября 2021 г.
- ^ Роклов Дж., Сьёдин Х., Уайлдер-Смит А. (май 2020 г.). «Вспышка COVID-19 на круизном лайнере Diamond Princess: оценка эпидемического потенциала и эффективности мер общественного здравоохранения» . Журнал туристической медицины . 27 (3). дои : 10.1093/jtm/taaa030 . ПМЦ 7107563 . ПМИД 32109273 .
- ^ Ке Р., Ромеро-Северсон Э., Санче С., Хенгартнер Н. (май 2021 г.). «Оценка репродуктивного числа R 0 SARS-CoV-2 в США и восьми европейских странах и последствия для вакцинации» . Журнал теоретической биологии . 517 : 110621. Бибкод : 2021JThBi.51710621K . дои : 10.1016/j.jtbi.2021.110621 . ПМЦ 7880839 . ПМИД 33587929 .
- ^ Лю Ю, Гейл А.А., Уайлдер-Смит А, Роклов Дж (март 2020 г.). «Репродуктивная численность COVID-19 выше по сравнению с коронавирусом SARS» . Журнал туристической медицины . 27 (2): тааа021. дои : 10.1093/jtm/taaa021 . ПМК 7074654 . ПМИД 32052846 .
- ^ Дэвис Н.Г., Эбботт С., Барнард Р.К., Джарвис С.И., Кучарски А.Дж., Мандей Дж.Д., Пирсон К.А., Рассел Т.В., Талли Д.С., Уошберн А.Д., Венселирс Т., Гимма А., Уэйтс В., Вонг К.Л., ван Зандворт К., Сильверман Дж.Д., Диас -Ордаз К., Кио Р., Эгго Р.М., Фанк С., Джит М., Аткинс К.Е., Эдмундс У.Дж. (апрель 2021 г.). «Оценочная трансмиссивность и влияние SARS-CoV-2 линии B.1.1.7 в Англии» . Наука . 372 (6538): eabg3055. дои : 10.1126/science.abg3055 . ПМЦ 8128288 . ПМИД 33658326 .
- ^ Лю Ю, Роклов Ю (октябрь 2021 г.). «Репродуктивная численность дельта-варианта SARS-CoV-2 намного выше по сравнению с предковым вирусом SARS-CoV-2» . Журнал туристической медицины . 28 (7): тааб124. дои : 10.1093/jtm/taab124 . ПМЦ 8436367 . ПМИД 34369565 .
- ^ Перейти обратно: а б с д «Информационная панель COVID-19 Центра системных наук и инженерии (CSSE) Университета Джона Хопкинса (JHU)» . АркГИС . Университет Джонса Хопкинса . Проверено 10 марта 2023 г.
- ^ Брансвелл Х. (30 января 2020 г.). «Ограниченные данные о коронавирусе могут искажать предположения о его тяжести» . СТАТ . Архивировано из оригинала 1 февраля 2020 года . Проверено 13 марта 2020 г.
- ^ Ву Дж.Т., Люн К., Люн ГМ (февраль 2020 г.). «Прогнозирование текущей погоды и прогнозирование потенциального внутреннего и международного распространения вспышки 2019-nCoV, возникшей в Ухане, Китай: исследование моделирования» . Ланцет . 395 (10225): 689–697. дои : 10.1016/S0140-6736(20)30260-9 . ПМЦ 7159271 . ПМИД 32014114 .
- ^ Бозли С., МакКарри Дж. (30 января 2020 г.). «Смерть от коронавируса в Китае резко возрастает, поскольку страны изо всех сил пытаются эвакуировать граждан» . Хранитель . Архивировано из оригинала 6 февраля 2020 года . Проверено 10 марта 2020 г.
- ^ Паулин А (25 февраля 2020 г.). «Коронавирус: Китай отплатит Африке за защиту общественного здравоохранения» . Солнце . Архивировано из оригинала 9 марта 2020 года . Проверено 10 марта 2020 г.
Дальнейшее чтение
- Бар-Он Ю.М., Фламхольц А., Филлипс Р., Майло Р. (апрель 2020 г.). «SARS-CoV-2 (COVID-19) в цифрах» . электронная жизнь . 9 . arXiv : 2003.12886 . Бибкод : 2020arXiv200312886B . doi : 10.7554/eLife.57309 . ПМЦ 7224694 . ПМИД 32228860 .
- Брюссов Х. (май 2020 г.). «Новый коронавирус – краткий обзор современных знаний» . Микробная биотехнология . 13 (3): 607–612. дои : 10.1111/1751-7915.13557 . ПМК 7111068 . ПМИД 32144890 .
- Каселла М., Райник М., Алим А., Дулебон С., Ди Наполи Р. (январь 2020 г.). «Особенности, оценка и лечение коронавируса (COVID-19) » СтатПерлс . ПМИД 32150360 . Архивировано из оригинала 6 апреля. Получено 4 апреля.
- Лабораторное тестирование на коронавирусную болезнь 2019 (COVID-19) при подозрении на заболевание человека (Отчет). Всемирная организация здравоохранения . 2 марта 2020 г. HDL : 10665/331329 .
- Зумпурлис В., Гулиельмаки М., Керлс Е., Балиу С., Спандидос Д.А. (октябрь 2020 г.). «[Комментарий] Пандемия COVID‐19 как научная и социальная проблема XXI века » Отчеты о молекулярной медицине (обзор). 22 (4): 3035–3048. дои : 10.3892/ммр.2020.11393 . ПМЦ 7453598 . ПМИД 32945405 .
Внешние ссылки
- «Коронавирусная болезнь 2019 (COVID-19)» . Центры по контролю и профилактике заболеваний (CDC) . 11 февраля 2020 г.
- «Пандемия коронавирусной инфекции (COVID-19)» . Всемирная организация здравоохранения (ВОЗ) .
- «Последовательности SARS-CoV-2 (коронавирус 2 тяжелого острого респираторного синдрома)» . Национальный центр биотехнологической информации (NCBI) .
- «Ресурсный центр по COVID-19» . Ланцет .
- "Коронавирус (COVID-19)" . Медицинский журнал Новой Англии .
- «Covid-19: новая вспышка коронавируса» . Уайли . Архивировано из оригинала 24 сентября 2020 года . Проверено 13 февраля 2020 г. .
- "SARS-CoV-2" . База данных вирусных патогенов и ресурс для анализа .
- «Белковые структуры, связанные с SARS-CoV-2» . Банк данных по белкам .